本教學課程將介紹如何使用 Google Earth Engine 建立物種分布模型。我們會先簡要介紹物種分布模型,然後預測及分析瀕危鳥類仙八色鶇 (學名:Pitta nympha) 的棲息地。
請先執行我
執行下列儲存格,初始化 API。輸出內容會包含操作說明,教您如何使用帳戶授予這個筆記本 Earth Engine 的存取權。
import ee
# Trigger the authentication flow.
ee.Authenticate()
# Initialize the library.
ee.Initialize(project='my-project')
物種分布模型簡介
我們將探討物種分布模型、使用 Google Earth Engine 處理這類模型的優點、模型所需的資料,以及工作流程的結構。
什麼是物種分布模擬?
物種分布模型 (以下簡稱 SDM) 是最常用的方法,可估算物種的實際或潛在地理分布。這項技術會先找出適合特定物種的環境條件,然後找出這些條件在地理位置上的分布情形。
近年來,SDM 已成為保育規劃的重要環節,因此開發了各種模型技術。在 Google Earth Engine (以下簡稱 GEE) 中導入 SDM,可輕鬆存取大規模環境資料,並運用強大的運算功能和機器學習演算法支援,快速建立模型。
SDM 必須提供的資料
SDM 通常會運用已知物種出現記錄和環境變數之間的關係,找出族群可持續生存的條件。換句話說,您必須提供兩種模型輸入資料:
- 已知物種的出現記錄
- 各種環境變數
這些資料會輸入演算法,以找出與物種存在相關聯的環境條件。
使用 GEE 的 SDM 工作流程
使用 GEE 進行 SDM 的工作流程如下:
- 收集及預先處理物種出現資料
- 感興趣區域的定義
- 新增 GEE 環境變數
- 產生虛擬缺席資料
- 模型擬合和預測
- 變數重要性和準確度評估
使用 GEE 進行棲地預測和分析
我們將以八色鳥 (Pitta nympha) 為個案研究,說明如何運用以 GEE 為基礎的 SDM。雖然這個例子選用的是特定物種,但研究人員只要稍微修改提供的原始碼,就能將這項方法套用至任何感興趣的目標物種。
仙八色鳥是韓國罕見的夏季候鳥和過境候鳥,由於最近朝鮮半島氣候變暖,分布範圍正在擴大。此物種被歸類為稀有物種、II 級瀕危野生動物、第 204 號天然紀念物,在國家紅皮書中評估為區域性滅絕 (RE),根據 IUCN 類別則為易危 (VU)。
對八色鶇的保育規劃進行 SDM 似乎相當有價值。現在,我們將透過 GEE 進行棲地預測和分析。
首先,匯入 Python 程式庫。import 陳述式會匯入整個模組的內容,而 from import 陳述式則允許從模組匯入特定物件。
# Import libraries
import geemap
import geemap.colormaps as cm
import pandas as pd, geopandas as gpd
import numpy as np, matplotlib.pyplot as plt
import os, requests, math, random
from ipyleaflet import TileLayer
from statsmodels.stats.outliers_influence import variance_inflation_factor
收集和預先處理物種出現資料
現在,我們來收集仙八色鳥的出現資料。即使目前無法存取感興趣物種的出現資料,您也可以透過 GBIF API 取得特定物種的觀測資料。GBIF API 是一種介面,可存取 GBIF 提供的物種分布資料,讓使用者搜尋、篩選及下載資料,並取得各種物種相關資訊。
在下列程式碼中,species_name 變數會指派物種的學名 (例如 Pitta nympha 代表仙八色鳥),而 country_code 變數則會指派國家/地區代碼 (例如 KR 代表韓國)。base_url 變數會儲存 GBIF API 的位址。params 是包含要在 API 要求中使用的參數的字典:
scientificName:設定要搜尋的物種學名。country:將搜尋範圍限制在特定國家/地區內。hasCoordinate:確保只搜尋含有座標的資料 (true)。basisOfRecord:只選擇人類觀察記錄 (HUMAN_OBSERVATION)。limit:將傳回的結果數上限設為 10000。
def get_gbif_species_data(species_name, country_code):
"""
Retrieves observational data for a specific species using the GBIF API and returns it as a pandas DataFrame.
Parameters:
species_name (str): The scientific name of the species to query.
country_code (str): The country code of the where the observation data will be queried.
Returns:
pd.DataFrame: A pandas DataFrame containing the observational data.
"""
base_url = "https://api.gbif.org/v1/occurrence/search"
params = {
"scientificName": species_name,
"country": country_code,
"hasCoordinate": "true",
"basisOfRecord": "HUMAN_OBSERVATION",
"limit": 10000,
}
try:
response = requests.get(base_url, params=params)
response.raise_for_status() # Raises an exception for a response error.
data = response.json()
occurrences = data.get("results", [])
if occurrences: # If data is present
df = pd.json_normalize(occurrences)
return df
else:
print("No data found for the given species and country code.")
return pd.DataFrame() # Returns an empty DataFrame
except requests.RequestException as e:
print(f"Request failed: {e}")
return pd.DataFrame() # Returns an empty DataFrame in case of an exception
使用先前設定的參數,向 GBIF API 查詢仙八色鳥 (Pitta nympha) 的觀測記錄,並將結果載入 DataFrame,檢查第一列。DataFrame 是用於處理表格格式資料的資料結構,由資料列和資料欄組成。如有需要,DataFrame 可以儲存為 CSV 檔案,然後再讀取回來。
# Retrieve Fairy Pitta data
df = get_gbif_species_data("Pitta nympha", "KR")
"""
# Save DataFrame to CSV and read back in.
df.to_csv("pitta_nympha_data.csv", index=False)
df = pd.read_csv("pitta_nympha_data.csv")
"""
df.head(1) # Display the first row of the DataFrame
接著,我們將 DataFrame 轉換為 GeoDataFrame,其中包含地理資訊的資料欄 (geometry),並檢查第一列。GeoDataFrame 可以儲存為 GeoPackage 檔案 (*.gpkg),並讀取回。
# Convert DataFrame to GeoDataFrame
gdf = gpd.GeoDataFrame(
df,
geometry=gpd.points_from_xy(df.decimalLongitude,
df.decimalLatitude),
crs="EPSG:4326"
)[["species", "year", "month", "geometry"]]
"""
# Convert GeoDataFrame to GeoPackage (requires pycrs module)
%pip install -U -q pycrs
gdf.to_file("pitta_nympha_data.gpkg", driver="GPKG")
gdf = gpd.read_file("pitta_nympha_data.gpkg")
"""
gdf.head(1) # Display the first row of the GeoDataFrame
這次我們建立的函式可將 GeoDataFrame 中的資料依年份和月份分布情形視覺化,並以圖表形式顯示,然後儲存為圖片檔案。熱度圖可讓我們快速掌握各物種在不同年份和月份的出現頻率,直觀呈現資料中的時間變化和模式。這有助於識別物種出現資料中的時間模式和季節性變化,以及快速偵測資料中的離群值或品質問題。
# Yearly and monthly data distribution heatmap
def plot_heatmap(gdf, h_size=8):
statistics = gdf.groupby(["month", "year"]).size().unstack(fill_value=0)
# Heatmap
plt.figure(figsize=(h_size, h_size - 6))
heatmap = plt.imshow(
statistics.values, cmap="YlOrBr", origin="upper", aspect="auto"
)
# Display values above each pixel
for i in range(len(statistics.index)):
for j in range(len(statistics.columns)):
plt.text(
j, i, statistics.values[i, j], ha="center", va="center", color="black"
)
plt.colorbar(heatmap, label="Count")
plt.title("Monthly Species Count by Year")
plt.xlabel("Year")
plt.ylabel("Month")
plt.xticks(range(len(statistics.columns)), statistics.columns)
plt.yticks(range(len(statistics.index)), statistics.index)
plt.tight_layout()
plt.savefig("heatmap_plot.png")
plt.show()
plot_heatmap(gdf)
1995 年的資料非常稀疏,與其他年份相比有明顯落差,8 月和 9 月的樣本也有限,且與其他時期相比,呈現不同的季節性特徵。排除這類資料有助於提升模型的穩定性和預測能力。
不過請注意,排除資料可能會提升模型的泛化能力,但也可能導致與研究目標相關的重要資訊遺失。因此,請務必審慎考慮後再做出這類決定。
# Filtering data by year and month
filtered_gdf = gdf[
(~gdf['year'].eq(1995)) &
(~gdf['month'].between(8, 9))
]
現在,經過篩選的 GeoDataFrame 會轉換為 Google Earth Engine 物件。
# Convert GeoDataFrame to Earth Engine object
data_raw = geemap.geopandas_to_ee(filtered_gdf)
接著,我們將 SDM 結果的點陣像素大小定義為 1 公里解析度。
# Spatial resolution setting (meters)
grain_size = 1000
如果同一個 1 公里解析度的點陣像素內有多個發生點,這些點很可能位於同一地理位置,且環境條件相同。直接在分析中使用這類資料,可能會導致結果出現偏差。
換句話說,我們需要限制地理區域取樣偏差的潛在影響。為此,我們只會保留每個 1 公里像素內的一個位置,並移除所有其他位置,讓模型更客觀地反映環境狀況。
def remove_duplicates(data, grain_size):
# Select one occurrence record per pixel at the chosen spatial resolution
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
rand_point_vals = random_raster.sampleRegions(
collection=ee.FeatureCollection(data), geometries=True
)
return rand_point_vals.distinct("random")
data = remove_duplicates(data_raw, grain_size)
# Before selection and after selection
print("Original data size:", data_raw.size().getInfo())
print("Final data size:", data.size().getInfo())
下圖顯示預先處理前後的地理位置取樣偏差比較結果,藍色代表預先處理前,紅色代表預先處理後。為方便比較,地圖已將中心點設在漢拿山國家公園內,該區域的仙八色鶇出現座標密度較高。
# Visualization of geographic sampling bias before (blue) and after (red) preprocessing
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
# Add the random raster layer
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
Map.addLayer(
random_raster,
{"min": 0, "max": 1, "palette": ["black", "white"], "opacity": 0.5},
"Random Raster",
)
# Add the original data layer in blue
Map.addLayer(data_raw, {"color": "blue"}, "Original data")
# Add the final data layer in red
Map.addLayer(data, {"color": "red"}, "Final data")
# Set the center of the map to the coordinates
Map.setCenter(126.712, 33.516, 14)
Map
感興趣區域的定義
定義感興趣的區域 (以下簡稱 AOI) 是研究人員用來表示要分析的地理區域。與「研究領域」一詞的意義相近。
在這個情境中,我們取得發生點圖層幾何的邊界框,並在周圍建立 50 公里的緩衝區 (最大容許值為 1,000 公尺),以定義 AOI。
# Define the AOI
aoi = data.geometry().bounds().buffer(distance=50000, maxError=1000)
# Add the AOI to the map
outline = ee.Image().byte().paint(
featureCollection=aoi, color=1, width=3)
Map.remove_layer("Random Raster")
Map.addLayer(outline, {'palette': 'FF0000'}, "AOI")
Map.centerObject(aoi, 6)
Map
新增 GEE 環境變數
現在,我們要為分析新增環境變數。GEE 提供各種環境變數的資料集,例如溫度、降水量、海拔高度、土地覆蓋和地形。這些資料集有助於我們全面分析可能影響仙八色鳥棲地偏好的各種因素。
在 SDM 中選取 GEE 環境變數時,應反映物種的棲地偏好特徵。為此,應先對仙八色鳥的棲地偏好進行研究和文獻回顧。本教學課程主要著重於使用 GEE 的 SDM 工作流程,因此省略了部分深入細節。
WorldClim V1 Bioclim:這個資料集提供 19 個生物氣候變數,這些變數是根據每月溫度和降水量資料計算得出。涵蓋 1960 年至 1991 年的資料,解析度為 927.67 公尺。
# WorldClim V1 Bioclim
bio = ee.Image("WORLDCLIM/V1/BIO")
NASA SRTM 數位高程 30 公尺:這個資料集包含太空梭雷達地形測繪任務 (SRTM) 的數位高程資料。這些資料主要是在 2000 年左右收集,解析度約為 30 公尺 (1 弧秒)。下列程式碼會根據 SRTM 資料計算海拔高度、坡度、坡向和陰影層。
# NASA SRTM Digital Elevation 30m
terrain = ee.Algorithms.Terrain(ee.Image("USGS/SRTMGL1_003"))
全球森林覆蓋變化 (GFCC) 樹木覆蓋率多年全球 30 公尺:Landsat 的植被連續場 (VCF) 資料集會估算植被高度超過 5 公尺時,垂直投影植被覆蓋率的比例。這項資料集提供以 2000 年、2005 年、2010 年和 2015 年為中心的四個時間週期,解析度為 30 公尺。這裡會使用這四個時間範圍的中位數值。
# Global Forest Cover Change (GFCC) Tree Cover Multi-Year Global 30m
tcc = ee.ImageCollection("NASA/MEASURES/GFCC/TC/v3")
median_tcc = (
tcc.filterDate("2000-01-01", "2015-12-31")
.select(["tree_canopy_cover"], ["TCC"])
.median()
)
bio (生物氣候變數)、terrain (地形) 和 median_tcc (樹冠層覆蓋率) 會合併為單一多波段影像。系統會從 terrain 中選取 elevation 頻帶,並為 elevation 大於 0 的位置建立 watermask。這會遮蓋海平面以下的區域 (例如海洋),並協助研究人員全面分析 AOI 的各種環境因素。
# Combine bands into a multi-band image
predictors = bio.addBands(terrain).addBands(median_tcc)
# Create a water mask
watermask = terrain.select('elevation').gt(0)
# Mask out ocean pixels and clip to the area of interest
predictors = predictors.updateMask(watermask).clip(aoi)
如果模型中同時包含高度相關的預測變數,可能會發生多重共線性問題。當模型中的自變數之間存在強烈的線性關係時,就會發生多重共線性現象,導致模型係數 (權重) 的估計值不穩定。這種不穩定性可能會降低模型的可靠性,並導致難以預測或解讀新資料。因此,我們會考量多重共線性,並繼續選取預測變數。
首先,我們會產生 5,000 個隨機點,然後在這些點擷取單一多波段影像的預測變數值。
# Generate 5,000 random points
data_cor = predictors.sample(scale=grain_size, numPixels=5000, geometries=True)
# Extract predictor variable values
pvals = predictors.sampleRegions(collection=data_cor, scale=grain_size)
我們會將每個點的擷取預測值轉換為 DataFrame,然後檢查第一列。
# Converting predictor values from Earth Engine to a DataFrame
pvals_df = geemap.ee_to_df(pvals)
pvals_df.head(1)
# Displaying the columns
columns = pvals_df.columns
print(columns)
計算指定預測變數之間的 Spearman 相關係數,並以熱度圖呈現。
def plot_correlation_heatmap(dataframe, h_size=10, show_labels=False):
# Calculate Spearman correlation coefficients
correlation_matrix = dataframe.corr(method="spearman")
# Create a heatmap
plt.figure(figsize=(h_size, h_size-2))
plt.imshow(correlation_matrix, cmap='coolwarm', interpolation='nearest')
# Optionally display values on the heatmap
if show_labels:
for i in range(correlation_matrix.shape[0]):
for j in range(correlation_matrix.shape[1]):
plt.text(j, i, f"{correlation_matrix.iloc[i, j]:.2f}",
ha='center', va='center', color='white', fontsize=8)
columns = dataframe.columns.tolist()
plt.xticks(range(len(columns)), columns, rotation=90)
plt.yticks(range(len(columns)), columns)
plt.title("Variables Correlation Matrix")
plt.colorbar(label="Spearman Correlation")
plt.savefig('correlation_heatmap_plot.png')
plt.show()
# Plot the correlation heatmap of variables
plot_correlation_heatmap(pvals_df)
Spearman 相關係數有助於瞭解預測變數之間的一般關聯性,但無法直接評估多個變數的互動方式,特別是偵測多重共線性。
變異數膨脹因子 (以下簡稱 VIF) 是一種統計指標,用於評估多重共線性,並引導變數選取。這項指標代表每個自變數與其他自變數的線性關係程度,高 VIF 值可能代表多重共線性。
一般來說,當 VIF 值超過 5 或 10 時,表示變數與其他變數有很強的關聯性,可能會影響模型的穩定性和可解讀性。在本教學課程中,我們使用 VIF 值小於 10 的條件來選取變數。根據 VIF,我們選取了下列 6 個變數。
# Filter variables based on Variance Inflation Factor (VIF)
def filter_variables_by_vif(dataframe, threshold=10):
original_columns = dataframe.columns.tolist()
remaining_columns = original_columns[:]
while True:
vif_data = dataframe[remaining_columns]
vif_values = [
variance_inflation_factor(vif_data.values, i)
for i in range(vif_data.shape[1])
]
max_vif_index = vif_values.index(max(vif_values))
max_vif = max(vif_values)
if max_vif < threshold:
break
print(f"Removing '{remaining_columns[max_vif_index]}' with VIF {max_vif:.2f}")
del remaining_columns[max_vif_index]
filtered_data = dataframe[remaining_columns]
bands = filtered_data.columns.tolist()
print("Bands:", bands)
return filtered_data, bands
filtered_pvals_df, bands = filter_variables_by_vif(pvals_df)
# Variable Selection Based on VIF
predictors = predictors.select(bands)
# Plot the correlation heatmap of variables
plot_correlation_heatmap(filtered_pvals_df, h_size=6, show_labels=True)
接著,我們在地圖上以視覺化方式呈現 6 個所選預測變數。

您可以使用下列程式碼,探索地圖視覺化效果的可用調色盤。舉例來說,terrain 調色盤看起來會像這樣。
cm.plot_colormaps(width=8.0, height=0.2)
cm.plot_colormap('terrain', width=8.0, height=0.2, orientation='horizontal')
# Elevation layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['elevation'], 'min': 0, 'max': 1800, 'palette': cm.palettes.terrain}
Map.addLayer(predictors, vis_params, 'elevation')
Map.add_colorbar(vis_params, label="Elevation (m)", orientation="vertical", layer_name="elevation")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio09
min_max_val = (
predictors.select("bio09")
.multiply(0.1)
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio09 (Mean temperature of driest quarter) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"min": math.floor(min_max_val["bio09_min"]),
"max": math.ceil(min_max_val["bio09_max"]),
"palette": cm.palettes.hot,
}
Map.addLayer(predictors.select("bio09").multiply(0.1), vis_params, "bio09")
Map.add_colorbar(
vis_params,
label="Mean temperature of driest quarter (℃)",
orientation="vertical",
layer_name="bio09",
)
Map.centerObject(aoi, 6)
Map
# Slope layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['slope'], 'min': 0, 'max': 25, 'palette': cm.palettes.RdYlGn_r}
Map.addLayer(predictors, vis_params, 'slope')
Map.add_colorbar(vis_params, label="Slope", orientation="vertical", layer_name="slope")
Map.centerObject(aoi, 6)
Map
# Aspect layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['aspect'], 'min': 0, 'max': 360, 'palette': cm.palettes.rainbow}
Map.addLayer(predictors, vis_params, 'aspect')
Map.add_colorbar(vis_params, label="Aspect", orientation="vertical", layer_name="aspect")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio14
min_max_val = (
predictors.select("bio14")
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio14 (Precipitation of driest month) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["bio14"],
"min": math.floor(min_max_val["bio14_min"]),
"max": math.ceil(min_max_val["bio14_max"]),
"palette": cm.palettes.Blues,
}
Map.addLayer(predictors, vis_params, "bio14")
Map.add_colorbar(
vis_params,
label="Precipitation of driest month (mm)",
orientation="vertical",
layer_name="bio14",
)
Map.centerObject(aoi, 6)
Map
# TCC layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["TCC"],
"min": 0,
"max": 100,
"palette": ["ffffff", "afce56", "5f9c00", "0e6a00", "003800"],
}
Map.addLayer(predictors, vis_params, "TCC")
Map.add_colorbar(
vis_params, label="Tree Canopy Cover (%)", orientation="vertical", layer_name="TCC"
)
Map.centerObject(aoi, 6)
Map
產生虛擬缺席資料
在 SDM 過程中,物種的輸入資料選取作業主要有兩種方法:
出現位置與背景位置比較法:這個方法會比較特定物種的出現位置,以及物種未出現的其他位置。這裡的背景資料不一定是指物種不存在的區域,而是用來反映研究區域的整體環境狀況。這項指標可用於區分適合物種生存的環境,以及較不適合的環境。
出現/未出現法:這種方法會比較物種出現的地點 (出現) 與確定未出現的地點 (未出現)。這裡的缺席資料代表已知物種不存在的特定地點。這並非研究區域的整體環境狀況,而是指出估計沒有該物種的地點。
實務上,收集真實的缺席資料往往很困難,因此經常使用人工生成的虛擬缺席資料。不過,請務必瞭解這種方法的限制和潛在錯誤,因為人工生成的偽缺席點可能無法準確反映真實的缺席區域。
選擇這兩種方法時,請考量資料可用性、研究目標、模型準確度和可靠性,以及時間和資源。在本例中,我們會使用從 GBIF 收集的發生次數資料,以及人工產生的偽缺席資料,透過「Presence-Absence」方法建立模型。
系統會透過「環境剖析方法」產生虛擬缺席資料,具體步驟如下:
使用 k-means 分群法進行環境分類:系統會根據歐幾里得距離,使用 k-means 分群演算法將研究區域內的像素劃分為兩個叢集。其中一個叢集代表與隨機選取的 100 個存在位置具有相似環境特徵的區域,另一個叢集則代表特徵不同的區域。
在不同叢集中產生虛擬缺席資料:在第一個步驟中識別出的第二個叢集 (與出席資料的環境特徵不同) 內,系統會隨機產生虛擬缺席點。這些偽缺席點代表預期不會有該物種的位置。
# Randomly select 100 locations for occurrence
pvals = predictors.sampleRegions(
collection=data.randomColumn().sort('random').limit(100),
properties=[],
scale=grain_size
)
# Perform k-means clustering
clusterer = ee.Clusterer.wekaKMeans(
nClusters=2,
distanceFunction="Euclidean"
).train(pvals)
cl_result = predictors.cluster(clusterer)
# Get cluster ID for locations similar to occurrence
cl_id = cl_result.sampleRegions(
collection=data.randomColumn().sort('random').limit(200),
properties=[],
scale=grain_size
)
# Define non-occurrence areas in dissimilar clusters
cl_id = ee.FeatureCollection(cl_id).reduceColumns(ee.Reducer.mode(),['cluster'])
cl_id = ee.Number(cl_id.get('mode')).subtract(1).abs()
cl_mask = cl_result.select(['cluster']).eq(cl_id)
# Presence location mask
presence_mask = data.reduceToImage(properties=['random'],
reducer=ee.Reducer.first()
).reproject('EPSG:4326', None,
grain_size).mask().neq(1).selfMask()
# Masking presence locations in non-occurrence areas and clipping to AOI
area_for_pa = presence_mask.updateMask(cl_mask).clip(aoi)
# Area for Pseudo-absence
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(area_for_pa, {'palette': 'black'}, 'AreaForPA')
Map.centerObject(aoi, 6)
Map
模型擬合和預測
現在我們要將資料分成訓練資料和測試資料。訓練資料會用於訓練模型,找出最佳參數,而測試資料則會用於評估先前訓練的模型。在此情況下,請務必考量空間自相關的概念。
空間自相關是 SDM 的重要元素,與 Tobler 定律相關。這體現了「萬物皆有連帶關係,但相近的事物比相遠的事物更具關聯性」的概念。空間自相關代表物種位置與環境變數之間的顯著關係。不過,如果訓練和測試資料之間存在空間自相關,這兩個資料集之間的獨立性可能會受到影響。這會大幅影響模型一般化能力的評估結果。
解決這個問題的方法之一是空間區塊交叉驗證技術,也就是將資料分成訓練和測試資料集。這項技術會將資料分割為多個區塊,並將每個區塊分別做為訓練和測試資料集,以減少空間自相關的影響。這可提升資料集之間的獨立性,更準確地評估模型的泛化能力。
具體程序如下:
- 建立空間區塊:將整個資料集劃分為大小相等的空間區塊 (例如 50 x 50 公里)。
- 指派訓練和測試集:每個空間區塊都會隨機指派至訓練集 (70%) 或測試集 (30%)。這可防止模型過度配適特定區域的資料,並盡量取得更普遍適用的結果。
- 疊代交叉驗證:整個程序會重複 n 次 (例如 10 次)。在每次疊代中,系統會再次將區塊隨機劃分為訓練集和測試集,以提升模型的穩定性和可靠性。
- 產生虛擬缺席資料:在每次疊代中,系統會隨機產生虛擬缺席資料,以評估模型效能。
Scale = 50000
grid = watermask.reduceRegions(
collection=aoi.coveringGrid(scale=Scale, proj='EPSG:4326'),
reducer=ee.Reducer.mean()).filter(ee.Filter.neq('mean', None))
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(grid, {}, "Grid for spatial block cross validation")
Map.addLayer(outline, {'palette': 'FF0000'}, "Study Area")
Map.centerObject(aoi, 6)
Map
現在可以調整模型。模型調整作業包括瞭解資料中的模式,並據此調整模型的參數 (權重和偏差)。這個程序可讓模型在收到新資料時,做出更準確的預測。為此,我們定義了名為 SDM() 的函式,用來調整模型。
我們將使用「隨機森林」演算法。
def sdm(x):
seed = ee.Number(x)
# Random block division for training and validation
rand_blk = ee.FeatureCollection(grid).randomColumn(seed=seed).sort("random")
training_grid = rand_blk.filter(ee.Filter.lt("random", split)) # Grid for training
testing_grid = rand_blk.filter(ee.Filter.gte("random", split)) # Grid for testing
# Presence points
presence_points = ee.FeatureCollection(data)
presence_points = presence_points.map(lambda feature: feature.set("PresAbs", 1))
tr_presence_points = presence_points.filter(
ee.Filter.bounds(training_grid)
) # Presence points for training
te_presence_points = presence_points.filter(
ee.Filter.bounds(testing_grid)
) # Presence points for testing
# Pseudo-absence points for training
tr_pseudo_abs_points = area_for_pa.sample(
region=training_grid,
scale=grain_size,
numPixels=tr_presence_points.size().add(300),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for training
tr_pseudo_abs_points = (
tr_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(tr_presence_points.size()))
)
tr_pseudo_abs_points = tr_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
te_pseudo_abs_points = area_for_pa.sample(
region=testing_grid,
scale=grain_size,
numPixels=te_presence_points.size().add(100),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for testing
te_pseudo_abs_points = (
te_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(te_presence_points.size()))
)
te_pseudo_abs_points = te_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
# Merge training and pseudo-absence points
training_partition = tr_presence_points.merge(tr_pseudo_abs_points)
testing_partition = te_presence_points.merge(te_pseudo_abs_points)
# Extract predictor variable values at training points
train_pvals = predictors.sampleRegions(
collection=training_partition,
properties=["PresAbs"],
scale=grain_size,
geometries=True,
)
# Random Forest classifier
classifier = ee.Classifier.smileRandomForest(
numberOfTrees=500,
variablesPerSplit=None,
minLeafPopulation=10,
bagFraction=0.5,
maxNodes=None,
seed=seed,
)
# Presence probability: Habitat suitability map
classifier_pr = classifier.setOutputMode("PROBABILITY").train(
train_pvals, "PresAbs", bands
)
classified_img_pr = predictors.select(bands).classify(classifier_pr)
# Binary presence/absence map: Potential distribution map
classifier_bin = classifier.setOutputMode("CLASSIFICATION").train(
train_pvals, "PresAbs", bands
)
classified_img_bin = predictors.select(bands).classify(classifier_bin)
return [
classified_img_pr,
classified_img_bin,
training_partition,
testing_partition,
], classifier_pr
空間區塊會分別以 70% 和 30% 的比例,用於模型訓練和模型測試。在每次疊代中,系統會在每個訓練和測試集中隨機產生偽缺席資料。因此,每次執行都會產生不同的存在和偽缺席資料集,用於模型訓練和測試。
split = 0.7
numiter = 10
# Random Seed
runif = lambda length: [random.randint(1, 1000) for _ in range(length)]
items = runif(numiter)
# Fixed seed
# items = [287, 288, 553, 226, 151, 255, 902, 267, 419, 538]
results_list = [] # Initialize SDM results list
importances_list = [] # Initialize variable importance list
for item in items:
result, trained = sdm(item)
# Accumulate SDM results into the list
results_list.extend(result)
# Accumulate variable importance into the list
importance = ee.Dictionary(trained.explain()).get('importance')
importances_list.extend(importance.getInfo().items())
# Flatten the SDM results list
results = ee.List(results_list).flatten()
現在,我們可以查看八色鳥的棲地適宜性地圖和潛在分布地圖。在本例中,棲地適宜性地圖是透過 mean() 函式計算所有圖片中每個像素位置的平均值而建立,而潛在分布地圖則是透過 mode() 函式,判斷所有圖片中每個像素位置最常出現的值而產生。
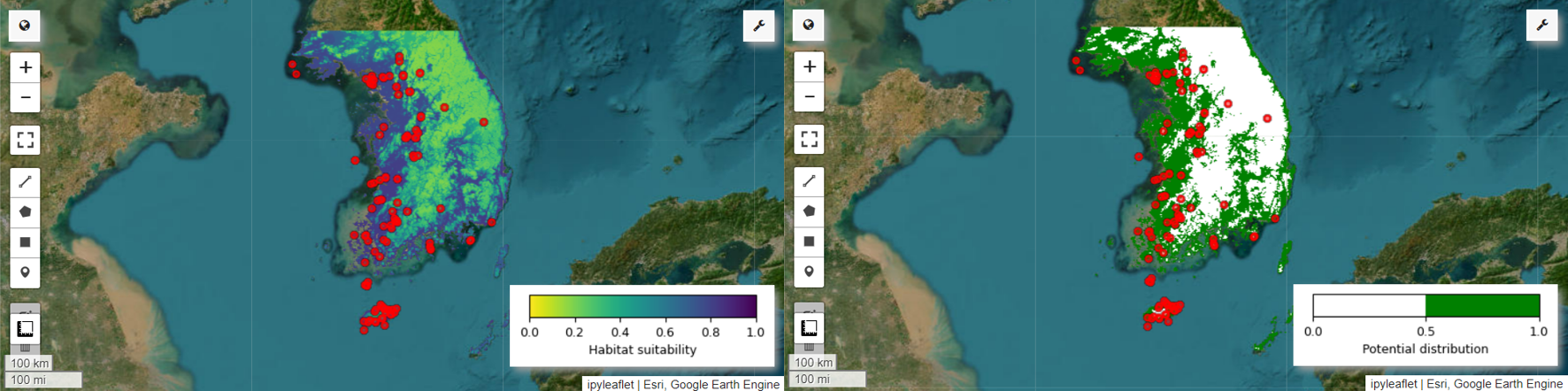
# Habitat suitability map
images = ee.List.sequence(
0, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x))
model_average = ee.ImageCollection.fromImages(images).mean()
Map = geemap.Map(layout={'height':'400px', 'width':'800px'}, basemap='Esri.WorldImagery')
vis_params = {
'min': 0,
'max': 1,
'palette': cm.palettes.viridis_r}
Map.addLayer(model_average, vis_params, 'Habitat suitability')
Map.add_colorbar(vis_params, label="Habitat suitability",
orientation="horizontal",
layer_name="Habitat suitability")
Map.addLayer(data, {'color':'red'}, 'Presence')
Map.centerObject(aoi, 6)
Map
# Potential distribution map
images2 = ee.List.sequence(1, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x)
)
distribution_map = ee.ImageCollection.fromImages(images2).mode()
Map = geemap.Map(
layout={"height": "400px", "width": "800px"}, basemap="Esri.WorldImagery"
)
vis_params = {"min": 0, "max": 1, "palette": ["white", "green"]}
Map.addLayer(distribution_map, vis_params, "Potential distribution")
Map.addLayer(data, {"color": "red"}, "Presence")
Map.add_colorbar(
vis_params,
label="Potential distribution",
discrete=True,
orientation="horizontal",
layer_name="Potential distribution",
)
Map.centerObject(data.geometry(), 6)
Map
變數重要性和準確度評估
隨機森林 (ee.Classifier.smileRandomForest) 是集成學習方法之一,會建構多個決策樹來進行預測。每棵決策樹都會從不同的資料子集獨立學習,並彙整結果,以做出更準確且穩定的預測。
變數重要性是一種評估指標,可評估隨機森林模型中每個變數對預測結果的影響。我們會使用先前定義的 importances_list 來計算及列印平均變數重要性。
def plot_variable_importance(importances_list):
# Extract each variable importance value into a list
variables = [item[0] for item in importances_list]
importances = [item[1] for item in importances_list]
# Calculate the average importance for each variable
average_importances = {}
for variable in set(variables):
indices = [i for i, var in enumerate(variables) if var == variable]
average_importance = np.mean([importances[i] for i in indices])
average_importances[variable] = average_importance
# Sort the importances in descending order of importance
sorted_importances = sorted(average_importances.items(),
key=lambda x: x[1], reverse=False)
variables = [item[0] for item in sorted_importances]
avg_importances = [item[1] for item in sorted_importances]
# Adjust the graph size
plt.figure(figsize=(8, 4))
# Plot the average importance as a horizontal bar chart
plt.barh(variables, avg_importances)
plt.xlabel('Importance')
plt.ylabel('Variables')
plt.title('Average Variable Importance')
# Display values above the bars
for i, v in enumerate(avg_importances):
plt.text(v + 0.02, i, f"{v:.2f}", va='center')
# Adjust the x-axis range
plt.xlim(0, max(avg_importances) + 5) # Adjust to the desired range
plt.tight_layout()
plt.savefig('variable_importance.png')
plt.show()
plot_variable_importance(importances_list)
我們會使用測試資料集,計算每次執行的 AUC-ROC 和 AUC-PR。接著,我們會在 n 次疊代中計算平均 AUC-ROC 和 AUC-PR。
AUC-ROC 代表「敏感度 (召回率) 與 1 - 特異度」圖表曲線下的面積,說明門檻變更時敏感度和特異度之間的關係。特異性是根據所有觀察到的非發生事件計算而得。因此,AUC-ROC 涵蓋混淆矩陣的所有象限。
AUC-PR 代表「精確度與喚回度 (敏感度)」圖表曲線下的面積,顯示門檻變化時精確度和喚回度之間的關係。精確度是根據所有預測的發生次數計算。因此,AUC-PR 不會納入真正負值 (TN)。
def print_pres_abs_sizes(TestingDatasets, numiter):
# Check and print the sizes of presence and pseudo-absence coordinates
def get_pres_abs_size(x):
fc = ee.FeatureCollection(TestingDatasets.get(x))
presence_size = fc.filter(ee.Filter.eq("PresAbs", 1)).size()
pseudo_absence_size = fc.filter(ee.Filter.eq("PresAbs", 0)).size()
return ee.List([presence_size, pseudo_absence_size])
sizes_info = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(get_pres_abs_size)
.getInfo()
)
for i, sizes in enumerate(sizes_info):
presence_size = sizes[0]
pseudo_absence_size = sizes[1]
print(
f"Iteration {i + 1}: Presence Size = {presence_size}, Pseudo-absence Size = {pseudo_absence_size}"
)
# Extracting the Testing Datasets
testing_datasets = ee.List.sequence(
3, ee.Number(numiter).multiply(4).subtract(1), 4
).map(lambda x: results.get(x))
print_pres_abs_sizes(testing_datasets, numiter)
def get_acc(hsm, t_data, grain_size):
pr_prob_vals = hsm.sampleRegions(
collection=t_data, properties=["PresAbs"], scale=grain_size
)
seq = ee.List.sequence(start=0, end=1, count=25) # Divide 0 to 1 into 25 intervals
def calculate_metrics(cutoff):
# Each element of the seq list is passed as cutoff(threshold value)
# Observed present = TP + FN
pres = pr_prob_vals.filterMetadata("PresAbs", "equals", 1)
# TP (True Positive)
tp = ee.Number(
pres.filterMetadata("classification", "greater_than", cutoff).size()
)
# TPR (True Positive Rate) = Recall = Sensitivity = TP / (TP + FN) = TP / Observed present
tpr = tp.divide(pres.size())
# Observed absent = FP + TN
abs = pr_prob_vals.filterMetadata("PresAbs", "equals", 0)
# FN (False Negative)
fn = ee.Number(
pres.filterMetadata("classification", "less_than", cutoff).size()
)
# TNR (True Negative Rate) = Specificity = TN / (FP + TN) = TN / Observed absent
tn = ee.Number(abs.filterMetadata("classification", "less_than", cutoff).size())
tnr = tn.divide(abs.size())
# FP (False Positive)
fp = ee.Number(
abs.filterMetadata("classification", "greater_than", cutoff).size()
)
# FPR (False Positive Rate) = FP / (FP + TN) = FP / Observed absent
fpr = fp.divide(abs.size())
# Precision = TP / (TP + FP) = TP / Predicted present
precision = tp.divide(tp.add(fp))
# SUMSS = SUM of Sensitivity and Specificity
sumss = tpr.add(tnr)
return ee.Feature(
None,
{
"cutoff": cutoff,
"TP": tp,
"TN": tn,
"FP": fp,
"FN": fn,
"TPR": tpr,
"TNR": tnr,
"FPR": fpr,
"Precision": precision,
"SUMSS": sumss,
},
)
return ee.FeatureCollection(seq.map(calculate_metrics))
def calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter):
# Calculate AUC-ROC and AUC-PR
def calculate_auc_metrics(x):
hsm = ee.Image(images.get(x))
t_data = ee.FeatureCollection(testing_datasets.get(x))
acc = get_acc(hsm, t_data, grain_size)
# Calculate AUC-ROC
x = ee.Array(acc.aggregate_array("FPR"))
y = ee.Array(acc.aggregate_array("TPR"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_roc = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
# Calculate AUC-PR
x = ee.Array(acc.aggregate_array("TPR"))
y = ee.Array(acc.aggregate_array("Precision"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_pr = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
return (auc_roc, auc_pr)
auc_metrics = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(calculate_auc_metrics)
.getInfo()
)
# Print AUC-ROC and AUC-PR for each iteration
df = pd.DataFrame(auc_metrics, columns=["AUC-ROC", "AUC-PR"])
df.index = [f"Iteration {i + 1}" for i in range(len(df))]
df.to_csv("auc_metrics.csv", index_label="Iteration")
print(df)
# Calculate mean and standard deviation of AUC-ROC and AUC-PR
mean_auc_roc, std_auc_roc = df["AUC-ROC"].mean(), df["AUC-ROC"].std()
mean_auc_pr, std_auc_pr = df["AUC-PR"].mean(), df["AUC-PR"].std()
print(f"Mean AUC-ROC = {mean_auc_roc:.4f} ± {std_auc_roc:.4f}")
print(f"Mean AUC-PR = {mean_auc_pr:.4f} ± {std_auc_pr:.4f}")
%%time
# Calculate AUC-ROC and AUC-PR
calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter)
本教學課程提供實用範例,說明如何使用 Google Earth Engine (GEE) 進行物種分布模型 (SDM) 建立作業。重要結論是,GEE 在 SDM 領域中具有多功能性和彈性。Earth Engine 具備強大的地理空間資料處理功能,可為研究人員和保育人士提供無限可能,協助他們瞭解及保護地球上的生物多樣性。只要運用本教學課程學到的知識和技能,就能探索並貢獻生態研究這個迷人的領域。

 在 Google Colab 中執行
在 Google Colab 中執行
 在 GitHub 上查看來源
在 GitHub 上查看來源