يمكن استخدام حقول التضمين كمدخلات/متنبئات للميزات في الانحدار بالطريقة نفسها التي تُستخدم بها للتصنيف.
في هذا البرنامج التعليمي، سنتعلّم كيفية استخدام طبقات حقول التضمين 64D كمدخلات لتحليل الانحدار المتعدد الذي يتوقّع الكتلة الحيوية فوق سطح الأرض (AGB).
تجمع مهمة التحقيق في ديناميكيات النظام البيئي العالمي (GEDI) التابعة لوكالة ناسا قياسات LIDAR على طول مقاطع عرضية أرضية بدقة مكانية تبلغ 30 مترًا على فواصل زمنية تبلغ 60 مترًا. سنستخدم مجموعة بيانات GEDI L4A Raster Aboveground Biomass Density التي تحتوي على تقديرات نقطية لكثافة الكتلة الحيوية فوق سطح الأرض (AGBD) والتي سيتم استخدامها كمتغيّر متوقّع في نموذج الانحدار.
اختيار منطقة
لنبدأ بتحديد منطقة اهتمام. في هذا البرنامج التعليمي، سنختار منطقة في سلسلة جبال غاتس الغربية في الهند ونحدّد مضلّعًا كمتغيّر هندسي. بدلاً من ذلك، يمكنك استخدام "أدوات الرسم" في "محرّر الرموز" لرسم مضلّع حول المنطقة التي تهمّك وسيتم حفظها كمتغيّر هندسي في عمليات الاستيراد. نستخدم أيضًا الخريطة الأساسية "صور الأقمار الصناعية" التي تسهّل تحديد المناطق التي تكثر فيها النباتات.
var geometry = ee.Geometry.Polygon([[
[74.322, 14.981],
[74.322, 14.765],
[74.648, 14.765],
[74.648, 14.980]
]]);
// Use the satellite basemap
Map.setOptions('SATELLITE');

الشكل: اختيار المنطقة محط الاهتمام لتوقّع الكتلة الحيوية فوق سطح الأرض
اختيار فترة زمنية
اختَر سنة نريد إجراء الانحدار لها. يُرجى العِلم أنّه يتم تجميع بيانات Satellite Embeddings على فترات سنوية، لذا نحدّد الفترة للعام بأكمله.
var startDate = ee.Date.fromYMD(2022, 1, 1);
var endDate = startDate.advance(1, 'year');
إعداد مجموعة بيانات Satellite Embedding
سيتم استخدام "صور القمر الصناعي المضمّنة" ذات الـ 64 نطاقًا كمتنبئ للانحدار. نحمّل مجموعة بيانات "تضمين صور الأقمار الصناعية"، ثم نطبّق فلترًا على الصور حسب السنة والمنطقة المحدّدتَين.
var embeddings = ee.ImageCollection('GOOGLE/SATELLITE_EMBEDDING/V1/ANNUAL');
var embeddingsFiltered = embeddings
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
يتم تقسيم صور "التضمين عبر الأقمار الصناعية" إلى مربّعات ويتم عرضها في العرض وفقًا لمناطق نظام الإحداثيات العالمي (UTM) الخاصة بالمربّع. ونتيجةً لذلك، نحصل على مربّعات متعدّدة لتضمين صور القمر الصناعي تغطّي المنطقة التي نريدها. للحصول على صورة واحدة، علينا دمجها. في Earth Engine، يتم تعيين الإسقاط التلقائي لمجموعة من الصور المدخلة، وهو نظام WGS84 بمقياس درجة واحدة. بما أنّنا سنجمع هذه الصورة المجمّعة ونعيد عرضها لاحقًا في البرنامج التعليمي، من المفيد الاحتفاظ بالعرض الأصلي. يمكننا استخراج معلومات الإسقاط من إحدى المربّعات وتعيينها على الصورة المجمّعة باستخدام الدالة setDefaultProjection().
// Extract the projection of the first band of the first image
var embeddingsProjection = ee.Image(embeddingsFiltered.first()).select(0).projection();
// Set the projection of the mosaic to the extracted projection
var embeddingsImage = embeddingsFiltered.mosaic()
.setDefaultProjection(embeddingsProjection);
إعداد فسيفساء GEDI L4A
بما أنّ تقديرات الكتلة الحيوية التي توفّرها أداة GEDI ستُستخدم لتدريب نموذج الانحدار، من الضروري فلترة بيانات GEDI غير الصالحة أو غير الموثوق بها قبل استخدامها. نطبّق عدة أقنعة لإزالة القياسات التي يُحتمل أن تكون خاطئة.
- إزالة جميع القياسات التي لا تستوفي متطلبات الجودة (l4_quality_flag = 0 وdegrade_flag > 0)
- إزالة جميع القياسات التي تتضمّن خطأ نسبيًا كبيرًا ("agbd_se" / "agbd" > %50)
- إزالة جميع القياسات على المنحدرات التي تزيد عن% 30 استنادًا إلى نموذج الارتفاع الرقمي (DEM) GLO-30 من Copernicus
أخيرًا، نختار جميع القياسات المتبقية للفترة الزمنية والمنطقة التي تهمّنا وننشئ صورة مجمّعة.
var gedi = ee.ImageCollection('LARSE/GEDI/GEDI04_A_002_MONTHLY');
// Function to select the highest quality GEDI data
var qualityMask = function(image) {
return image.updateMask(image.select('l4_quality_flag').eq(1))
.updateMask(image.select('degrade_flag').eq(0));
};
// Function to mask unreliable GEDI measurements
// with a relative standard error > 50%
// agbd_se / agbd > 0.5
var errorMask = function(image) {
var relative_se = image.select('agbd_se')
.divide(image.select('agbd'));
return image.updateMask(relative_se.lte(0.5));
};
// Function to mask GEDI measurements on slopes > 30%
var slopeMask = function(image) {
// Use Copernicus GLO-30 DEM for calculating slope
var glo30 = ee.ImageCollection('COPERNICUS/DEM/GLO30');
var glo30Filtered = glo30
.filter(ee.Filter.bounds(geometry))
.select('DEM');
// Extract the projection
var demProj = glo30Filtered.first().select(0).projection();
// The dataset consists of individual images
// Create a mosaic and set the projection
var elevation = glo30Filtered.mosaic().rename('dem')
.setDefaultProjection(demProj);
// Compute the slope
var slope = ee.Terrain.slope(elevation);
return image.updateMask(slope.lt(30));
};
var gediFiltered = gedi
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
var gediProjection = ee.Image(gediFiltered.first())
.select('agbd').projection();
var gediProcessed = gediFiltered
.map(qualityMask)
.map(errorMask)
.map(slopeMask);
var gediMosaic = gediProcessed.mosaic()
.select('agbd').setDefaultProjection(gediProjection);
// Visualize the GEDI Mosaic
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(gediMosaic, gediVis, 'GEDI L4A (Filtered)', false);

الشكل: ملاحظات جاهزة حول الكتلة الحيوية من GEDI
إعادة أخذ العيّنات وتجميع البيانات
قبل أخذ عيّنات من وحدات البكسل لتدريب نموذج انحدار، نعيد أخذ العيّنات وإعادة إسقاط المدخلات على شبكة وحدات البكسل نفسها. تبلغ دقة قياسات GEDI الأفقية +/- 9 أمتار. تحدث مشكلة عند مطابقة قيم AGB في GEDI مع وحدات البكسل المضمّنة في Satellite. للتغلّب على هذه المشكلة، نعيد أخذ عيّنات من جميع الصور المدخلة ونجمعها في شبكة بكسل أكبر مع قيم متوسطة من وحدات البكسل الأصلية. يساعد ذلك أيضًا في إزالة التشويش من البيانات والمساعدة في إنشاء نموذج أفضل لتعلُّم الآلة.
// Choose the grid size and projection
var gridScale = 100;
var gridProjection = ee.Projection('EPSG:3857')
.atScale(gridScale);
// Create a stacked image with predictor and predicted variables
var stacked = embeddingsImage.addBands(gediMosaic);
// Set the resampling mode
var stacked = stacked.resample('bilinear');
// Aggregate pixels with 'mean' statistics
var stackedResampled = stacked
.reduceResolution({
reducer: ee.Reducer.mean(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// As larger GEDI pixels contain masked original
// pixels, it has a transparency mask.
// We update the mask to remove the transparency
var stackedResampled = stackedResampled
.updateMask(stackedResampled.mask().gt(0));
إنّ إعادة عرض وحدات البكسل وتجميعها عملية مكلفة، ومن الممارسات الجيدة تصدير الصورة المجمّعة الناتجة كأصل واستخدام الصورة المحسوبة مسبقًا في الخطوات اللاحقة. سيساعد ذلك في التغلّب على أخطاء انتهت مهلة الحساب أو تجاوزت ذاكرة المستخدم عند العمل مع مناطق كبيرة.
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var mosaicExportImage = 'gedi_mosaic';
var mosaicExportImagePath = exportFolder + mosaicExportImage;
Export.image.toAsset({
image: stackedResampled.clip(geometry),
description: 'GEDI_Mosaic_Export',
assetId: mosaicExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
ابدأ مهمة التصدير وانتظِر حتى تنتهي. بعد الانتهاء من ذلك، نستورد مادة العرض ونواصل إنشاء النموذج.
// Use the exported asset
var stackedResampled = ee.Image(mosaicExportImagePath);
استخراج ميزات التدريب
لدينا بيانات الإدخال جاهزة لاستخراج ميزات التدريب. نستخدم نطاقات Satellite Embedding كمتغيرات تابعة (متوقّعة) وقيم GEDI AGBD كمتغير مستقل (متوقّع) في نموذج الانحدار. يمكننا استخراج القيم المتطابقة في كل بكسل وإعداد مجموعة بيانات التدريب. يتم إخفاء معظم صورة GEDI، ولا تحتوي إلا على قيم في مجموعة فرعية صغيرة من وحدات البكسل. إذا استخدمنا sample()، سيتم عرض قيم فارغة في الغالب. للتغلّب على هذه المشكلة، ننشئ نطاقًا من الفئات من قناع GEDI ونستخدم stratifiedSample() لضمان أخذ عينات من وحدات البكسل غير المقنّعة.
var predictors = embeddingsImage.bandNames();
var predicted = gediMosaic.bandNames().get(0);
print('predictors', predictors);
print('predicted', predicted);
var predictorImage = stackedResampled.select(predictors);
var predictedImage = stackedResampled.select([predicted]);
var classMask = predictedImage.mask().toInt().rename('class');
var numSamples = 1000;
// We set classPoints to [0, numSamples]
// This will give us 0 points for class 0 (masked areas)
// and numSample points for class 1 (non-masked areas)
var training = stackedResampled.addBands(classMask)
.stratifiedSample({
numPoints: numSamples,
classBand: 'class',
region: geometry,
scale: gridScale,
classValues: [0, 1],
classPoints: [0, numSamples],
dropNulls: true,
tileScale: 16,
});
print('Number of Features Extracted', training.size());
print('Sample Training Feature', training.first());
تدريب نموذج انحدار
نحن الآن جاهزون لتدريب النموذج. يمكن استخدام العديد من المصنِّفات في Earth Engine لتنفيذ مهام التصنيف والانحدار. بما أنّنا نريد توقّع قيمة رقمية (بدلاً من فئة)، يمكننا ضبط المصنّف على التشغيل في وضع REGRESSION والتدريب باستخدام بيانات التدريب. بعد تدريب النموذج، يمكننا مقارنة القيمة المتوقّعة للنموذج بالقيم المُدخلة وحساب متوسط الخطأ التربيعي (rmse) ومعامل الارتباط r^2 للتحقّق من أداء النموذج.
// Use the RandomForest classifier and set the
// output mode to REGRESSION
var model = ee.Classifier.smileRandomForest(50)
.setOutputMode('REGRESSION')
.train({
features: training,
classProperty: predicted,
inputProperties: predictors
});
// Get model's predictions for training samples
var predicted = training.classify({
classifier: model,
outputName: 'agbd_predicted'
});
// Calculate RMSE
var calculateRmse = function(input) {
var observed = ee.Array(
input.aggregate_array('agbd'));
var predicted = ee.Array(
input.aggregate_array('agbd_predicted'));
var rmse = observed.subtract(predicted).pow(2)
.reduce('mean', [0]).sqrt().get([0]);
return rmse;
};
var rmse = calculateRmse(predicted);
print('RMSE', rmse);
// Create a plot of observed vs. predicted values
var chart = ui.Chart.feature.byFeature({
features: predicted.select(['agbd', 'agbd_predicted']),
xProperty: 'agbd',
yProperties: ['agbd_predicted'],
}).setChartType('ScatterChart')
.setOptions({
title: 'Aboveground Biomass Density (Mg/Ha)',
dataOpacity: 0.8,
hAxis: {'title': 'Observed'},
vAxis: {'title': 'Predicted'},
legend: {position: 'right'},
series: {
0: {
visibleInLegend: false,
color: '#525252',
pointSize: 3,
pointShape: 'triangle',
},
},
trendlines: {
0: {
type: 'linear',
color: 'black',
lineWidth: 1,
pointSize: 0,
labelInLegend: 'Linear Fit',
visibleInLegend: true,
showR2: true
}
},
chartArea: {left: 100, bottom: 100, width: '50%'},
});
print(chart);
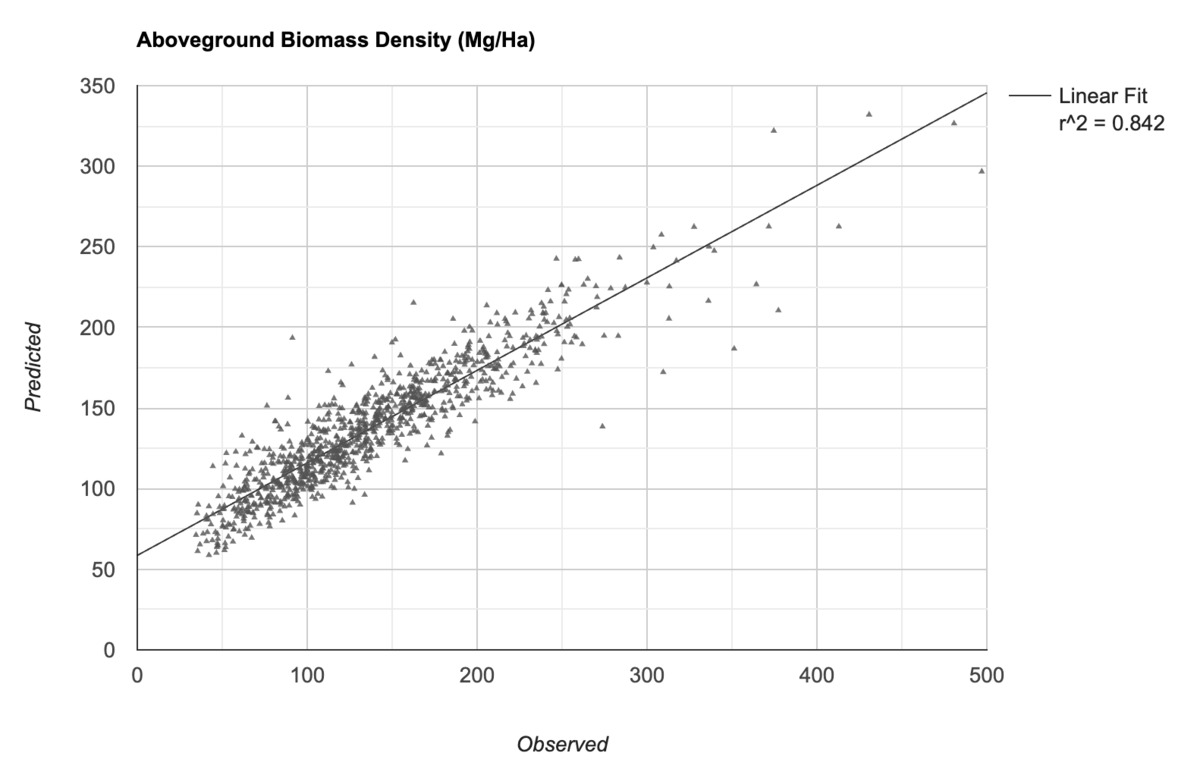
الشكل: القيم المرصودة مقارنةً بقيم AGBD المتوقّعة في النموذج
إنشاء توقّعات للقيم غير المعروفة
بعد أن نصبح راضين عن النموذج، يمكننا استخدامه لإنشاء توقّعات في مواقع جغرافية غير معروفة من الصورة التي تحتوي على نطاقات المتغيّر التابع.
// We set the band name of the output image as 'agbd'
var predictedImage = stackedResampled.classify({
classifier: model,
outputName: 'agbd'
});
أصبحت الصورة التي تحتوي على قيم AGBD المتوقّعة في كل بكسل جاهزة للتصدير. سنستخدم هذا في الجزء التالي لعرض النتائج بشكل مرئي.
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var predictedExportImage = 'predicted_agbd';
var predictedExportImagePath = exportFolder + predictedExportImage;
Export.image.toAsset({
image: predictedImage.clip(geometry),
description: 'Predicted_Image_Export',
assetId: predictedExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
ابدأ مهمة التصدير وانتظِر حتى تنتهي. بعد الانتهاء، نستورد "مادة العرض" ونعرض النتائج.
var predictedImage = ee.Image(predictedExportImagePath);
// Visualize the image
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(predictedImage, gediVis, 'Predicted AGBD');

الشكل: متوسط الكتلة الحيوية فوق سطح الأرض المتوقّع. تشير درجات اللون الأخضر الداكنة إلى كثافة الكتلة الحيوية المتوقّعة الأكبر
تقدير إجمالي الكتلة الحيوية
لدينا الآن قيم متوقّعة لكتلة المادة الحيوية فوق سطح الأرض لكل وحدة بكسل في الصورة، ويمكن استخدامها لتقدير إجمالي مخزون كتلة المادة الحيوية فوق سطح الأرض في المنطقة. ولكن يجب أولاً إزالة جميع وحدات البكسل التي تنتمي إلى مناطق غير مغطاة بالنباتات. يمكننا استخدام مجموعة بيانات ESA WorldCover الخاصة بغطاء الأرض واختيار وحدات البكسل التي تحتوي على نباتات.
// GEDI data is processed only for certain landcovers
// from Plant Functional Types (PFT) classification
// https://doi.org/10.1029/2022EA002516
// Here we use ESA WorldCover v200 product to
// select landcovers representing vegetated areas
var worldcover = ee.ImageCollection('ESA/WorldCover/v200').first();
// Aggregate pixels to the same grid as other dataset
// with 'mode' value.
// i.e. The landcover with highest occurrence within the grid
var worldcoverResampled = worldcover
.reduceResolution({
reducer: ee.Reducer.mode(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// Select grids for the following classes
// | Class Name | Value |
// | Forests | 10 |
// | Shrubland | 20 |
// | Grassland | 30 |
// | Cropland | 40 |
// | Mangroves | 95 |
var landCoverMask = worldcoverResampled.eq(10)
.or(worldcoverResampled.eq(20))
.or(worldcoverResampled.eq(30))
.or(worldcoverResampled.eq(40))
.or(worldcoverResampled.eq(95));
var predictedImageMasked = predictedImage
.updateMask(landCoverMask);
Map.addLayer(predictedImageMasked, gediVis, 'Predicted AGBD (Masked)');

الشكل: تقدير الكتلة الحيوية فوق سطح الأرض (AGBD) مع إخفاء المناطق غير النباتية
وحدات قيم AGBD في GEDI هي ميغاغرام لكل هكتار (Mg/ha). للحصول على إجمالي الكتلة الحيوية فوق سطح الأرض، نضرب كل بكسل في مساحته بالهكتارات ونجمع قيمها.
var pixelAreaHa = ee.Image.pixelArea().divide(10000);
var predictedAgb = predictedImageMasked.multiply(pixelAreaHa);
var stats = predictedAgb.reduceRegion({
reducer: ee.Reducer.sum(),
geometry: geometry,
scale: gridScale,
maxPixels: 1e10,
tileScale: 16
});
// Result is a dictionary with key for each band
var totalAgb = stats.getNumber('agbd');
print('Total AGB (Mg)', totalAgb);
جرِّب النص البرمجي الكامل لهذا الدليل التعليمي في "أداة تعديل الرموز البرمجية" في Earth Engine.