Les champs d'embedding peuvent être utilisés comme entrées/variables de prédiction de caractéristiques pour la régression de la même manière qu'ils sont utilisés pour la classification.
Dans ce tutoriel, nous allons apprendre à utiliser les couches de champs d'intégration 64D comme entrées pour une analyse de régression multiple permettant de prédire la biomasse aérienne (AGB).
La mission Global Ecosystem Dynamics Investigation (GEDI) de la NASA collecte des mesures LIDAR le long de transects au sol avec une résolution spatiale de 30 mètres à des intervalles de 60 mètres. Nous allons utiliser l'ensemble de données GEDI L4A Raster Aboveground Biomass Density, qui contient des estimations ponctuelles de la densité de biomasse aérienne (AGBD, Aboveground Biomass Density). Ces estimations serviront de variable prédite dans le modèle de régression.
Sélectionnez une région
Commençons par définir une région d'intérêt. Pour ce tutoriel, nous allons choisir une région des Ghâts occidentaux en Inde et définir un polygone comme variable de géométrie. Vous pouvez également utiliser les outils de dessin de l'éditeur de code pour dessiner un polygone autour de la région d'intérêt, qui sera enregistré en tant que variable de géométrie dans les importations. Nous utilisons également la carte de base satellite, qui permet de localiser facilement les zones végétalisées.
var geometry = ee.Geometry.Polygon([[
[74.322, 14.981],
[74.322, 14.765],
[74.648, 14.765],
[74.648, 14.980]
]]);
// Use the satellite basemap
Map.setOptions('SATELLITE');

Figure : Sélection de la zone d'intérêt pour la prédiction de la biomasse aérienne
Sélectionner une période
Choisissez une année pour laquelle nous souhaitons exécuter la régression. N'oubliez pas que les embeddings satellite sont agrégés à intervalles annuels. Nous définissons donc la période pour l'année entière.
var startDate = ee.Date.fromYMD(2022, 1, 1);
var endDate = startDate.advance(1, 'year');
Préparer l'ensemble de données d'embeddings satellite
Les images d'intégration satellite à 64 bandes seront utilisées comme prédicteur pour la régression. Nous chargeons l'ensemble de données sur l'intégration satellite, puis nous filtrons les images pour l'année et la région choisies.
var embeddings = ee.ImageCollection('GOOGLE/SATELLITE_EMBEDDING/V1/ANNUAL');
var embeddingsFiltered = embeddings
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
Les images satellite intégrées sont divisées en tuiles et diffusées dans la projection des zones UTM pour la tuile. Nous obtenons ainsi plusieurs tuiles d'intégration satellite couvrant la région d'intérêt. Pour obtenir une seule image, nous devons les mosaïquer. Dans Earth Engine, une mosaïque d'images d'entrée est associée à la projection par défaut, qui est WGS84 avec une échelle de 1 degré. Comme nous agrégerons et reprojeterons cette mosaïque plus loin dans le tutoriel, il est utile de conserver la projection d'origine. Nous pouvons extraire les informations de projection de l'une des tuiles et les définir sur la mosaïque à l'aide de la fonction setDefaultProjection().
// Extract the projection of the first band of the first image
var embeddingsProjection = ee.Image(embeddingsFiltered.first()).select(0).projection();
// Set the projection of the mosaic to the extracted projection
var embeddingsImage = embeddingsFiltered.mosaic()
.setDefaultProjection(embeddingsProjection);
Préparer la mosaïque GEDI L4A
Étant donné que les estimations de biomasse GEDI seront utilisées pour entraîner notre modèle de régression, il est essentiel de filtrer les données GEDI non valides ou non fiables avant de les utiliser. Nous appliquons plusieurs masques pour supprimer les mesures potentiellement erronées.
- Supprimez toutes les mesures qui ne répondent pas aux exigences de qualité (l4_quality_flag = 0 et degrade_flag > 0).
- Supprimez toutes les mesures présentant une erreur relative élevée ("agbd_se" / "agbd" > 50 %).
- Supprimer toutes les mesures sur les pentes supérieures à 30 % en fonction du modèle numérique d'élévation (DEM) Copernicus GLO-30
Enfin, nous sélectionnons toutes les mesures restantes pour la période et la région qui nous intéressent, puis nous créons une mosaïque.
var gedi = ee.ImageCollection('LARSE/GEDI/GEDI04_A_002_MONTHLY');
// Function to select the highest quality GEDI data
var qualityMask = function(image) {
return image.updateMask(image.select('l4_quality_flag').eq(1))
.updateMask(image.select('degrade_flag').eq(0));
};
// Function to mask unreliable GEDI measurements
// with a relative standard error > 50%
// agbd_se / agbd > 0.5
var errorMask = function(image) {
var relative_se = image.select('agbd_se')
.divide(image.select('agbd'));
return image.updateMask(relative_se.lte(0.5));
};
// Function to mask GEDI measurements on slopes > 30%
var slopeMask = function(image) {
// Use Copernicus GLO-30 DEM for calculating slope
var glo30 = ee.ImageCollection('COPERNICUS/DEM/GLO30');
var glo30Filtered = glo30
.filter(ee.Filter.bounds(geometry))
.select('DEM');
// Extract the projection
var demProj = glo30Filtered.first().select(0).projection();
// The dataset consists of individual images
// Create a mosaic and set the projection
var elevation = glo30Filtered.mosaic().rename('dem')
.setDefaultProjection(demProj);
// Compute the slope
var slope = ee.Terrain.slope(elevation);
return image.updateMask(slope.lt(30));
};
var gediFiltered = gedi
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
var gediProjection = ee.Image(gediFiltered.first())
.select('agbd').projection();
var gediProcessed = gediFiltered
.map(qualityMask)
.map(errorMask)
.map(slopeMask);
var gediMosaic = gediProcessed.mosaic()
.select('agbd').setDefaultProjection(gediProjection);
// Visualize the GEDI Mosaic
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(gediMosaic, gediVis, 'GEDI L4A (Filtered)', false);

Figure : Observations de biomasse GEDI préparées
Rééchantillonner et agréger les entrées
Avant d'échantillonner les pixels pour entraîner un modèle de régression, nous rééchantillonnons et reprojetons les entrées sur la même grille de pixels. Les mesures GEDI ont une précision horizontale de +/- 9 m. Cela pose problème lorsque les valeurs AGB GEDI sont mises en correspondance avec les pixels d'embedding satellite. Pour résoudre ce problème, nous rééchantillonnons et agrégeons toutes les images d'entrée dans une grille de pixels plus grande avec des valeurs moyennes issues des pixels d'origine. Cela permet également de supprimer le bruit des données et de créer un meilleur modèle de machine learning.
// Choose the grid size and projection
var gridScale = 100;
var gridProjection = ee.Projection('EPSG:3857')
.atScale(gridScale);
// Create a stacked image with predictor and predicted variables
var stacked = embeddingsImage.addBands(gediMosaic);
// Set the resampling mode
var stacked = stacked.resample('bilinear');
// Aggregate pixels with 'mean' statistics
var stackedResampled = stacked
.reduceResolution({
reducer: ee.Reducer.mean(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// As larger GEDI pixels contain masked original
// pixels, it has a transparency mask.
// We update the mask to remove the transparency
var stackedResampled = stackedResampled
.updateMask(stackedResampled.mask().gt(0));
La reprojection et l'agrégation des pixels sont des opérations coûteuses. Il est conseillé d'exporter l'image empilée résultante en tant qu'élément et d'utiliser l'image précalculée dans les étapes suivantes. Cela vous aidera à surmonter les erreurs Computation timed out ou User memory exceeded lorsque vous travaillez avec de grandes régions.
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var mosaicExportImage = 'gedi_mosaic';
var mosaicExportImagePath = exportFolder + mosaicExportImage;
Export.image.toAsset({
image: stackedResampled.clip(geometry),
description: 'GEDI_Mosaic_Export',
assetId: mosaicExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
Démarrez la tâche d'exportation et attendez qu'elle se termine. Une fois cela fait, nous importons l'élément et continuons à créer le modèle.
// Use the exported asset
var stackedResampled = ee.Image(mosaicExportImagePath);
Extraire les caractéristiques d'entraînement
Nos données d'entrée sont prêtes pour l'extraction des caractéristiques d'entraînement. Nous utilisons les bandes d'embedding satellite comme variables dépendantes (prédicteurs) et les valeurs GEDI AGBD comme variable indépendante (prédite) dans le modèle de régression. Nous pouvons extraire les valeurs coïncidentes à chaque pixel et préparer notre ensemble de données d'entraînement. Notre image GEDI est principalement masquée et ne contient des valeurs que pour un petit sous-ensemble de pixels. Si nous utilisons sample(), la plupart des valeurs renvoyées seront vides. Pour surmonter ce problème, nous créons une bande de classe à partir du masque GEDI et utilisons stratifiedSample() pour nous assurer d'échantillonner à partir des pixels non masqués.
var predictors = embeddingsImage.bandNames();
var predicted = gediMosaic.bandNames().get(0);
print('predictors', predictors);
print('predicted', predicted);
var predictorImage = stackedResampled.select(predictors);
var predictedImage = stackedResampled.select([predicted]);
var classMask = predictedImage.mask().toInt().rename('class');
var numSamples = 1000;
// We set classPoints to [0, numSamples]
// This will give us 0 points for class 0 (masked areas)
// and numSample points for class 1 (non-masked areas)
var training = stackedResampled.addBands(classMask)
.stratifiedSample({
numPoints: numSamples,
classBand: 'class',
region: geometry,
scale: gridScale,
classValues: [0, 1],
classPoints: [0, numSamples],
dropNulls: true,
tileScale: 16,
});
print('Number of Features Extracted', training.size());
print('Sample Training Feature', training.first());
Entraîner un modèle de régression
Nous sommes maintenant prêts à entraîner le modèle. De nombreux classificateurs dans Earth Engine peuvent être utilisés pour les tâches de classification et de régression. Comme nous voulons prédire une valeur numérique (au lieu d'une classe), nous pouvons définir le classificateur pour qu'il s'exécute en mode REGRESSION et l'entraîner à l'aide des données d'entraînement. Une fois le modèle entraîné, nous pouvons comparer sa prédiction aux valeurs d'entrée et calculer la racine carrée de l'erreur quadratique moyenne (rmse) et le coefficient de corrélation r^2 pour vérifier les performances du modèle.
// Use the RandomForest classifier and set the
// output mode to REGRESSION
var model = ee.Classifier.smileRandomForest(50)
.setOutputMode('REGRESSION')
.train({
features: training,
classProperty: predicted,
inputProperties: predictors
});
// Get model's predictions for training samples
var predicted = training.classify({
classifier: model,
outputName: 'agbd_predicted'
});
// Calculate RMSE
var calculateRmse = function(input) {
var observed = ee.Array(
input.aggregate_array('agbd'));
var predicted = ee.Array(
input.aggregate_array('agbd_predicted'));
var rmse = observed.subtract(predicted).pow(2)
.reduce('mean', [0]).sqrt().get([0]);
return rmse;
};
var rmse = calculateRmse(predicted);
print('RMSE', rmse);
// Create a plot of observed vs. predicted values
var chart = ui.Chart.feature.byFeature({
features: predicted.select(['agbd', 'agbd_predicted']),
xProperty: 'agbd',
yProperties: ['agbd_predicted'],
}).setChartType('ScatterChart')
.setOptions({
title: 'Aboveground Biomass Density (Mg/Ha)',
dataOpacity: 0.8,
hAxis: {'title': 'Observed'},
vAxis: {'title': 'Predicted'},
legend: {position: 'right'},
series: {
0: {
visibleInLegend: false,
color: '#525252',
pointSize: 3,
pointShape: 'triangle',
},
},
trendlines: {
0: {
type: 'linear',
color: 'black',
lineWidth: 1,
pointSize: 0,
labelInLegend: 'Linear Fit',
visibleInLegend: true,
showR2: true
}
},
chartArea: {left: 100, bottom: 100, width: '50%'},
});
print(chart);
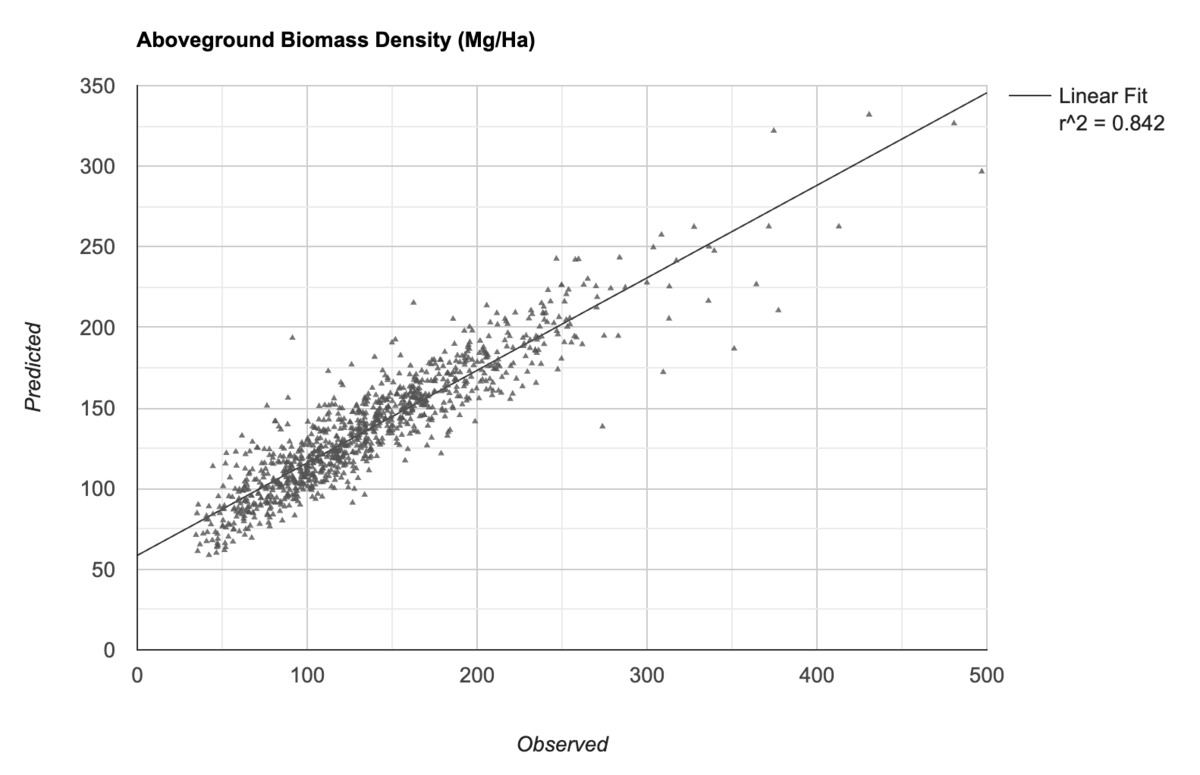
Figure : valeurs d'AGBD observées par rapport à celles prédites par le modèle
Générer des prédictions pour les valeurs inconnues
Une fois que nous sommes satisfaits du modèle, nous pouvons l'utiliser pour générer des prédictions à des emplacements inconnus à partir de l'image contenant des bandes de prédiction.
// We set the band name of the output image as 'agbd'
var predictedImage = stackedResampled.classify({
classifier: model,
outputName: 'agbd'
});
L'image contenant les valeurs de DABA prédites pour chaque pixel est maintenant prête à être exportée. Nous l'utiliserons dans la partie suivante pour visualiser les résultats.
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var predictedExportImage = 'predicted_agbd';
var predictedExportImagePath = exportFolder + predictedExportImage;
Export.image.toAsset({
image: predictedImage.clip(geometry),
description: 'Predicted_Image_Export',
assetId: predictedExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
Démarrez la tâche d'exportation et attendez qu'elle se termine. Une fois cela fait, nous importons le composant et visualisons les résultats.
var predictedImage = ee.Image(predictedExportImagePath);
// Visualize the image
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(predictedImage, gediVis, 'Predicted AGBD');

Figure : AGBD prédit. Plus la couleur verte est foncée, plus la densité de biomasse prévue est élevée.
Estimer la biomasse totale
Nous disposons désormais de valeurs AGBD prédites pour chaque pixel de l'image, qui peuvent être utilisées pour estimer le stock total de biomasse aérienne (AGB) dans la région. Mais nous devons d'abord supprimer tous les pixels appartenant aux zones non végétalisées. Nous pouvons utiliser l'ensemble de données sur la couverture terrestre ESA WorldCover et sélectionner les pixels végétalisés.
// GEDI data is processed only for certain landcovers
// from Plant Functional Types (PFT) classification
// https://doi.org/10.1029/2022EA002516
// Here we use ESA WorldCover v200 product to
// select landcovers representing vegetated areas
var worldcover = ee.ImageCollection('ESA/WorldCover/v200').first();
// Aggregate pixels to the same grid as other dataset
// with 'mode' value.
// i.e. The landcover with highest occurrence within the grid
var worldcoverResampled = worldcover
.reduceResolution({
reducer: ee.Reducer.mode(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// Select grids for the following classes
// | Class Name | Value |
// | Forests | 10 |
// | Shrubland | 20 |
// | Grassland | 30 |
// | Cropland | 40 |
// | Mangroves | 95 |
var landCoverMask = worldcoverResampled.eq(10)
.or(worldcoverResampled.eq(20))
.or(worldcoverResampled.eq(30))
.or(worldcoverResampled.eq(40))
.or(worldcoverResampled.eq(95));
var predictedImageMasked = predictedImage
.updateMask(landCoverMask);
Map.addLayer(predictedImageMasked, gediVis, 'Predicted AGBD (Masked)');

Figure : AGBD prédit avec les zones non végétalisées masquées
Les valeurs AGBD de GEDI sont exprimées en mégagrammes par hectare (Mg/ha). Pour obtenir l'AGB total, nous multiplions chaque pixel par sa superficie en hectares et additionnons leurs valeurs.
var pixelAreaHa = ee.Image.pixelArea().divide(10000);
var predictedAgb = predictedImageMasked.multiply(pixelAreaHa);
var stats = predictedAgb.reduceRegion({
reducer: ee.Reducer.sum(),
geometry: geometry,
scale: gridScale,
maxPixels: 1e10,
tileScale: 16
});
// Result is a dictionary with key for each band
var totalAgb = stats.getNumber('agbd');
print('Total AGB (Mg)', totalAgb);
Essayez le script complet de ce tutoriel dans l'éditeur de code Earth Engine.