Поля встраивания можно использовать в качестве входных данных/предикторов признаков для регрессии таким же образом, как они используются для классификации.
В этом уроке мы научимся использовать слои поля внедрения 64D в качестве входных данных для множественного регрессионного анализа, прогнозирующего надземную биомассу (AGB).
Миссия NASA по исследованию глобальной динамики экосистем (GEDI) собирает данные лидара вдоль наземных трансект с пространственным разрешением 30 м и интервалом 60 м. Мы будем использовать набор данных GEDI L4A Raster Aboveground Biomass Density, содержащий точечные оценки плотности надземной биомассы (AGBD), который будет использоваться в качестве прогнозируемой переменной в регрессионной модели.
Выберите регион
Начнём с определения интересующей области. В этом уроке мы выберем область в Западных Гатах (Индия) и определим полигон в качестве геометрической переменной. Кроме того, вы можете использовать инструменты рисования в редакторе кода, чтобы нарисовать полигон вокруг интересующей области, который будет сохранён как геометрическая переменная в импортируемых данных. Мы также используем базовую карту спутниковой съёмки, что упрощает обнаружение участков с растительностью.
var geometry = ee.Geometry.Polygon([[
[74.322, 14.981],
[74.322, 14.765],
[74.648, 14.765],
[74.648, 14.980]
]]);
// Use the satellite basemap
Map.setOptions('SATELLITE');

Рисунок: Выбор интересующей области для прогнозирования надземной биомассы
Выберите период времени
Выберите год, для которого мы хотим построить регрессию. Помните, что сателлитные эмбеддинги агрегируются с годовыми интервалами, поэтому мы определяем период для всего года.
var startDate = ee.Date.fromYMD(2022, 1, 1);
var endDate = startDate.advance(1, 'year');
Подготовьте набор данных для встраивания спутников
В качестве предиктора для регрессии будут использоваться 64-канальные изображения, полученные с помощью метода Satellite Embedding. Мы загружаем набор данных Satellite Embedding и фильтруем изображения для выбранного года и региона.
var embeddings = ee.ImageCollection('GOOGLE/SATELLITE_EMBEDDING/V1/ANNUAL');
var embeddingsFiltered = embeddings
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
Изображения, полученные с помощью Satellite Embedding, сгруппированы в тайлы и представлены в проекции зон UTM для каждого тайла. В результате мы получаем несколько тайлов, полученных с помощью Satellite Embedding, покрывающих интересующую область. Чтобы получить единое изображение, необходимо объединить их в мозаику. В Earth Engine мозаике входных изображений назначается проекция по умолчанию — WGS84 с масштабом 1 градус. Поскольку далее в этом руководстве мы будем объединять и перепроецировать эту мозаику, полезно сохранить исходную проекцию. Мы можем извлечь информацию о проекции из одного из тайлов и установить её для мозаики с помощью функции setDefaultProjection() .
// Extract the projection of the first band of the first image
var embeddingsProjection = ee.Image(embeddingsFiltered.first()).select(0).projection();
// Set the projection of the mosaic to the extracted projection
var embeddingsImage = embeddingsFiltered.mosaic()
.setDefaultProjection(embeddingsProjection);
Подготовьте мозаику GEDI L4A
Поскольку оценки биомассы GEDI будут использоваться для обучения нашей регрессионной модели, крайне важно отфильтровать недействительные или ненадёжные данные GEDI перед их использованием. Мы применяем несколько масок для удаления потенциально ошибочных измерений.
- Удалить все измерения, не соответствующие требованиям качества (l4_quality_flag = 0 и degrade_flag > 0)
- Удалить все измерения с высокой относительной погрешностью ('agbd_se' / 'agbd' > 50%).
- Удалить все измерения на уклонах > 30% на основе цифрового режима рельефа Copernicus GLO-30 (ЦМР)
Наконец, мы выбираем все оставшиеся измерения за интересующий нас период времени и регион и создаем мозаику.
var gedi = ee.ImageCollection('LARSE/GEDI/GEDI04_A_002_MONTHLY');
// Function to select the highest quality GEDI data
var qualityMask = function(image) {
return image.updateMask(image.select('l4_quality_flag').eq(1))
.updateMask(image.select('degrade_flag').eq(0));
};
// Function to mask unreliable GEDI measurements
// with a relative standard error > 50%
// agbd_se / agbd > 0.5
var errorMask = function(image) {
var relative_se = image.select('agbd_se')
.divide(image.select('agbd'));
return image.updateMask(relative_se.lte(0.5));
};
// Function to mask GEDI measurements on slopes > 30%
var slopeMask = function(image) {
// Use Copernicus GLO-30 DEM for calculating slope
var glo30 = ee.ImageCollection('COPERNICUS/DEM/GLO30');
var glo30Filtered = glo30
.filter(ee.Filter.bounds(geometry))
.select('DEM');
// Extract the projection
var demProj = glo30Filtered.first().select(0).projection();
// The dataset consists of individual images
// Create a mosaic and set the projection
var elevation = glo30Filtered.mosaic().rename('dem')
.setDefaultProjection(demProj);
// Compute the slope
var slope = ee.Terrain.slope(elevation);
return image.updateMask(slope.lt(30));
};
var gediFiltered = gedi
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
var gediProjection = ee.Image(gediFiltered.first())
.select('agbd').projection();
var gediProcessed = gediFiltered
.map(qualityMask)
.map(errorMask)
.map(slopeMask);
var gediMosaic = gediProcessed.mosaic()
.select('agbd').setDefaultProjection(gediProjection);
// Visualize the GEDI Mosaic
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(gediMosaic, gediVis, 'GEDI L4A (Filtered)', false);

Рисунок: Подготовленные данные наблюдений биомассы GEDI
Повторная выборка и агрегация входных данных
Перед выборкой пикселей для обучения регрессионной модели мы повторно выборим и перепроецируем входные данные на ту же пиксельную сетку. Измерения GEDI имеют горизонтальную точность +/- 9 м. Это создает проблемы при сопоставлении значений GEDI AGB с пикселями Satellite Embedding. Чтобы решить эту проблему, мы повторно выборим и агрегируем все входные изображения в более крупную пиксельную сетку со средними значениями исходных пикселей. Это также помогает устранить шум из данных и построить более эффективную модель машинного обучения.
// Choose the grid size and projection
var gridScale = 100;
var gridProjection = ee.Projection('EPSG:3857')
.atScale(gridScale);
// Create a stacked image with predictor and predicted variables
var stacked = embeddingsImage.addBands(gediMosaic);
// Set the resampling mode
var stacked = stacked.resample('bilinear');
// Aggregate pixels with 'mean' statistics
var stackedResampled = stacked
.reduceResolution({
reducer: ee.Reducer.mean(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// As larger GEDI pixels contain masked original
// pixels, it has a transparency mask.
// We update the mask to remove the transparency
var stackedResampled = stackedResampled
.updateMask(stackedResampled.mask().gt(0));
Перепроецирование и агрегация пикселей — это затратная операция, поэтому рекомендуется экспортировать полученное сложенное изображение как ресурс и использовать предварительно вычисленное изображение на последующих этапах. Это поможет избежать ошибок, связанных с истечением времени ожидания вычислений или нехваткой пользовательской памяти при работе с большими областями.
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var mosaicExportImage = 'gedi_mosaic';
var mosaicExportImagePath = exportFolder + mosaicExportImage;
Export.image.toAsset({
image: stackedResampled.clip(geometry),
description: 'GEDI_Mosaic_Export',
assetId: mosaicExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
Запускаем задачу экспорта и дожидаемся её завершения. После этого импортируем ассет и продолжаем построение модели.
// Use the exported asset
var stackedResampled = ee.Image(mosaicExportImagePath);
Извлечь особенности обучения
Наши входные данные готовы к извлечению обучающих признаков. Мы используем полосы вложений спутников в качестве зависимых переменных (предикторов), а значения GEDI AGBD — в качестве независимых переменных (предсказываемых) в регрессионной модели. Мы можем извлечь совпадающие значения для каждого пикселя и подготовить обучающий набор данных. Наше изображение GEDI в основном замаскировано и содержит значения лишь в небольшом подмножестве пикселей. При использовании sample() будут возвращены преимущественно пустые значения. Чтобы решить эту проблему, мы создаём полосу класса из маски GEDI и используем stratifiedSample() , чтобы обеспечить выборку из не замаскированных пикселей.
var predictors = embeddingsImage.bandNames();
var predicted = gediMosaic.bandNames().get(0);
print('predictors', predictors);
print('predicted', predicted);
var predictorImage = stackedResampled.select(predictors);
var predictedImage = stackedResampled.select([predicted]);
var classMask = predictedImage.mask().toInt().rename('class');
var numSamples = 1000;
// We set classPoints to [0, numSamples]
// This will give us 0 points for class 0 (masked areas)
// and numSample points for class 1 (non-masked areas)
var training = stackedResampled.addBands(classMask)
.stratifiedSample({
numPoints: numSamples,
classBand: 'class',
region: geometry,
scale: gridScale,
classValues: [0, 1],
classPoints: [0, numSamples],
dropNulls: true,
tileScale: 16,
});
print('Number of Features Extracted', training.size());
print('Sample Training Feature', training.first());
Обучить регрессионную модель
Теперь мы готовы к обучению модели. Многие классификаторы в Earth Engine можно использовать как для задач классификации, так и для задач регрессии . Поскольку мы хотим предсказать числовое значение (а не класс), мы можем настроить классификатор на работу в режиме REGRESSION и провести обучение на обучающих данных. После обучения модели мы можем сравнить её прогноз с входными значениями и вычислить среднеквадратичную ошибку ( rmse ) и коэффициент корреляции r^2 для проверки её эффективности.
// Use the RandomForest classifier and set the
// output mode to REGRESSION
var model = ee.Classifier.smileRandomForest(50)
.setOutputMode('REGRESSION')
.train({
features: training,
classProperty: predicted,
inputProperties: predictors
});
// Get model's predictions for training samples
var predicted = training.classify({
classifier: model,
outputName: 'agbd_predicted'
});
// Calculate RMSE
var calculateRmse = function(input) {
var observed = ee.Array(
input.aggregate_array('agbd'));
var predicted = ee.Array(
input.aggregate_array('agbd_predicted'));
var rmse = observed.subtract(predicted).pow(2)
.reduce('mean', [0]).sqrt().get([0]);
return rmse;
};
var rmse = calculateRmse(predicted);
print('RMSE', rmse);
// Create a plot of observed vs. predicted values
var chart = ui.Chart.feature.byFeature({
features: predicted.select(['agbd', 'agbd_predicted']),
xProperty: 'agbd',
yProperties: ['agbd_predicted'],
}).setChartType('ScatterChart')
.setOptions({
title: 'Aboveground Biomass Density (Mg/Ha)',
dataOpacity: 0.8,
hAxis: {'title': 'Observed'},
vAxis: {'title': 'Predicted'},
legend: {position: 'right'},
series: {
0: {
visibleInLegend: false,
color: '#525252',
pointSize: 3,
pointShape: 'triangle',
},
},
trendlines: {
0: {
type: 'linear',
color: 'black',
lineWidth: 1,
pointSize: 0,
labelInLegend: 'Linear Fit',
visibleInLegend: true,
showR2: true
}
},
chartArea: {left: 100, bottom: 100, width: '50%'},
});
print(chart);
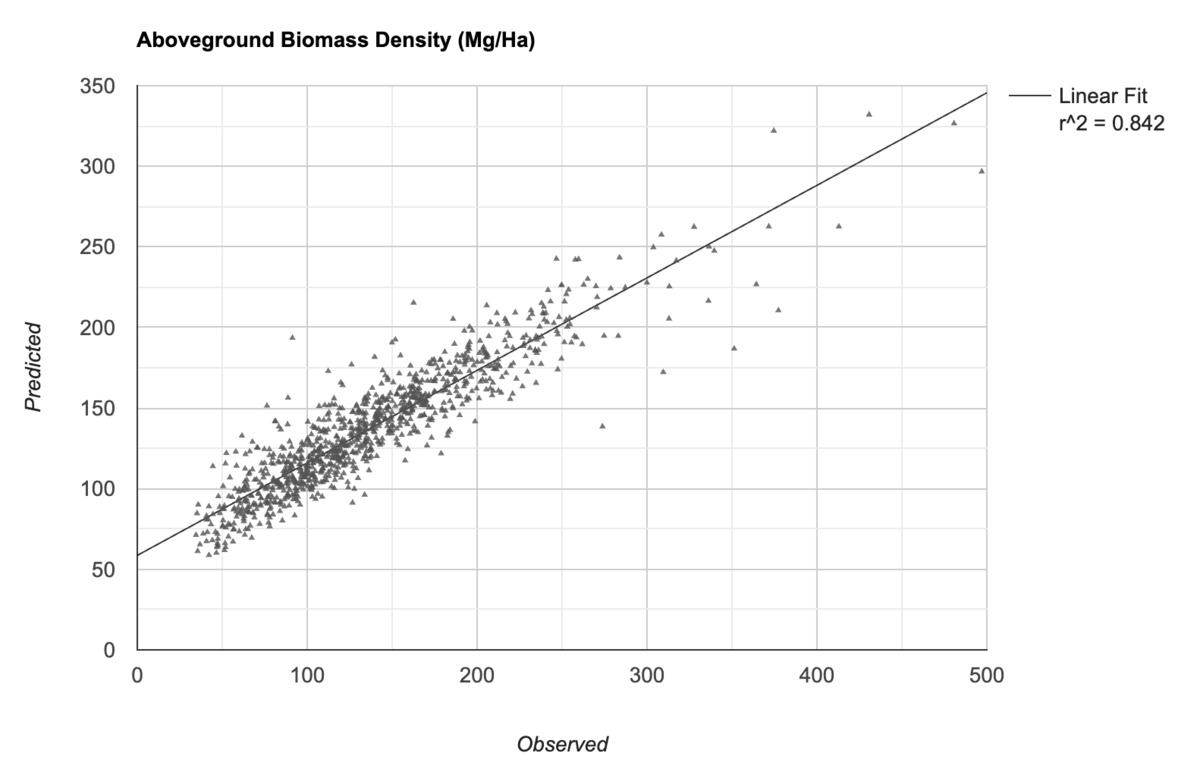
Рисунок: Наблюдаемые и предсказанные моделью значения AGBD
Генерация прогнозов для неизвестных значений
Как только мы будем удовлетворены моделью, мы сможем использовать обученную модель для генерации прогнозов в неизвестных местах на основе изображения, содержащего предикторные полосы.
// We set the band name of the output image as 'agbd'
var predictedImage = stackedResampled.classify({
classifier: model,
outputName: 'agbd'
});
Изображение, содержащее предсказанные значения AGBD для каждого пикселя, теперь готово к экспорту. Мы используем его в следующей части для визуализации результатов.
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var predictedExportImage = 'predicted_agbd';
var predictedExportImagePath = exportFolder + predictedExportImage;
Export.image.toAsset({
image: predictedImage.clip(geometry),
description: 'Predicted_Image_Export',
assetId: predictedExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
Запускаем задачу экспорта и дожидаемся её завершения. После завершения импортируем актив и визуализируем результаты.
var predictedImage = ee.Image(predictedExportImagePath);
// Visualize the image
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(predictedImage, gediVis, 'Predicted AGBD');

Рисунок: Прогнозируемая AGBD. Более тёмный зелёный цвет соответствует более высокой прогнозируемой плотности биомассы.
Оценить общую биомассу
Теперь у нас есть прогнозируемые значения AGBD для каждого пикселя изображения, которые можно использовать для оценки общего запаса надземной биомассы (AGB) в регионе. Но сначала необходимо удалить все пиксели, относящиеся к участкам без растительности. Мы можем использовать набор данных ESA WorldCover о ландшафтном покрове и выбрать пиксели с растительностью.
// GEDI data is processed only for certain landcovers
// from Plant Functional Types (PFT) classification
// https://doi.org/10.1029/2022EA002516
// Here we use ESA WorldCover v200 product to
// select landcovers representing vegetated areas
var worldcover = ee.ImageCollection('ESA/WorldCover/v200').first();
// Aggregate pixels to the same grid as other dataset
// with 'mode' value.
// i.e. The landcover with highest occurrence within the grid
var worldcoverResampled = worldcover
.reduceResolution({
reducer: ee.Reducer.mode(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// Select grids for the following classes
// | Class Name | Value |
// | Forests | 10 |
// | Shrubland | 20 |
// | Grassland | 30 |
// | Cropland | 40 |
// | Mangroves | 95 |
var landCoverMask = worldcoverResampled.eq(10)
.or(worldcoverResampled.eq(20))
.or(worldcoverResampled.eq(30))
.or(worldcoverResampled.eq(40))
.or(worldcoverResampled.eq(95));
var predictedImageMasked = predictedImage
.updateMask(landCoverMask);
Map.addLayer(predictedImageMasked, gediVis, 'Predicted AGBD (Masked)');

Рисунок: Прогнозируемое AGBD с замаскированными не покрытыми растительностью участками
Единицы измерения значений GEDI AGBD — мегаграммы на гектар (Мг/га). Чтобы получить общий показатель AGB, мы умножаем каждый пиксель на его площадь в гектарах и суммируем их значения.
var pixelAreaHa = ee.Image.pixelArea().divide(10000);
var predictedAgb = predictedImageMasked.multiply(pixelAreaHa);
var stats = predictedAgb.reduceRegion({
reducer: ee.Reducer.sum(),
geometry: geometry,
scale: gridScale,
maxPixels: 1e10,
tileScale: 16
});
// Result is a dictionary with key for each band
var totalAgb = stats.getNumber('agbd');
print('Total AGB (Mg)', totalAgb);
Попробуйте полный сценарий для этого урока в редакторе кода Earth Engine .
