در این آموزش متدولوژی مدل سازی توزیع گونه ها با استفاده از موتور گوگل ارث معرفی می شود. مروری کوتاه بر مدلسازی پراکنش گونهها ارائه خواهد شد و به دنبال آن فرآیند پیشبینی و تجزیه و تحلیل زیستگاه گونههای پرنده در حال انقراض معروف به Fairy pitta (نام علمی: Pitta nympha ) ارائه میشود.
اول منو بدو
سلول زیر را برای مقداردهی اولیه API اجرا کنید. خروجی حاوی دستورالعمل هایی در مورد نحوه دسترسی این نوت بوک به Earth Engine با استفاده از حساب شما خواهد بود.
import ee
# Trigger the authentication flow.
ee.Authenticate()
# Initialize the library.
ee.Initialize(project='my-project')
مروری کوتاه بر مدلسازی توزیع گونهها
بیایید بررسی کنیم که مدلهای توزیع گونهها چیست، مزایای استفاده از Google Earth Engine برای پردازش آنها، دادههای مورد نیاز برای مدلها، و نحوه ساختار گردش کار.
مدل سازی توزیع گونه ها چیست؟
مدلسازی توزیع گونهها (SDM در زیر) رایجترین روش مورد استفاده برای تخمین پراکنش جغرافیایی واقعی یا بالقوه یک گونه است. این شامل توصیف شرایط محیطی مناسب برای یک گونه خاص و سپس شناسایی این شرایط مناسب از نظر جغرافیایی است.
SDM به عنوان یک جزء حیاتی در برنامه ریزی حفاظت در سال های اخیر ظهور کرده است و تکنیک های مدل سازی مختلفی برای این منظور توسعه داده شده است. پیادهسازی SDM در Google Earth Engine (زیر GEE) دسترسی آسان به دادههای محیطی در مقیاس بزرگ را همراه با قابلیتهای محاسباتی قدرتمند و پشتیبانی از الگوریتمهای یادگیری ماشین فراهم میکند که امکان مدلسازی سریع را فراهم میکند.
داده های مورد نیاز برای SDM
SDM معمولاً از رابطه بین سوابق وقوع گونه های شناخته شده و متغیرهای محیطی برای شناسایی شرایطی استفاده می کند که تحت آن یک جمعیت می تواند پایدار بماند. به عبارت دیگر، دو نوع داده ورودی مدل مورد نیاز است:
- سوابق وقوع گونه های شناخته شده
- متغیرهای مختلف محیطی
این داده ها برای شناسایی شرایط محیطی مرتبط با حضور گونه ها به الگوریتم ها وارد می شوند.
گردش کار SDM با استفاده از GEE
گردش کار برای SDM با استفاده از GEE به شرح زیر است:
- جمع آوری و پیش پردازش داده های وقوع گونه ها
- تعریف منطقه مورد علاقه
- اضافه شدن متغیرهای محیطی GEE
- تولید داده های شبه غیبت
- برازش و پیش بینی مدل
- ارزیابی اهمیت و دقت متغیر
پیش بینی و تحلیل زیستگاه با استفاده از GEE
فیری پیتا ( Pitta nympha ) به عنوان مطالعه موردی برای نشان دادن کاربرد SDM مبتنی بر GEE استفاده خواهد شد. در حالی که این گونه خاص برای یک مثال انتخاب شده است، محققان می توانند این روش را برای هر گونه هدف مورد علاقه با تغییرات جزئی در کد منبع ارائه شده اعمال کنند.
پری پیتا یک مهاجر تابستانی نادر و مهاجر در کره جنوبی است که به دلیل گرم شدن آب و هوای اخیر در شبه جزیره کره، منطقه پراکنش آن در حال گسترش است. این گونه به عنوان یک گونه نادر، حیات وحش در خطر انقراض کلاس II، بنای یادبود طبیعی شماره 204، به عنوان منطقه منقرض شده (RE) در فهرست قرمز ملی، و آسیب پذیر (VU) بر اساس طبقه بندی IUCN طبقه بندی شده است.
به نظر می رسد انجام SDM برای برنامه ریزی حفاظت از Fairy pitta بسیار ارزشمند است. اکنون بیایید پیشبینی و تحلیل زیستگاه را از طریق GEE ادامه دهیم.
ابتدا، کتابخانههای پایتون وارد میشوند. دستور import کل محتویات یک ماژول را وارد میکند، در حالی که دستور from import امکان وارد کردن اشیاء خاص از یک ماژول را میدهد.
# Import libraries
import geemap
import geemap.colormaps as cm
import pandas as pd, geopandas as gpd
import numpy as np, matplotlib.pyplot as plt
import os, requests, math, random
from ipyleaflet import TileLayer
from statsmodels.stats.outliers_influence import variance_inflation_factor
جمع آوری و پیش پردازش داده های وقوع گونه ها
اکنون، بیایید دادههای وقوع را برای Fairy pitta جمعآوری کنیم. حتی اگر در حال حاضر به دادههای وقوع گونههای مورد علاقه دسترسی ندارید، میتوانید دادههای مشاهدهای در مورد گونههای خاص از طریق GBIF API به دست آورید. GBIF API رابطی است که امکان دسترسی به داده های توزیع گونه های ارائه شده توسط GBIF را فراهم می کند و کاربران را قادر می سازد تا داده ها را جستجو، فیلتر و دانلود کنند و همچنین اطلاعات مختلف مربوط به گونه ها را به دست آورند.
در کد زیر به متغیر species_name نام علمی گونه اختصاص داده شده است (مثلا Pitta nympha برای Fairy pitta) و متغیر country_code کد کشور (مثلا KR برای کره جنوبی) اختصاص داده شده است. متغیر base_url آدرس GBIF API را ذخیره می کند. params یک فرهنگ لغت حاوی پارامترهایی است که در درخواست API استفاده می شود:
-
scientificName: نام علمی گونه مورد جستجو را تنظیم می کند. -
country: جستجو را به یک کشور خاص محدود می کند. -
hasCoordinate: اطمینان حاصل می کند که فقط داده های دارای مختصات (درست) جستجو می شوند. -
basisOfRecord: فقط سوابق مشاهدات انسانی را انتخاب می کند (HUMAN_OBSERVATION). -
limit: حداکثر تعداد نتایج بازگشتی را روی 10000 تنظیم می کند.
def get_gbif_species_data(species_name, country_code):
"""
Retrieves observational data for a specific species using the GBIF API and returns it as a pandas DataFrame.
Parameters:
species_name (str): The scientific name of the species to query.
country_code (str): The country code of the where the observation data will be queried.
Returns:
pd.DataFrame: A pandas DataFrame containing the observational data.
"""
base_url = "https://api.gbif.org/v1/occurrence/search"
params = {
"scientificName": species_name,
"country": country_code,
"hasCoordinate": "true",
"basisOfRecord": "HUMAN_OBSERVATION",
"limit": 10000,
}
try:
response = requests.get(base_url, params=params)
response.raise_for_status() # Raises an exception for a response error.
data = response.json()
occurrences = data.get("results", [])
if occurrences: # If data is present
df = pd.json_normalize(occurrences)
return df
else:
print("No data found for the given species and country code.")
return pd.DataFrame() # Returns an empty DataFrame
except requests.RequestException as e:
print(f"Request failed: {e}")
return pd.DataFrame() # Returns an empty DataFrame in case of an exception
با استفاده از پارامترهای تنظیم شده قبلی، از API GBIF برای سوابق مشاهده ای Fairy pitta ( Pitta nympha ) پرس و جو می کنیم و نتایج را در یک DataFrame بارگذاری می کنیم تا ردیف اول را بررسی کنیم. DataFrame یک ساختار داده برای مدیریت داده های با فرمت جدول است که از سطرها و ستون ها تشکیل شده است. در صورت لزوم، DataFrame را می توان به عنوان یک فایل CSV ذخیره کرد و دوباره در آن خواند.
# Retrieve Fairy Pitta data
df = get_gbif_species_data("Pitta nympha", "KR")
"""
# Save DataFrame to CSV and read back in.
df.to_csv("pitta_nympha_data.csv", index=False)
df = pd.read_csv("pitta_nympha_data.csv")
"""
df.head(1) # Display the first row of the DataFrame
سپس DataFrame را به یک GeoDataFrame تبدیل می کنیم که شامل ستونی برای اطلاعات جغرافیایی ( geometry ) می شود و ردیف اول را بررسی می کنیم. یک GeoDataFrame را می توان به عنوان یک فایل GeoPackage (*.gpkg) ذخیره کرد و دوباره در آن خواند.
# Convert DataFrame to GeoDataFrame
gdf = gpd.GeoDataFrame(
df,
geometry=gpd.points_from_xy(df.decimalLongitude,
df.decimalLatitude),
crs="EPSG:4326"
)[["species", "year", "month", "geometry"]]
"""
# Convert GeoDataFrame to GeoPackage (requires pycrs module)
%pip install -U -q pycrs
gdf.to_file("pitta_nympha_data.gpkg", driver="GPKG")
gdf = gpd.read_file("pitta_nympha_data.gpkg")
"""
gdf.head(1) # Display the first row of the GeoDataFrame
این بار ما تابعی ایجاد کردهایم که توزیع دادهها را بر اساس سال و ماه از GeoDataFrame تجسم کرده و به صورت نمودار نمایش میدهیم که میتوان آن را بهعنوان یک فایل تصویری ذخیره کرد. استفاده از نقشه حرارتی به ما این امکان را می دهد که به سرعت فراوانی وقوع گونه ها را بر اساس سال و ماه درک کنیم و تصویری بصری از تغییرات زمانی و الگوهای موجود در داده ها ارائه کنیم. این امکان را برای شناسایی الگوهای زمانی و تغییرات فصلی در دادههای وقوع گونهها و همچنین تشخیص سریع موارد پرت یا کیفیت در دادهها فراهم میکند.
# Yearly and monthly data distribution heatmap
def plot_heatmap(gdf, h_size=8):
statistics = gdf.groupby(["month", "year"]).size().unstack(fill_value=0)
# Heatmap
plt.figure(figsize=(h_size, h_size - 6))
heatmap = plt.imshow(
statistics.values, cmap="YlOrBr", origin="upper", aspect="auto"
)
# Display values above each pixel
for i in range(len(statistics.index)):
for j in range(len(statistics.columns)):
plt.text(
j, i, statistics.values[i, j], ha="center", va="center", color="black"
)
plt.colorbar(heatmap, label="Count")
plt.title("Monthly Species Count by Year")
plt.xlabel("Year")
plt.ylabel("Month")
plt.xticks(range(len(statistics.columns)), statistics.columns)
plt.yticks(range(len(statistics.index)), statistics.index)
plt.tight_layout()
plt.savefig("heatmap_plot.png")
plt.show()
plot_heatmap(gdf)
داده های سال 95 بسیار پراکنده و دارای شکاف های قابل توجهی نسبت به سال های دیگر است و ماه های مرداد و شهریور نیز نمونه های محدودی دارند و ویژگی های فصلی متفاوتی را نسبت به سایر دوره ها نشان می دهند. حذف این داده ها می تواند به بهبود پایداری و قدرت پیش بینی مدل کمک کند.
با این حال، توجه به این نکته مهم است که حذف داده ها ممکن است توانایی تعمیم مدل را افزایش دهد، اما همچنین می تواند منجر به از دست رفتن اطلاعات ارزشمند مرتبط با اهداف تحقیق شود. بنابراین، چنین تصمیماتی باید با دقت نظر گرفته شود.
# Filtering data by year and month
filtered_gdf = gdf[
(~gdf['year'].eq(1995)) &
(~gdf['month'].between(8, 9))
]
اکنون، GeoDataFrame فیلتر شده به یک شیء موتور Google Earth تبدیل می شود.
# Convert GeoDataFrame to Earth Engine object
data_raw = geemap.geopandas_to_ee(filtered_gdf)
در مرحله بعد، اندازه پیکسل شطرنجی نتایج SDM را به صورت وضوح 1 کیلومتر تعریف می کنیم.
# Spatial resolution setting (meters)
grain_size = 1000
هنگامی که چندین نقطه وقوع در یک پیکسل شطرنجی با وضوح 1 کیلومتر وجود دارد، احتمال زیادی وجود دارد که شرایط محیطی مشابهی در یک مکان جغرافیایی یکسان داشته باشند. استفاده از چنین داده هایی به طور مستقیم در تجزیه و تحلیل می تواند سوگیری را در نتایج ایجاد کند.
به عبارت دیگر، ما باید تأثیر بالقوه سوگیری نمونهگیری جغرافیایی را محدود کنیم. برای دستیابی به این هدف، ما فقط یک مکان را در هر پیکسل 1 کیلومتری حفظ می کنیم و بقیه را حذف می کنیم و به مدل اجازه می دهیم شرایط محیطی را به طور عینی تری منعکس کند.
def remove_duplicates(data, grain_size):
# Select one occurrence record per pixel at the chosen spatial resolution
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
rand_point_vals = random_raster.sampleRegions(
collection=ee.FeatureCollection(data), geometries=True
)
return rand_point_vals.distinct("random")
data = remove_duplicates(data_raw, grain_size)
# Before selection and after selection
print("Original data size:", data_raw.size().getInfo())
print("Final data size:", data.size().getInfo())
تجسم مقایسه سوگیری نمونه برداری جغرافیایی قبل از پیش پردازش (به رنگ آبی) و پس از پیش پردازش (به رنگ قرمز) در زیر نشان داده شده است. برای تسهیل مقایسه، نقشه بر روی منطقه با غلظت بالایی از مختصات وقوع فیری پیتا در پارک ملی هالاسان متمرکز شده است.
# Visualization of geographic sampling bias before (blue) and after (red) preprocessing
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
# Add the random raster layer
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
Map.addLayer(
random_raster,
{"min": 0, "max": 1, "palette": ["black", "white"], "opacity": 0.5},
"Random Raster",
)
# Add the original data layer in blue
Map.addLayer(data_raw, {"color": "blue"}, "Original data")
# Add the final data layer in red
Map.addLayer(data, {"color": "red"}, "Final data")
# Set the center of the map to the coordinates
Map.setCenter(126.712, 33.516, 14)
Map
تعریف منطقه مورد علاقه
تعریف منطقه مورد علاقه (AOI زیر) به اصطلاحی است که توسط محققان برای نشان دادن منطقه جغرافیایی که می خواهند تجزیه و تحلیل کنند، استفاده می کنند. معنایی مشابه با عبارت Study Area دارد.
در این زمینه، جعبه مرزی هندسه لایه نقطه وقوع را به دست آوردیم و یک بافر 50 کیلومتری در اطراف آن (با حداکثر تحمل 1000 متر) برای تعریف AOI ایجاد کردیم.
# Define the AOI
aoi = data.geometry().bounds().buffer(distance=50000, maxError=1000)
# Add the AOI to the map
outline = ee.Image().byte().paint(
featureCollection=aoi, color=1, width=3)
Map.remove_layer("Random Raster")
Map.addLayer(outline, {'palette': 'FF0000'}, "AOI")
Map.centerObject(aoi, 6)
Map
اضافه شدن متغیرهای محیطی GEE
حالا بیایید متغیرهای محیطی را به تحلیل اضافه کنیم. GEE طیف گسترده ای از مجموعه داده ها را برای متغیرهای محیطی مانند دما، بارش، ارتفاع، پوشش زمین و زمین فراهم می کند. این مجموعه داده ها ما را قادر می سازد تا به طور جامع عوامل مختلفی را که ممکن است بر ترجیحات زیستگاهی Fairy pitta تأثیر بگذارد، تجزیه و تحلیل کنیم.
انتخاب متغیرهای محیطی GEE در SDM باید منعکس کننده ویژگی های ترجیح زیستگاه گونه باشد. برای انجام این کار، تحقیقات قبلی و بررسی ادبیات در مورد ترجیحات زیستگاهی Fairy pitta باید انجام شود. این آموزش در درجه اول بر روی گردش کار SDM با استفاده از GEE تمرکز دارد، بنابراین برخی از جزئیات عمیق حذف شده اند.
WorldClim V1 Bioclim : این مجموعه داده 19 متغیر زیست اقلیم را ارائه می دهد که از داده های دما و بارش ماهانه مشتق شده است. بازه زمانی 1960 تا 1991 را پوشش می دهد و وضوح 927.67 متر دارد.
# WorldClim V1 Bioclim
bio = ee.Image("WORLDCLIM/V1/BIO")
ارتفاع دیجیتال ناسا SRTM 30 متر : این مجموعه داده حاوی داده های ارتفاعی دیجیتال از ماموریت توپوگرافی رادار شاتل (SRTM) است. داده ها اساساً در حدود سال 2000 جمع آوری شده و با وضوح تقریبی 30 متر (1 قوس در ثانیه) ارائه شده است. کد زیر لایه های ارتفاع، شیب، جنبه و سایه تپه را از داده های SRTM محاسبه می کند.
# NASA SRTM Digital Elevation 30m
terrain = ee.Algorithms.Terrain(ee.Image("USGS/SRTMGL1_003"))
تغییر پوشش جنگلی جهانی (GFCC) پوشش درختی چند ساله جهانی 30 متر : مجموعه داده های مزارع پیوسته گیاهی (VCF) از Landsat نسبت پوشش گیاهی پیش بینی شده عمودی را زمانی که ارتفاع پوشش گیاهی بیشتر از 5 متر است، تخمین می زند. این مجموعه داده برای چهار دوره زمانی با محوریت سال های 2000، 2005، 2010 و 2015 با وضوح 30 متر ارائه شده است. در اینجا، مقادیر میانه از این چهار دوره زمانی استفاده می شود.
# Global Forest Cover Change (GFCC) Tree Cover Multi-Year Global 30m
tcc = ee.ImageCollection("NASA/MEASURES/GFCC/TC/v3")
median_tcc = (
tcc.filterDate("2000-01-01", "2015-12-31")
.select(["tree_canopy_cover"], ["TCC"])
.median()
)
bio (متغیرهای Bioclimatic)، terrain (توپوگرافی) و median_tcc (پوشش تاج درخت) در یک تصویر چند باندی واحد ترکیب شدهاند. نوار elevation از terrain انتخاب می شود و برای مکان هایی که elevation بیشتر از 0 است یک watermask ایجاد می شود. این امر مناطق زیر سطح دریا (به عنوان مثال اقیانوس) را پوشانده و محقق را آماده می کند تا عوامل محیطی مختلف را برای AOI به طور جامع تجزیه و تحلیل کند.
# Combine bands into a multi-band image
predictors = bio.addBands(terrain).addBands(median_tcc)
# Create a water mask
watermask = terrain.select('elevation').gt(0)
# Mask out ocean pixels and clip to the area of interest
predictors = predictors.updateMask(watermask).clip(aoi)
زمانی که متغیرهای پیشبینیکننده بسیار همبسته با هم در یک مدل گنجانده میشوند، مسائل چند خطی میتواند ایجاد شود. چند خطی بودن پدیده ای است که زمانی رخ می دهد که روابط خطی قوی بین متغیرهای مستقل در یک مدل وجود داشته باشد که منجر به بی ثباتی در برآورد ضرایب (وزن) مدل می شود. این بیثباتی میتواند قابلیت اطمینان مدل را کاهش دهد و پیشبینیها یا تفسیرهایی را برای چالشهای دادههای جدید ایجاد کند. بنابراین، چند خطی را در نظر می گیریم و فرآیند انتخاب متغیرهای پیش بینی را ادامه می دهیم.
ابتدا 5000 نقطه تصادفی تولید می کنیم و سپس مقادیر متغیر پیش بینی کننده تصویر تک باندی را در آن نقاط استخراج می کنیم.
# Generate 5,000 random points
data_cor = predictors.sample(scale=grain_size, numPixels=5000, geometries=True)
# Extract predictor variable values
pvals = predictors.sampleRegions(collection=data_cor, scale=grain_size)
مقادیر پیش بینی استخراج شده برای هر نقطه را به DataFrame تبدیل می کنیم و سپس ردیف اول را بررسی می کنیم.
# Converting predictor values from Earth Engine to a DataFrame
pvals_df = geemap.ee_to_df(pvals)
pvals_df.head(1)
# Displaying the columns
columns = pvals_df.columns
print(columns)
محاسبه ضرایب همبستگی اسپیرمن بین متغیرهای پیش بینی داده شده و تجسم آنها در یک نقشه حرارتی.
def plot_correlation_heatmap(dataframe, h_size=10, show_labels=False):
# Calculate Spearman correlation coefficients
correlation_matrix = dataframe.corr(method="spearman")
# Create a heatmap
plt.figure(figsize=(h_size, h_size-2))
plt.imshow(correlation_matrix, cmap='coolwarm', interpolation='nearest')
# Optionally display values on the heatmap
if show_labels:
for i in range(correlation_matrix.shape[0]):
for j in range(correlation_matrix.shape[1]):
plt.text(j, i, f"{correlation_matrix.iloc[i, j]:.2f}",
ha='center', va='center', color='white', fontsize=8)
columns = dataframe.columns.tolist()
plt.xticks(range(len(columns)), columns, rotation=90)
plt.yticks(range(len(columns)), columns)
plt.title("Variables Correlation Matrix")
plt.colorbar(label="Spearman Correlation")
plt.savefig('correlation_heatmap_plot.png')
plt.show()
# Plot the correlation heatmap of variables
plot_correlation_heatmap(pvals_df)
ضریب همبستگی اسپیرمن برای درک ارتباط کلی بین متغیرهای پیشبینیکننده مفید است، اما مستقیماً چگونگی تعامل چندین متغیر را ارزیابی نمیکند، بهویژه تشخیص چند خطی بودن.
عامل تورم واریانس (VIF زیر) یک معیار آماری است که برای ارزیابی چند خطی بودن و هدایت انتخاب متغیر استفاده میشود. این نشان دهنده میزان رابطه خطی هر متغیر مستقل با سایر متغیرهای مستقل است و مقادیر بالای VIF می تواند شاهدی بر چند خطی بودن باشد.
به طور معمول، هنگامی که مقادیر VIF از 5 یا 10 تجاوز می کند، نشان می دهد که متغیر دارای همبستگی قوی با سایر متغیرها است که به طور بالقوه ثبات و تفسیرپذیری مدل را به خطر می اندازد. در این آموزش از معیار مقادیر VIF کمتر از 10 برای انتخاب متغیر استفاده شده است. 6 متغیر زیر بر اساس VIF انتخاب شدند.
# Filter variables based on Variance Inflation Factor (VIF)
def filter_variables_by_vif(dataframe, threshold=10):
original_columns = dataframe.columns.tolist()
remaining_columns = original_columns[:]
while True:
vif_data = dataframe[remaining_columns]
vif_values = [
variance_inflation_factor(vif_data.values, i)
for i in range(vif_data.shape[1])
]
max_vif_index = vif_values.index(max(vif_values))
max_vif = max(vif_values)
if max_vif < threshold:
break
print(f"Removing '{remaining_columns[max_vif_index]}' with VIF {max_vif:.2f}")
del remaining_columns[max_vif_index]
filtered_data = dataframe[remaining_columns]
bands = filtered_data.columns.tolist()
print("Bands:", bands)
return filtered_data, bands
filtered_pvals_df, bands = filter_variables_by_vif(pvals_df)
# Variable Selection Based on VIF
predictors = predictors.select(bands)
# Plot the correlation heatmap of variables
plot_correlation_heatmap(filtered_pvals_df, h_size=6, show_labels=True)
در مرحله بعد، بیایید 6 متغیر پیش بینی انتخاب شده را روی نقشه تجسم کنیم. 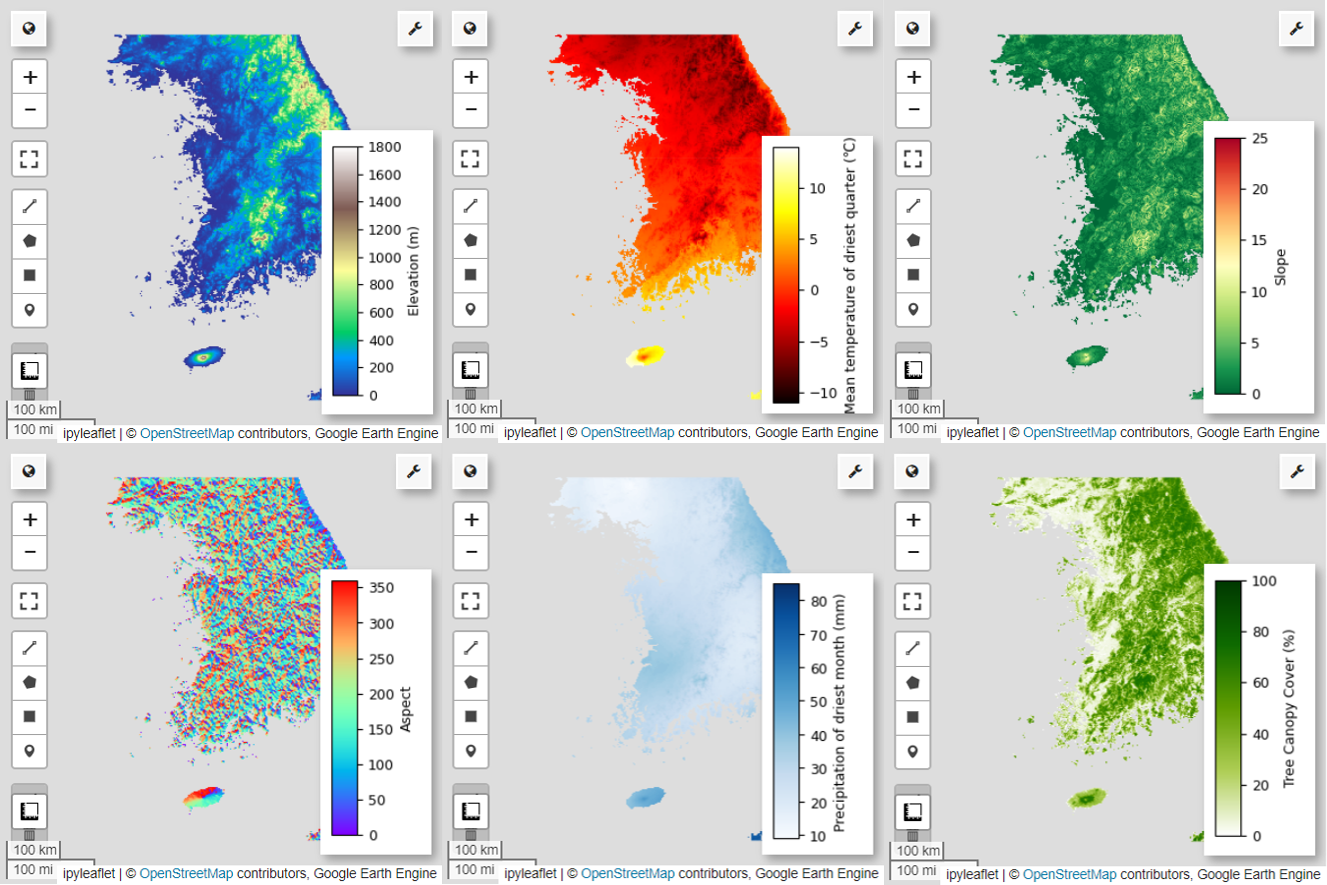
با استفاده از کد زیر می توانید پالت های موجود برای تجسم نقشه را بررسی کنید. برای مثال، پالت terrain به این شکل است. cm.plot_colormaps(width=8.0, height=0.2)
cm.plot_colormap('terrain', width=8.0, height=0.2, orientation='horizontal')
# Elevation layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['elevation'], 'min': 0, 'max': 1800, 'palette': cm.palettes.terrain}
Map.addLayer(predictors, vis_params, 'elevation')
Map.add_colorbar(vis_params, label="Elevation (m)", orientation="vertical", layer_name="elevation")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio09
min_max_val = (
predictors.select("bio09")
.multiply(0.1)
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio09 (Mean temperature of driest quarter) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"min": math.floor(min_max_val["bio09_min"]),
"max": math.ceil(min_max_val["bio09_max"]),
"palette": cm.palettes.hot,
}
Map.addLayer(predictors.select("bio09").multiply(0.1), vis_params, "bio09")
Map.add_colorbar(
vis_params,
label="Mean temperature of driest quarter (℃)",
orientation="vertical",
layer_name="bio09",
)
Map.centerObject(aoi, 6)
Map
# Slope layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['slope'], 'min': 0, 'max': 25, 'palette': cm.palettes.RdYlGn_r}
Map.addLayer(predictors, vis_params, 'slope')
Map.add_colorbar(vis_params, label="Slope", orientation="vertical", layer_name="slope")
Map.centerObject(aoi, 6)
Map
# Aspect layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['aspect'], 'min': 0, 'max': 360, 'palette': cm.palettes.rainbow}
Map.addLayer(predictors, vis_params, 'aspect')
Map.add_colorbar(vis_params, label="Aspect", orientation="vertical", layer_name="aspect")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio14
min_max_val = (
predictors.select("bio14")
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio14 (Precipitation of driest month) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["bio14"],
"min": math.floor(min_max_val["bio14_min"]),
"max": math.ceil(min_max_val["bio14_max"]),
"palette": cm.palettes.Blues,
}
Map.addLayer(predictors, vis_params, "bio14")
Map.add_colorbar(
vis_params,
label="Precipitation of driest month (mm)",
orientation="vertical",
layer_name="bio14",
)
Map.centerObject(aoi, 6)
Map
# TCC layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["TCC"],
"min": 0,
"max": 100,
"palette": ["ffffff", "afce56", "5f9c00", "0e6a00", "003800"],
}
Map.addLayer(predictors, vis_params, "TCC")
Map.add_colorbar(
vis_params, label="Tree Canopy Cover (%)", orientation="vertical", layer_name="TCC"
)
Map.centerObject(aoi, 6)
Map
تولید داده های شبه غیبت
در فرآیند SDM، انتخاب داده های ورودی برای یک گونه عمدتاً با استفاده از دو روش انجام می شود:
روش حضور-پیشینه : این روش مکان هایی را که یک گونه خاص مشاهده شده است (حضور) با سایر مکان هایی که گونه مشاهده نشده است (پس زمینه) مقایسه می کند. در اینجا، دادههای پسزمینه لزوماً به معنای مناطقی نیست که گونه وجود ندارد، بلکه بهمنظور منعکسکننده شرایط کلی محیطی منطقه مورد مطالعه تنظیم شده است. برای تشخیص محیط های مناسبی که گونه ها می توانند وجود داشته باشند از محیط های کمتر مناسب استفاده می شود.
روش حضور-غیاب : این روش مکان هایی را که گونه در آن مشاهده شده است (حضور) با مکان هایی که به طور قطعی مشاهده نشده است (غیاب) مقایسه می کند. در اینجا، دادههای غیبت نشاندهنده مکانهای خاصی است که گونهها وجود ندارند. شرایط کلی محیطی منطقه مورد مطالعه را منعکس نمی کند، بلکه به مکان هایی اشاره می کند که تخمین زده می شود گونه وجود نداشته باشد.
در عمل، جمعآوری دادههای غیاب واقعی اغلب دشوار است، بنابراین دادههای شبه غیبت که بهطور مصنوعی تولید میشوند اغلب استفاده میشوند. با این حال، اذعان به محدودیتها و خطاهای احتمالی این روش بسیار مهم است، زیرا نقاط شبه غیبت ایجاد شده مصنوعی ممکن است مناطق غیبت واقعی را به دقت منعکس نکنند.
انتخاب بین این دو روش به در دسترس بودن داده ها، اهداف تحقیق، دقت و قابلیت اطمینان مدل و همچنین زمان و منابع بستگی دارد. در اینجا، از دادههای وقوع جمعآوریشده از GBIF و دادههای شبه غیبت تولید شده مصنوعی برای مدلسازی با استفاده از روش «حضور-غیبت» استفاده میکنیم.
تولید داده های شبه غیبت از طریق "رویکرد پروفایل زیست محیطی" انجام می شود و مراحل مشخص به شرح زیر است:
طبقهبندی محیطی با استفاده از خوشهبندی k-means: الگوریتم خوشهبندی k-means، بر اساس فاصله اقلیدسی، برای تقسیم پیکسلهای داخل منطقه مورد مطالعه به دو خوشه استفاده خواهد شد. یک خوشه مناطقی با ویژگی های محیطی مشابه را به 100 مکان حضور تصادفی انتخاب شده نشان می دهد، در حالی که خوشه دیگر مناطقی با ویژگی های متفاوت را نشان می دهد.
تولید دادههای شبه غیبت در خوشههای غیرمشابه: در دومین خوشه شناساییشده در مرحله اول (که ویژگیهای محیطی متفاوتی با دادههای حضور دارد)، نقاط شبه غیبت بهطور تصادفی ایجاد میشوند. این نقاط شبه غیبت نشان دهنده مکان هایی است که انتظار نمی رود گونه وجود داشته باشد.
# Randomly select 100 locations for occurrence
pvals = predictors.sampleRegions(
collection=data.randomColumn().sort('random').limit(100),
properties=[],
scale=grain_size
)
# Perform k-means clustering
clusterer = ee.Clusterer.wekaKMeans(
nClusters=2,
distanceFunction="Euclidean"
).train(pvals)
cl_result = predictors.cluster(clusterer)
# Get cluster ID for locations similar to occurrence
cl_id = cl_result.sampleRegions(
collection=data.randomColumn().sort('random').limit(200),
properties=[],
scale=grain_size
)
# Define non-occurrence areas in dissimilar clusters
cl_id = ee.FeatureCollection(cl_id).reduceColumns(ee.Reducer.mode(),['cluster'])
cl_id = ee.Number(cl_id.get('mode')).subtract(1).abs()
cl_mask = cl_result.select(['cluster']).eq(cl_id)
# Presence location mask
presence_mask = data.reduceToImage(properties=['random'],
reducer=ee.Reducer.first()
).reproject('EPSG:4326', None,
grain_size).mask().neq(1).selfMask()
# Masking presence locations in non-occurrence areas and clipping to AOI
area_for_pa = presence_mask.updateMask(cl_mask).clip(aoi)
# Area for Pseudo-absence
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(area_for_pa, {'palette': 'black'}, 'AreaForPA')
Map.centerObject(aoi, 6)
Map
برازش و پیش بینی مدل
اکنون داده ها را به داده های آموزشی و داده های آزمایشی تقسیم می کنیم. از داده های آموزشی برای یافتن پارامترهای بهینه با آموزش مدل استفاده می شود، در حالی که از داده های آزمون برای ارزیابی مدل آموزش داده شده از قبل استفاده می شود. مفهوم مهمی که در این زمینه باید در نظر گرفته شود، خودهمبستگی فضایی است.
خودهمبستگی فضایی یک عنصر اساسی در SDM است که با قانون توبلر مرتبط است. این مفهوم را مجسم می کند که "همه چیز به هر چیز دیگری مربوط است، اما چیزهای نزدیک بیشتر از چیزهای دور مرتبط هستند". خودهمبستگی فضایی نشان دهنده رابطه معنادار بین مکان گونه ها و متغیرهای محیطی است. با این حال، اگر خود همبستگی فضایی بین دادههای آموزش و آزمون وجود داشته باشد، استقلال بین دو مجموعه داده میتواند به خطر بیفتد. این به طور قابل توجهی بر ارزیابی توانایی تعمیم مدل تأثیر می گذارد.
یک روش برای پرداختن به این مشکل، تکنیک اعتبارسنجی متقاطع بلوک فضایی است که شامل تقسیم داده ها به مجموعه داده های آموزشی و آزمایشی است. این تکنیک شامل تقسیم داده ها به بلوک های متعدد، استفاده از هر بلوک به طور مستقل به عنوان مجموعه داده های آموزشی و آزمایشی برای کاهش تأثیر همبستگی خودکار مکانی است. این امر استقلال بین مجموعههای داده را افزایش میدهد و امکان ارزیابی دقیقتری از توانایی تعمیم مدل را فراهم میکند.
روال خاص به شرح زیر است:
- ایجاد بلوک های فضایی: کل مجموعه داده را به بلوک های فضایی با اندازه مساوی (مثلاً 50x50 کیلومتر) تقسیم کنید.
- تخصیص مجموعه های آموزشی و آزمایشی: هر بلوک فضایی به طور تصادفی به مجموعه آموزشی (70٪) یا مجموعه آزمایشی (30٪) اختصاص داده می شود. این مانع از تطبیق بیش از حد مدل به داده های مناطق خاص می شود و هدف آن دستیابی به نتایج کلی تر است.
- اعتبارسنجی متقاطع تکراری: کل فرآیند n بار (مثلاً 10 بار) تکرار می شود. در هر تکرار، بلوکها بهطور تصادفی به مجموعههای آموزشی و آزمایشی تقسیم میشوند که هدف آن بهبود پایداری و قابلیت اطمینان مدل است.
- تولید دادههای شبه عدم حضور: در هر تکرار، دادههای شبه عدم حضور بهطور تصادفی برای ارزیابی عملکرد مدل تولید میشوند.
Scale = 50000
grid = watermask.reduceRegions(
collection=aoi.coveringGrid(scale=Scale, proj='EPSG:4326'),
reducer=ee.Reducer.mean()).filter(ee.Filter.neq('mean', None))
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(grid, {}, "Grid for spatial block cross validation")
Map.addLayer(outline, {'palette': 'FF0000'}, "Study Area")
Map.centerObject(aoi, 6)
Map
اکنون می توانیم مدل را متناسب کنیم. برازش یک مدل شامل درک الگوهای موجود در داده ها و تنظیم پارامترهای مدل (وزن ها و سوگیری ها) بر این اساس است. این فرآیند مدل را قادر می سازد تا پیش بینی های دقیق تری را هنگام ارائه داده های جدید انجام دهد. برای این منظور، تابعی به نام SDM() برای برازش مدل تعریف کرده ایم.
ما از الگوریتم Random Forest استفاده خواهیم کرد.
def sdm(x):
seed = ee.Number(x)
# Random block division for training and validation
rand_blk = ee.FeatureCollection(grid).randomColumn(seed=seed).sort("random")
training_grid = rand_blk.filter(ee.Filter.lt("random", split)) # Grid for training
testing_grid = rand_blk.filter(ee.Filter.gte("random", split)) # Grid for testing
# Presence points
presence_points = ee.FeatureCollection(data)
presence_points = presence_points.map(lambda feature: feature.set("PresAbs", 1))
tr_presence_points = presence_points.filter(
ee.Filter.bounds(training_grid)
) # Presence points for training
te_presence_points = presence_points.filter(
ee.Filter.bounds(testing_grid)
) # Presence points for testing
# Pseudo-absence points for training
tr_pseudo_abs_points = area_for_pa.sample(
region=training_grid,
scale=grain_size,
numPixels=tr_presence_points.size().add(300),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for training
tr_pseudo_abs_points = (
tr_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(tr_presence_points.size()))
)
tr_pseudo_abs_points = tr_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
te_pseudo_abs_points = area_for_pa.sample(
region=testing_grid,
scale=grain_size,
numPixels=te_presence_points.size().add(100),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for testing
te_pseudo_abs_points = (
te_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(te_presence_points.size()))
)
te_pseudo_abs_points = te_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
# Merge training and pseudo-absence points
training_partition = tr_presence_points.merge(tr_pseudo_abs_points)
testing_partition = te_presence_points.merge(te_pseudo_abs_points)
# Extract predictor variable values at training points
train_pvals = predictors.sampleRegions(
collection=training_partition,
properties=["PresAbs"],
scale=grain_size,
geometries=True,
)
# Random Forest classifier
classifier = ee.Classifier.smileRandomForest(
numberOfTrees=500,
variablesPerSplit=None,
minLeafPopulation=10,
bagFraction=0.5,
maxNodes=None,
seed=seed,
)
# Presence probability: Habitat suitability map
classifier_pr = classifier.setOutputMode("PROBABILITY").train(
train_pvals, "PresAbs", bands
)
classified_img_pr = predictors.select(bands).classify(classifier_pr)
# Binary presence/absence map: Potential distribution map
classifier_bin = classifier.setOutputMode("CLASSIFICATION").train(
train_pvals, "PresAbs", bands
)
classified_img_bin = predictors.select(bands).classify(classifier_bin)
return [
classified_img_pr,
classified_img_bin,
training_partition,
testing_partition,
], classifier_pr
بلوک های فضایی به ترتیب به 70 درصد برای آموزش مدل و 30 درصد برای آزمایش مدل تقسیم می شوند. داده های شبه غیبت به طور تصادفی در هر مجموعه آموزشی و آزمایشی در هر تکرار ایجاد می شود. در نتیجه، هر اجرا مجموعههای متفاوتی از دادههای حضور و شبه غیبت را برای آموزش و آزمایش مدل به دست میدهد.
split = 0.7
numiter = 10
# Random Seed
runif = lambda length: [random.randint(1, 1000) for _ in range(length)]
items = runif(numiter)
# Fixed seed
# items = [287, 288, 553, 226, 151, 255, 902, 267, 419, 538]
results_list = [] # Initialize SDM results list
importances_list = [] # Initialize variable importance list
for item in items:
result, trained = sdm(item)
# Accumulate SDM results into the list
results_list.extend(result)
# Accumulate variable importance into the list
importance = ee.Dictionary(trained.explain()).get('importance')
importances_list.extend(importance.getInfo().items())
# Flatten the SDM results list
results = ee.List(results_list).flatten()
اکنون میتوانیم نقشه تناسب زیستگاه و نقشه پراکنش بالقوه Fairy pitta را تجسم کنیم. در این مورد، نقشه تناسب زیستگاه با استفاده از تابع mean() برای محاسبه میانگین برای هر مکان پیکسل در تمام تصاویر ایجاد میشود و نقشه توزیع پتانسیل با استفاده از تابع mode() برای تعیین بیشترین مقدار ایجاد میشود. در هر مکان پیکسل در تمام تصاویر.

# Habitat suitability map
images = ee.List.sequence(
0, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x))
model_average = ee.ImageCollection.fromImages(images).mean()
Map = geemap.Map(layout={'height':'400px', 'width':'800px'}, basemap='Esri.WorldImagery')
vis_params = {
'min': 0,
'max': 1,
'palette': cm.palettes.viridis_r}
Map.addLayer(model_average, vis_params, 'Habitat suitability')
Map.add_colorbar(vis_params, label="Habitat suitability",
orientation="horizontal",
layer_name="Habitat suitability")
Map.addLayer(data, {'color':'red'}, 'Presence')
Map.centerObject(aoi, 6)
Map
# Potential distribution map
images2 = ee.List.sequence(1, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x)
)
distribution_map = ee.ImageCollection.fromImages(images2).mode()
Map = geemap.Map(
layout={"height": "400px", "width": "800px"}, basemap="Esri.WorldImagery"
)
vis_params = {"min": 0, "max": 1, "palette": ["white", "green"]}
Map.addLayer(distribution_map, vis_params, "Potential distribution")
Map.addLayer(data, {"color": "red"}, "Presence")
Map.add_colorbar(
vis_params,
label="Potential distribution",
discrete=True,
orientation="horizontal",
layer_name="Potential distribution",
)
Map.centerObject(data.geometry(), 6)
Map
ارزیابی اهمیت و دقت متغیر
Random Forest ( ee.Classifier.smileRandomForest ) یکی از روشهای یادگیری گروهی است که با ساخت درختهای تصمیمگیری متعدد برای پیشبینی عمل میکند. هر درخت تصمیم بهطور مستقل از زیر مجموعههای مختلف دادهها یاد میگیرد و نتایج آنها برای پیشبینی دقیقتر و پایدارتر تجمیع میشوند.
اهمیت متغیر معیاری است که تأثیر هر متغیر را بر پیشبینیهای مدل جنگل تصادفی ارزیابی میکند. برای محاسبه و چاپ میانگین اهمیت متغیر، از importances_list از قبل تعریف شده استفاده خواهیم کرد.
def plot_variable_importance(importances_list):
# Extract each variable importance value into a list
variables = [item[0] for item in importances_list]
importances = [item[1] for item in importances_list]
# Calculate the average importance for each variable
average_importances = {}
for variable in set(variables):
indices = [i for i, var in enumerate(variables) if var == variable]
average_importance = np.mean([importances[i] for i in indices])
average_importances[variable] = average_importance
# Sort the importances in descending order of importance
sorted_importances = sorted(average_importances.items(),
key=lambda x: x[1], reverse=False)
variables = [item[0] for item in sorted_importances]
avg_importances = [item[1] for item in sorted_importances]
# Adjust the graph size
plt.figure(figsize=(8, 4))
# Plot the average importance as a horizontal bar chart
plt.barh(variables, avg_importances)
plt.xlabel('Importance')
plt.ylabel('Variables')
plt.title('Average Variable Importance')
# Display values above the bars
for i, v in enumerate(avg_importances):
plt.text(v + 0.02, i, f"{v:.2f}", va='center')
# Adjust the x-axis range
plt.xlim(0, max(avg_importances) + 5) # Adjust to the desired range
plt.tight_layout()
plt.savefig('variable_importance.png')
plt.show()
plot_variable_importance(importances_list)
با استفاده از مجموعه داده های تست، ما AUC-ROC و AUC-PR را برای هر اجرا محاسبه می کنیم. سپس، میانگین AUC-ROC و AUC-PR را در n تکرار محاسبه می کنیم.
AUC-ROC ناحیه زیر منحنی نمودار «حساسیت (یادآوری) در مقابل 1-ویژگی» را نشان میدهد، که رابطه بین حساسیت و ویژگی را با تغییر آستانه نشان میدهد. ویژگی بر اساس همه موارد غیرقابل مشاهده است. بنابراین، AUC-ROC تمام ربع های ماتریس سردرگمی را در بر می گیرد.
AUC-PR نشاندهنده ناحیه زیر منحنی نمودار «دقت در مقابل یادآوری (حساسیت)» است که رابطه بین دقت و یادآوری را با تغییر آستانه نشان میدهد. دقت بر اساس تمام رخدادهای پیش بینی شده است. از این رو، AUC-PR شامل منفی های واقعی (TN) نمی شود.
def print_pres_abs_sizes(TestingDatasets, numiter):
# Check and print the sizes of presence and pseudo-absence coordinates
def get_pres_abs_size(x):
fc = ee.FeatureCollection(TestingDatasets.get(x))
presence_size = fc.filter(ee.Filter.eq("PresAbs", 1)).size()
pseudo_absence_size = fc.filter(ee.Filter.eq("PresAbs", 0)).size()
return ee.List([presence_size, pseudo_absence_size])
sizes_info = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(get_pres_abs_size)
.getInfo()
)
for i, sizes in enumerate(sizes_info):
presence_size = sizes[0]
pseudo_absence_size = sizes[1]
print(
f"Iteration {i + 1}: Presence Size = {presence_size}, Pseudo-absence Size = {pseudo_absence_size}"
)
# Extracting the Testing Datasets
testing_datasets = ee.List.sequence(
3, ee.Number(numiter).multiply(4).subtract(1), 4
).map(lambda x: results.get(x))
print_pres_abs_sizes(testing_datasets, numiter)
def get_acc(hsm, t_data, grain_size):
pr_prob_vals = hsm.sampleRegions(
collection=t_data, properties=["PresAbs"], scale=grain_size
)
seq = ee.List.sequence(start=0, end=1, count=25) # Divide 0 to 1 into 25 intervals
def calculate_metrics(cutoff):
# Each element of the seq list is passed as cutoff(threshold value)
# Observed present = TP + FN
pres = pr_prob_vals.filterMetadata("PresAbs", "equals", 1)
# TP (True Positive)
tp = ee.Number(
pres.filterMetadata("classification", "greater_than", cutoff).size()
)
# TPR (True Positive Rate) = Recall = Sensitivity = TP / (TP + FN) = TP / Observed present
tpr = tp.divide(pres.size())
# Observed absent = FP + TN
abs = pr_prob_vals.filterMetadata("PresAbs", "equals", 0)
# FN (False Negative)
fn = ee.Number(
pres.filterMetadata("classification", "less_than", cutoff).size()
)
# TNR (True Negative Rate) = Specificity = TN / (FP + TN) = TN / Observed absent
tn = ee.Number(abs.filterMetadata("classification", "less_than", cutoff).size())
tnr = tn.divide(abs.size())
# FP (False Positive)
fp = ee.Number(
abs.filterMetadata("classification", "greater_than", cutoff).size()
)
# FPR (False Positive Rate) = FP / (FP + TN) = FP / Observed absent
fpr = fp.divide(abs.size())
# Precision = TP / (TP + FP) = TP / Predicted present
precision = tp.divide(tp.add(fp))
# SUMSS = SUM of Sensitivity and Specificity
sumss = tpr.add(tnr)
return ee.Feature(
None,
{
"cutoff": cutoff,
"TP": tp,
"TN": tn,
"FP": fp,
"FN": fn,
"TPR": tpr,
"TNR": tnr,
"FPR": fpr,
"Precision": precision,
"SUMSS": sumss,
},
)
return ee.FeatureCollection(seq.map(calculate_metrics))
def calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter):
# Calculate AUC-ROC and AUC-PR
def calculate_auc_metrics(x):
hsm = ee.Image(images.get(x))
t_data = ee.FeatureCollection(testing_datasets.get(x))
acc = get_acc(hsm, t_data, grain_size)
# Calculate AUC-ROC
x = ee.Array(acc.aggregate_array("FPR"))
y = ee.Array(acc.aggregate_array("TPR"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_roc = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
# Calculate AUC-PR
x = ee.Array(acc.aggregate_array("TPR"))
y = ee.Array(acc.aggregate_array("Precision"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_pr = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
return (auc_roc, auc_pr)
auc_metrics = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(calculate_auc_metrics)
.getInfo()
)
# Print AUC-ROC and AUC-PR for each iteration
df = pd.DataFrame(auc_metrics, columns=["AUC-ROC", "AUC-PR"])
df.index = [f"Iteration {i + 1}" for i in range(len(df))]
df.to_csv("auc_metrics.csv", index_label="Iteration")
print(df)
# Calculate mean and standard deviation of AUC-ROC and AUC-PR
mean_auc_roc, std_auc_roc = df["AUC-ROC"].mean(), df["AUC-ROC"].std()
mean_auc_pr, std_auc_pr = df["AUC-PR"].mean(), df["AUC-PR"].std()
print(f"Mean AUC-ROC = {mean_auc_roc:.4f} ± {std_auc_roc:.4f}")
print(f"Mean AUC-PR = {mean_auc_pr:.4f} ± {std_auc_pr:.4f}")
%%time
# Calculate AUC-ROC and AUC-PR
calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter)
این آموزش یک مثال کاربردی از استفاده از موتور Google Earth (GEE) برای مدلسازی توزیع گونهها (SDM) ارائه کرده است. یک نکته مهم، تطبیق پذیری و انعطاف پذیری GEE در زمینه SDM است. استفاده از قابلیتهای پردازش دادههای مکانی قدرتمند Earth Engine، فرصتهای بیپایانی را برای محققان و حافظان محیط زیست برای درک و حفظ تنوع زیستی در سیاره ما باز میکند. با استفاده از دانش و مهارتهای بهدستآمده از این آموزش، افراد میتوانند به کشف و مشارکت در این زمینه جذاب تحقیقات زیستمحیطی کمک کنند.

 در Google Colab اجرا شود
در Google Colab اجرا شود مشاهده منبع در GitHub
مشاهده منبع در GitHub