इस ट्यूटोरियल में, Google Earth Engine का इस्तेमाल करके, प्रजातियों के डिस्ट्रिब्यूशन मॉडल बनाने के तरीके के बारे में बताया जाएगा. इसमें, प्रजातियों के डिस्ट्रिब्यूशन मॉडलिंग के बारे में खास जानकारी दी जाएगी. इसके बाद, लुप्तप्राय पक्षी की प्रजाति के हैबिटैट का अनुमान लगाने और उसका विश्लेषण करने की प्रोसेस के बारे में बताया जाएगा. इस पक्षी को परी पट्टा (वैज्ञानिक नाम: पिट्टा निम्फ़ा) के नाम से जाना जाता है.
मुझे सबसे पहले चलाओ
एपीआई को शुरू करने के लिए, इस सेल को चलाएं. आउटपुट में, अपने खाते का इस्तेमाल करके Earth Engine को इस नोटबुक का ऐक्सेस देने के तरीके के बारे में निर्देश शामिल होंगे.
import ee
# Trigger the authentication flow.
ee.Authenticate()
# Initialize the library.
ee.Initialize(project='my-project')
प्रजाति के डिस्ट्रिब्यूशन मॉडलिंग के बारे में खास जानकारी
आइए, जानते हैं कि प्रजातियों के डिस्ट्रिब्यूशन मॉडल क्या होते हैं, उन्हें प्रोसेस करने के लिए Google Earth Engine का इस्तेमाल करने के क्या फ़ायदे हैं, मॉडल के लिए ज़रूरी डेटा क्या है, और वर्कफ़्लो कैसे स्ट्रक्चर किया जाता है.
प्रजाति के डिस्ट्रिब्यूशन का मॉडल क्या है?
किसी प्रजाति के भौगोलिक वितरण का अनुमान लगाने के लिए, प्रजाति वितरण मॉडलिंग (नीचे एसडीएम) का इस्तेमाल सबसे ज़्यादा किया जाता है. इसमें किसी खास प्रजाति के लिए सही पर्यावरणीय स्थितियों का पता लगाया जाता है. इसके बाद, यह पता लगाया जाता है कि ये सही स्थितियां भौगोलिक रूप से कहां-कहां मौजूद हैं.
एसडीएम, हाल के सालों में संरक्षण की योजना बनाने का एक अहम हिस्सा बन गया है. इसके लिए, मॉडलिंग की कई तकनीकों को विकसित किया गया है. Google Earth Engine (यहां GEE) में एसडीएम लागू करने से, बड़े पैमाने पर पर्यावरण से जुड़े डेटा को आसानी से ऐक्सेस किया जा सकता है. साथ ही, इसमें कंप्यूटिंग की बेहतर सुविधाएं और मशीन लर्निंग एल्गोरिदम के लिए सहायता मिलती है. इससे तेज़ी से मॉडलिंग की जा सकती है.
एसडीएम के लिए ज़रूरी डेटा
एसडीएम, आम तौर पर किसी प्रजाति के ज्ञात होने के रिकॉर्ड और पर्यावरणीय वैरिएबल के बीच के संबंध का इस्तेमाल करता है. इससे यह पता चलता है कि किसी प्रजाति की आबादी किन स्थितियों में बनी रह सकती है. दूसरे शब्दों में कहें, तो मॉडल के इनपुट डेटा के लिए दो तरह के डेटा की ज़रूरत होती है:
- जानवरों की जानी-पहचानी प्रजातियों के मिलने की जानकारी देने वाले रिकॉर्ड
- अलग-अलग एनवायरमेंट वैरिएबल
इन डेटा को एल्गोरिदम में डाला जाता है, ताकि प्रजातियों की मौजूदगी से जुड़ी पर्यावरणीय स्थितियों की पहचान की जा सके.
GEE का इस्तेमाल करके SDM का वर्कफ़्लो
GEE का इस्तेमाल करके एसडीएम के लिए वर्कफ़्लो इस तरह है:
- प्रजातियों के दिखने से जुड़े डेटा को इकट्ठा करना और उसे पहले से प्रोसेस करना
- दिलचस्पी के आधार पर टारगेट की जाने वाली जगह की परिभाषा
- GEE एनवायरमेंट वैरिएबल जोड़े गए
- छद्म-अनुपस्थिति डेटा जनरेट करना
- मॉडल फ़िटिंग और अनुमान
- वैरिएबल की अहमियत और सटीक आकलन
GEE का इस्तेमाल करके, हैबिटैट का अनुमान लगाना और उसका विश्लेषण करना
GEE पर आधारित एसडीएम के इस्तेमाल को दिखाने के लिए, परी पट्टा (पिट्टा निम्फ़ा) को केस स्टडी के तौर पर इस्तेमाल किया जाएगा. इस उदाहरण के लिए, इस खास प्रजाति को चुना गया है. हालांकि, शोधकर्ता इस तरीके को किसी भी टारगेट प्रजाति पर लागू कर सकते हैं. इसके लिए, उन्हें दिए गए सोर्स कोड में थोड़ा बदलाव करना होगा.
फ़ेयरी पिट्टा, दक्षिण कोरिया में गर्मियों के दौरान बहुत कम दिखती है. यह कोरियाई प्रायद्वीप में जलवायु के गर्म होने की वजह से, अब ज़्यादा इलाकों में दिखती है. इसे दुर्लभ प्रजाति, क्लास II के विलुप्त होने के खतरे में पड़े वन्यजीव, नैचुरल मॉन्यूमेंट नंबर 204 के तौर पर क्लासिफ़ाई किया गया है. साथ ही, नैशनल रेड लिस्ट में इसे क्षेत्रीय तौर पर विलुप्त (आरई) और आईयूसीएन की कैटगरी के हिसाब से खतरे में (वीयू) के तौर पर आंका गया है.
फ़ेयरी पिट्टा के संरक्षण के लिए योजना बनाने में एसडीएम का इस्तेमाल करना काफ़ी फ़ायदेमंद साबित हो सकता है. अब, GEE की मदद से हैबिटैट का अनुमान लगाने और उसका विश्लेषण करने की प्रोसेस शुरू करते हैं.
सबसे पहले, Python लाइब्रेरी इंपोर्ट की जाती हैं. import स्टेटमेंट, किसी मॉड्यूल का पूरा कॉन्टेंट इंपोर्ट करता है. वहीं, from import स्टेटमेंट की मदद से, किसी मॉड्यूल से कुछ ऑब्जेक्ट इंपोर्ट किए जा सकते हैं.
# Import libraries
import geemap
import geemap.colormaps as cm
import pandas as pd, geopandas as gpd
import numpy as np, matplotlib.pyplot as plt
import os, requests, math, random
from ipyleaflet import TileLayer
from statsmodels.stats.outliers_influence import variance_inflation_factor
प्रजातियों के दिखने से जुड़े डेटा को इकट्ठा करना और उसे पहले से प्रोसेस करना
अब, फ़ेयरी पिट्टा के दिखने का डेटा इकट्ठा करते हैं. अगर फ़िलहाल आपके पास, दिलचस्पी वाली प्रजातियों के दिखने से जुड़ा डेटा ऐक्सेस करने की सुविधा नहीं है, तो GBIF API के ज़रिए किसी खास प्रजाति के बारे में ऑब्ज़र्वेशनल डेटा पाया जा सकता है. GBIF API एक इंटरफ़ेस है. इसकी मदद से, GBIF की ओर से उपलब्ध कराए गए प्रजातियों के डिस्ट्रिब्यूशन डेटा को ऐक्सेस किया जा सकता है. इससे उपयोगकर्ता, डेटा को खोज सकते हैं, फ़िल्टर कर सकते हैं, और डाउनलोड कर सकते हैं. साथ ही, प्रजातियों से जुड़ी अलग-अलग तरह की जानकारी पा सकते हैं.
नीचे दिए गए कोड में, species_name वैरिएबल को प्रजाति का वैज्ञानिक नाम असाइन किया गया है. जैसे, परी पट्टा के लिए Pitta nympha), और country_code वैरिएबल को देश का कोड असाइन किया जाता है. जैसे, KR, दक्षिण कोरिया के लिए). base_url वैरिएबल, GBIF API का पता सेव करता है. params एक डिक्शनरी है. इसमें एपीआई अनुरोध में इस्तेमाल किए जाने वाले पैरामीटर शामिल होते हैं:
scientificName: इससे खोजी जाने वाली प्रजाति का वैज्ञानिक नाम सेट किया जाता है.country: इससे खोज के नतीजे किसी देश के हिसाब से सीमित हो जाते हैं.hasCoordinate: इससे यह पक्का किया जाता है कि सिर्फ़ कोऑर्डिनेट (सही) वाले डेटा को खोजा जाए.basisOfRecord: यह सिर्फ़ इंसानों के किए गए ऑब्ज़र्वेशन (HUMAN_OBSERVATION) के रिकॉर्ड चुनता है.limit: इससे, ज़्यादा से ज़्यादा 10,000 नतीजे दिखते हैं.
def get_gbif_species_data(species_name, country_code):
"""
Retrieves observational data for a specific species using the GBIF API and returns it as a pandas DataFrame.
Parameters:
species_name (str): The scientific name of the species to query.
country_code (str): The country code of the where the observation data will be queried.
Returns:
pd.DataFrame: A pandas DataFrame containing the observational data.
"""
base_url = "https://api.gbif.org/v1/occurrence/search"
params = {
"scientificName": species_name,
"country": country_code,
"hasCoordinate": "true",
"basisOfRecord": "HUMAN_OBSERVATION",
"limit": 10000,
}
try:
response = requests.get(base_url, params=params)
response.raise_for_status() # Raises an exception for a response error.
data = response.json()
occurrences = data.get("results", [])
if occurrences: # If data is present
df = pd.json_normalize(occurrences)
return df
else:
print("No data found for the given species and country code.")
return pd.DataFrame() # Returns an empty DataFrame
except requests.RequestException as e:
print(f"Request failed: {e}")
return pd.DataFrame() # Returns an empty DataFrame in case of an exception
पहले सेट किए गए पैरामीटर का इस्तेमाल करके, हम GBIF API से फ़ेयरी पिट्टा (Pitta nympha) के ऑब्ज़र्वेशनल रिकॉर्ड के लिए क्वेरी करते हैं. इसके बाद, नतीजों को DataFrame में लोड करते हैं, ताकि पहली लाइन की जांच की जा सके. डेटाफ़्रेम, टेबल के फ़ॉर्मैट में मौजूद डेटा को मैनेज करने के लिए इस्तेमाल किया जाने वाला डेटा स्ट्रक्चर है. इसमें पंक्तियां और कॉलम होते हैं. अगर ज़रूरी हो, तो DataFrame को CSV फ़ाइल के तौर पर सेव किया जा सकता है और फिर से पढ़ा जा सकता है.
# Retrieve Fairy Pitta data
df = get_gbif_species_data("Pitta nympha", "KR")
"""
# Save DataFrame to CSV and read back in.
df.to_csv("pitta_nympha_data.csv", index=False)
df = pd.read_csv("pitta_nympha_data.csv")
"""
df.head(1) # Display the first row of the DataFrame
इसके बाद, हम DataFrame को GeoDataFrame में बदलते हैं. इसमें भौगोलिक जानकारी (geometry) के लिए एक कॉलम शामिल होता है. साथ ही, हम पहली पंक्ति की जांच करते हैं. GeoDataFrame को GeoPackage फ़ाइल (*.gpkg) के तौर पर सेव किया जा सकता है. साथ ही, इसे वापस पढ़ा जा सकता है.
# Convert DataFrame to GeoDataFrame
gdf = gpd.GeoDataFrame(
df,
geometry=gpd.points_from_xy(df.decimalLongitude,
df.decimalLatitude),
crs="EPSG:4326"
)[["species", "year", "month", "geometry"]]
"""
# Convert GeoDataFrame to GeoPackage (requires pycrs module)
%pip install -U -q pycrs
gdf.to_file("pitta_nympha_data.gpkg", driver="GPKG")
gdf = gpd.read_file("pitta_nympha_data.gpkg")
"""
gdf.head(1) # Display the first row of the GeoDataFrame
इस बार, हमने GeoDataFrame से साल और महीने के हिसाब से डेटा के डिस्ट्रिब्यूशन को विज़ुअलाइज़ करने के लिए एक फ़ंक्शन बनाया है. साथ ही, इसे ग्राफ़ के तौर पर दिखाया है. इसके बाद, इसे इमेज फ़ाइल के तौर पर सेव किया जा सकता है. हीटमैप का इस्तेमाल करके, हमें यह पता चलता है कि किसी प्रजाति के दिखने की फ़्रीक्वेंसी साल और महीने के हिसाब से कितनी है. इससे हमें डेटा में समय के साथ हुए बदलावों और पैटर्न को आसानी से समझने में मदद मिलती है. इससे, प्रजातियों के होने से जुड़े डेटा में समय के साथ होने वाले बदलावों और मौसमी बदलावों की पहचान की जा सकती है. साथ ही, डेटा में मौजूद आउटलायर या क्वालिटी से जुड़ी समस्याओं का तुरंत पता लगाया जा सकता है.
# Yearly and monthly data distribution heatmap
def plot_heatmap(gdf, h_size=8):
statistics = gdf.groupby(["month", "year"]).size().unstack(fill_value=0)
# Heatmap
plt.figure(figsize=(h_size, h_size - 6))
heatmap = plt.imshow(
statistics.values, cmap="YlOrBr", origin="upper", aspect="auto"
)
# Display values above each pixel
for i in range(len(statistics.index)):
for j in range(len(statistics.columns)):
plt.text(
j, i, statistics.values[i, j], ha="center", va="center", color="black"
)
plt.colorbar(heatmap, label="Count")
plt.title("Monthly Species Count by Year")
plt.xlabel("Year")
plt.ylabel("Month")
plt.xticks(range(len(statistics.columns)), statistics.columns)
plt.yticks(range(len(statistics.index)), statistics.index)
plt.tight_layout()
plt.savefig("heatmap_plot.png")
plt.show()
plot_heatmap(gdf)
साल 1995 का डेटा बहुत कम है. इसमें अन्य सालों की तुलना में काफ़ी अंतर है. साथ ही, अगस्त और सितंबर के महीनों में भी सीमित सैंपल हैं. इनमें अन्य अवधियों की तुलना में, मौसम के हिसाब से अलग-अलग विशेषताएं दिखती हैं. इस डेटा को शामिल न करने से, मॉडल की स्थिरता और अनुमान लगाने की क्षमता को बेहतर बनाने में मदद मिल सकती है.
हालांकि, यह ध्यान रखना ज़रूरी है कि डेटा को शामिल न करने से, मॉडल की सामान्यीकरण क्षमता बढ़ सकती है. लेकिन, इससे रिसर्च के लक्ष्यों से जुड़ी अहम जानकारी भी मिट सकती है. इसलिए, ऐसे फ़ैसले सोच-समझकर लेने चाहिए.
# Filtering data by year and month
filtered_gdf = gdf[
(~gdf['year'].eq(1995)) &
(~gdf['month'].between(8, 9))
]
अब फ़िल्टर किए गए GeoDataFrame को Google Earth Engine ऑब्जेक्ट में बदल दिया जाता है.
# Convert GeoDataFrame to Earth Engine object
data_raw = geemap.geopandas_to_ee(filtered_gdf)
इसके बाद, हम एसडीएम के नतीजों के रास्टर पिक्सल साइज़ को 1 कि॰मी॰ के रिज़ॉल्यूशन के तौर पर तय करेंगे.
# Spatial resolution setting (meters)
grain_size = 1000
जब एक ही किलोमीटर के रिज़ॉल्यूशन वाले रास्टर पिक्सल में एक से ज़्यादा बार होने वाली घटनाएं मौजूद होती हैं, तो इस बात की संभावना ज़्यादा होती है कि वे एक ही भौगोलिक जगह पर एक जैसी पर्यावरणीय स्थितियों को शेयर करती हैं. इस तरह के डेटा का इस्तेमाल सीधे तौर पर विश्लेषण में करने से, नतीजों में पूर्वाग्रह आ सकता है.
दूसरे शब्दों में कहें, तो हमें भौगोलिक सैंपलिंग के पूर्वाग्रह के संभावित असर को कम करना होगा. इसके लिए, हम हर एक कि॰मी॰ के पिक्सल में सिर्फ़ एक जगह की जानकारी सेव करेंगे और बाकी सभी को हटा देंगे. इससे मॉडल, पर्यावरण की स्थितियों को ज़्यादा सटीक तरीके से दिखा पाएगा.
def remove_duplicates(data, grain_size):
# Select one occurrence record per pixel at the chosen spatial resolution
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
rand_point_vals = random_raster.sampleRegions(
collection=ee.FeatureCollection(data), geometries=True
)
return rand_point_vals.distinct("random")
data = remove_duplicates(data_raw, grain_size)
# Before selection and after selection
print("Original data size:", data_raw.size().getInfo())
print("Final data size:", data.size().getInfo())
नीचे दी गई इमेज में, प्रीप्रोसेसिंग से पहले (नीले रंग में) और प्रीप्रोसेसिंग के बाद (लाल रंग में) भौगोलिक सैंपलिंग के पूर्वाग्रह की तुलना करने वाला विज़ुअलाइज़ेशन दिखाया गया है. तुलना करने में आसानी हो, इसके लिए मैप को उस इलाके के हिसाब से सेट किया गया है जहां हालासां नैशनल पार्क में फ़ेयरी पिट्टा के दिखने की जानकारी देने वाले कोऑर्डिनेट ज़्यादा हैं.
# Visualization of geographic sampling bias before (blue) and after (red) preprocessing
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
# Add the random raster layer
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
Map.addLayer(
random_raster,
{"min": 0, "max": 1, "palette": ["black", "white"], "opacity": 0.5},
"Random Raster",
)
# Add the original data layer in blue
Map.addLayer(data_raw, {"color": "blue"}, "Original data")
# Add the final data layer in red
Map.addLayer(data, {"color": "red"}, "Final data")
# Set the center of the map to the coordinates
Map.setCenter(126.712, 33.516, 14)
Map
दिलचस्पी के आधार पर टारगेट की जाने वाली जगह की परिभाषा
दिलचस्पी वाली जगह (नीचे एओआई) को तय करने का मतलब, उस भौगोलिक इलाके से है जिसका विश्लेषण रिसर्चर करना चाहते हैं. इसका मतलब, स्टडी एरिया से मिलता-जुलता है.
इस संदर्भ में, हमने घटना की जगह की लेयर की ज्यामिति का बाउंडिंग बॉक्स हासिल किया. साथ ही, एओआई तय करने के लिए, इसके चारों ओर 50 किलोमीटर का बफ़र बनाया. इसमें ज़्यादा से ज़्यादा 1,000 मीटर का अंतर हो सकता है.
# Define the AOI
aoi = data.geometry().bounds().buffer(distance=50000, maxError=1000)
# Add the AOI to the map
outline = ee.Image().byte().paint(
featureCollection=aoi, color=1, width=3)
Map.remove_layer("Random Raster")
Map.addLayer(outline, {'palette': 'FF0000'}, "AOI")
Map.centerObject(aoi, 6)
Map
GEE एनवायरमेंट वैरिएबल जोड़े गए
अब, विश्लेषण में एनवायरमेंट वैरिएबल जोड़ते हैं. GEE, पर्यावरण से जुड़े वैरिएबल के लिए कई तरह के डेटासेट उपलब्ध कराता है. जैसे, तापमान, बारिश, ऊंचाई, पेड़ों से ढकी जगह, और इलाके की बनावट. इन डेटासेट की मदद से, हम अलग-अलग फ़ैक्टर का बारीकी से विश्लेषण कर पाते हैं. इससे हमें यह पता चलता है कि फ़ेयरी पिट्टा के रहने की जगह की पसंद पर किन फ़ैक्टर का असर पड़ सकता है.
एसडीएम में GEE एनवायरमेंटल वैरिएबल का चुनाव, प्रजातियों की पसंद के मुताबिक उनके हैबिटैट की विशेषताओं को दिखाता है. इसके लिए, फ़ेयरी पिट्टा के रहने की जगहों के बारे में पहले से रिसर्च की जानी चाहिए और साहित्य की समीक्षा की जानी चाहिए. इस ट्यूटोरियल में मुख्य रूप से, GEE का इस्तेमाल करके एसडीएम के वर्कफ़्लो पर फ़ोकस किया गया है. इसलिए, कुछ ज़्यादा जानकारी शामिल नहीं की गई है.
WorldClim V1 Bioclim: इस डेटासेट में, हर महीने के तापमान और बारिश के डेटा से मिले 19 बायोक्लाइमेटिक वैरिएबल शामिल होते हैं. इसमें 1960 से 1991 तक की अवधि शामिल है. इसका रिज़ॉल्यूशन 927.67 मीटर है.
# WorldClim V1 Bioclim
bio = ee.Image("WORLDCLIM/V1/BIO")
NASA SRTM Digital Elevation 30m: इस डेटासेट में, शटल रडार टोपोग्राफ़ी मिशन (एसआरटीएम) का डिजिटल एलीवेशन डेटा शामिल है. इस डेटा को मुख्य रूप से साल 2000 के आस-पास इकट्ठा किया गया था. इसे करीब 30 मीटर (एक आर्क-सेकंड) के रिज़ॉल्यूशन पर उपलब्ध कराया गया है. नीचे दिए गए कोड में, SRTM डेटा से ऊंचाई, ढलान, पहलू, और हिलशेड लेयर की गणना की गई है.
# NASA SRTM Digital Elevation 30m
terrain = ee.Algorithms.Terrain(ee.Image("USGS/SRTMGL1_003"))
Global Forest Cover Change (GFCC) Tree Cover Multi-Year Global 30m: Landsat का Vegetation Continuous Fields (VCF) डेटासेट, वर्टिकल तौर पर प्रोजेक्ट किए गए वनस्पति कवर के अनुपात का अनुमान लगाता है. ऐसा तब होता है, जब वनस्पति की ऊंचाई 5 मीटर से ज़्यादा होती है. यह डेटासेट, 2000, 2005, 2010, और 2015 के आस-पास के चार समयावधियों के लिए उपलब्ध है. इसका रिज़ॉल्यूशन 30 मीटर है. यहां, इन चार समयावधियों की मीडियन वैल्यू का इस्तेमाल किया गया है.
# Global Forest Cover Change (GFCC) Tree Cover Multi-Year Global 30m
tcc = ee.ImageCollection("NASA/MEASURES/GFCC/TC/v3")
median_tcc = (
tcc.filterDate("2000-01-01", "2015-12-31")
.select(["tree_canopy_cover"], ["TCC"])
.median()
)
bio (बायोक्लाइमैटिक वैरिएबल), terrain (टपोग्राफ़ी), और median_tcc (पेड़ों से ढकी जगह) को एक ही मल्टीबैंड इमेज में मिला दिया जाता है. elevation बैंड को terrain से चुना जाता है. साथ ही, उन जगहों के लिए watermask बनाया जाता है जहां elevation, 0 से ज़्यादा है. इससे समुद्र तल से नीचे के इलाकों (जैसे, समुद्र) को मास्क किया जाता है. साथ ही, शोधकर्ता को एओआई के लिए, पर्यावरण से जुड़े अलग-अलग फ़ैक्टर का पूरी तरह से विश्लेषण करने में मदद मिलती है.
# Combine bands into a multi-band image
predictors = bio.addBands(terrain).addBands(median_tcc)
# Create a water mask
watermask = terrain.select('elevation').gt(0)
# Mask out ocean pixels and clip to the area of interest
predictors = predictors.updateMask(watermask).clip(aoi)
जब एक मॉडल में, आपस में काफ़ी हद तक जुड़े हुए वैरिएबल को एक साथ शामिल किया जाता है, तो मल्टीकोलिनियरिटी की समस्याएं हो सकती हैं. मल्टीकोलिनियरिटी एक ऐसी स्थिति होती है जब किसी मॉडल में इंडिपेंडेंट वैरिएबल के बीच मज़बूत लीनियर रिलेशनशिप होते हैं. इससे मॉडल के कोएफ़िशिएंट (वज़न) के अनुमान में अस्थिरता आती है. इस वजह से, मॉडल की विश्वसनीयता कम हो सकती है. साथ ही, नए डेटा के लिए अनुमान लगाना या उसकी व्याख्या करना मुश्किल हो सकता है. इसलिए, हम मल्टीकोलिनियरिटी पर विचार करेंगे और अनुमान लगाने वाले वैरिएबल चुनने की प्रोसेस को आगे बढ़ाएंगे.
सबसे पहले, हम 5,000 रैंडम पॉइंट जनरेट करेंगे. इसके बाद, उन पॉइंट पर सिंगल मल्टीबैंड इमेज के अनुमान लगाने वाले वैरिएबल की वैल्यू निकालेंगे.
# Generate 5,000 random points
data_cor = predictors.sample(scale=grain_size, numPixels=5000, geometries=True)
# Extract predictor variable values
pvals = predictors.sampleRegions(collection=data_cor, scale=grain_size)
हम हर पॉइंट के लिए निकाली गई अनुमान लगाने वाली वैल्यू को DataFrame में बदलेंगे. इसके बाद, पहली लाइन की जांच करेंगे.
# Converting predictor values from Earth Engine to a DataFrame
pvals_df = geemap.ee_to_df(pvals)
pvals_df.head(1)
# Displaying the columns
columns = pvals_df.columns
print(columns)
दिए गए अनुमान लगाने वाले वैरिएबल के बीच स्पीयरमैन के सहसंबंध गुणांक का हिसाब लगाना और उन्हें हीटमैप में दिखाना.
def plot_correlation_heatmap(dataframe, h_size=10, show_labels=False):
# Calculate Spearman correlation coefficients
correlation_matrix = dataframe.corr(method="spearman")
# Create a heatmap
plt.figure(figsize=(h_size, h_size-2))
plt.imshow(correlation_matrix, cmap='coolwarm', interpolation='nearest')
# Optionally display values on the heatmap
if show_labels:
for i in range(correlation_matrix.shape[0]):
for j in range(correlation_matrix.shape[1]):
plt.text(j, i, f"{correlation_matrix.iloc[i, j]:.2f}",
ha='center', va='center', color='white', fontsize=8)
columns = dataframe.columns.tolist()
plt.xticks(range(len(columns)), columns, rotation=90)
plt.yticks(range(len(columns)), columns)
plt.title("Variables Correlation Matrix")
plt.colorbar(label="Spearman Correlation")
plt.savefig('correlation_heatmap_plot.png')
plt.show()
# Plot the correlation heatmap of variables
plot_correlation_heatmap(pvals_df)
स्पीयरमैन कोरिलेशन कोएफ़िशिएंट, अनुमान लगाने वाले वैरिएबल के बीच सामान्य संबंध को समझने के लिए उपयोगी होता है. हालांकि, यह सीधे तौर पर यह आकलन नहीं करता कि कई वैरिएबल कैसे इंटरैक्ट करते हैं. खास तौर पर, यह मल्टीकोलिनियरिटी का पता नहीं लगाता.
वैरिएंस इनफ़्लेशन फ़ैक्टर (नीचे दिया गया VIF) एक सांख्यिकीय मेट्रिक है. इसका इस्तेमाल मल्टीकोलिनियरिटी का आकलन करने और वैरिएबल चुनने के लिए किया जाता है. इससे यह पता चलता है कि हर इंडिपेंडेंट वैरिएबल का अन्य इंडिपेंडेंट वैरिएबल के साथ लीनियर रिलेशनशिप कितना है. साथ ही, वीआईएफ़ की ज़्यादा वैल्यू, मल्टीकोलिनियरिटी का सबूत हो सकती हैं.
आम तौर पर, जब वीआईएफ़ की वैल्यू 5 या 10 से ज़्यादा होती है, तो इसका मतलब है कि वैरिएबल का अन्य वैरिएबल के साथ काफ़ी जुड़ाव है. इससे मॉडल की स्थिरता और व्याख्या पर असर पड़ सकता है. इस ट्यूटोरियल में, वैरिएबल चुनने के लिए VIF वैल्यू के 10 से कम होने की शर्त का इस्तेमाल किया गया था. वीआईएफ़ के आधार पर, इन छह वैरिएबल को चुना गया था.
# Filter variables based on Variance Inflation Factor (VIF)
def filter_variables_by_vif(dataframe, threshold=10):
original_columns = dataframe.columns.tolist()
remaining_columns = original_columns[:]
while True:
vif_data = dataframe[remaining_columns]
vif_values = [
variance_inflation_factor(vif_data.values, i)
for i in range(vif_data.shape[1])
]
max_vif_index = vif_values.index(max(vif_values))
max_vif = max(vif_values)
if max_vif < threshold:
break
print(f"Removing '{remaining_columns[max_vif_index]}' with VIF {max_vif:.2f}")
del remaining_columns[max_vif_index]
filtered_data = dataframe[remaining_columns]
bands = filtered_data.columns.tolist()
print("Bands:", bands)
return filtered_data, bands
filtered_pvals_df, bands = filter_variables_by_vif(pvals_df)
# Variable Selection Based on VIF
predictors = predictors.select(bands)
# Plot the correlation heatmap of variables
plot_correlation_heatmap(filtered_pvals_df, h_size=6, show_labels=True)
इसके बाद, मैप पर चुने गए छह अनुमान लगाने वाले वैरिएबल को विज़ुअलाइज़ करते हैं.
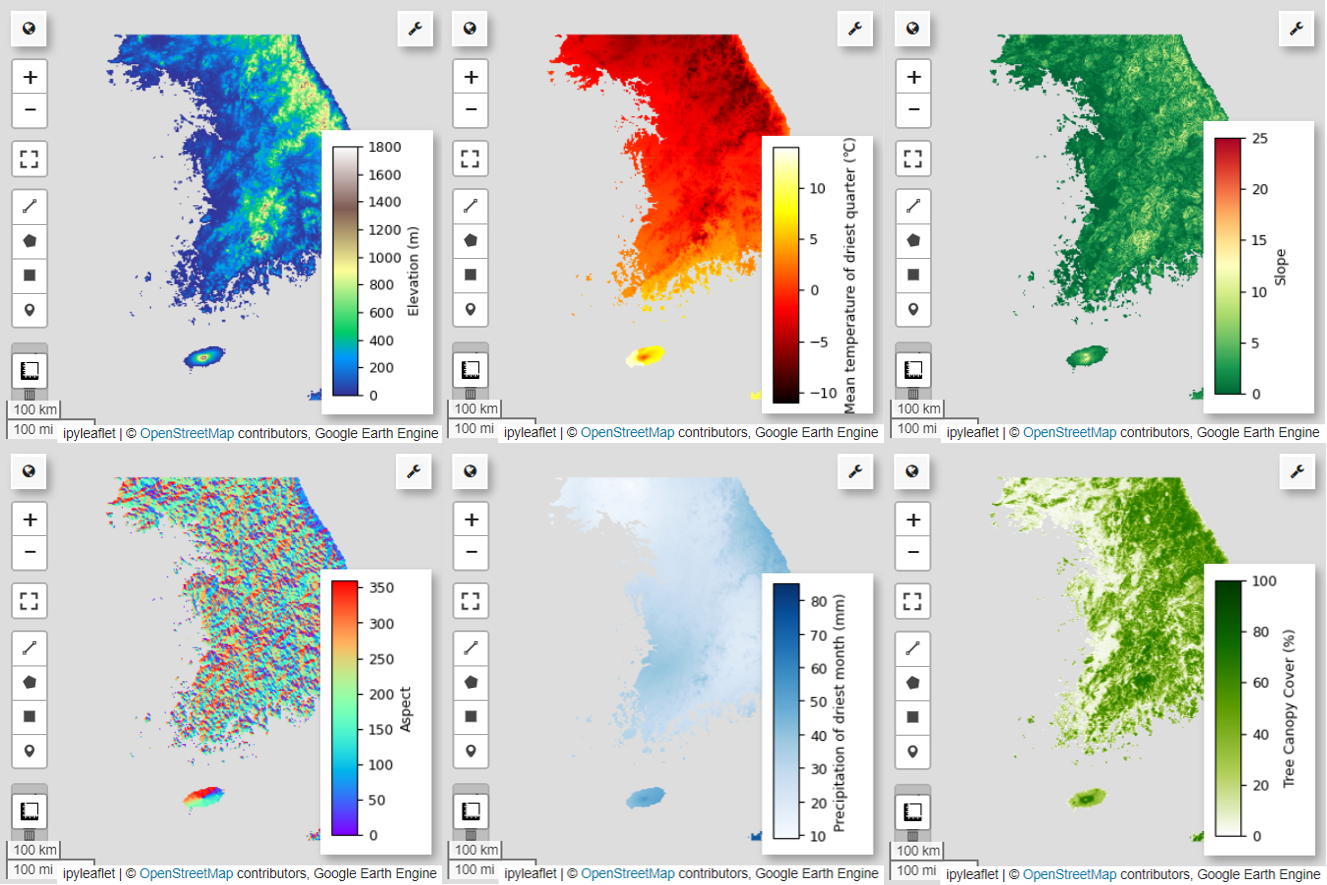
यहां दिए गए कोड का इस्तेमाल करके, मैप विज़ुअलाइज़ेशन के लिए उपलब्ध पैलेट देखे जा सकते हैं. उदाहरण के लिए, terrain पैलेट ऐसा दिखता है.
cm.plot_colormaps(width=8.0, height=0.2)
cm.plot_colormap('terrain', width=8.0, height=0.2, orientation='horizontal')
# Elevation layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['elevation'], 'min': 0, 'max': 1800, 'palette': cm.palettes.terrain}
Map.addLayer(predictors, vis_params, 'elevation')
Map.add_colorbar(vis_params, label="Elevation (m)", orientation="vertical", layer_name="elevation")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio09
min_max_val = (
predictors.select("bio09")
.multiply(0.1)
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio09 (Mean temperature of driest quarter) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"min": math.floor(min_max_val["bio09_min"]),
"max": math.ceil(min_max_val["bio09_max"]),
"palette": cm.palettes.hot,
}
Map.addLayer(predictors.select("bio09").multiply(0.1), vis_params, "bio09")
Map.add_colorbar(
vis_params,
label="Mean temperature of driest quarter (℃)",
orientation="vertical",
layer_name="bio09",
)
Map.centerObject(aoi, 6)
Map
# Slope layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['slope'], 'min': 0, 'max': 25, 'palette': cm.palettes.RdYlGn_r}
Map.addLayer(predictors, vis_params, 'slope')
Map.add_colorbar(vis_params, label="Slope", orientation="vertical", layer_name="slope")
Map.centerObject(aoi, 6)
Map
# Aspect layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['aspect'], 'min': 0, 'max': 360, 'palette': cm.palettes.rainbow}
Map.addLayer(predictors, vis_params, 'aspect')
Map.add_colorbar(vis_params, label="Aspect", orientation="vertical", layer_name="aspect")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio14
min_max_val = (
predictors.select("bio14")
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio14 (Precipitation of driest month) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["bio14"],
"min": math.floor(min_max_val["bio14_min"]),
"max": math.ceil(min_max_val["bio14_max"]),
"palette": cm.palettes.Blues,
}
Map.addLayer(predictors, vis_params, "bio14")
Map.add_colorbar(
vis_params,
label="Precipitation of driest month (mm)",
orientation="vertical",
layer_name="bio14",
)
Map.centerObject(aoi, 6)
Map
# TCC layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["TCC"],
"min": 0,
"max": 100,
"palette": ["ffffff", "afce56", "5f9c00", "0e6a00", "003800"],
}
Map.addLayer(predictors, vis_params, "TCC")
Map.add_colorbar(
vis_params, label="Tree Canopy Cover (%)", orientation="vertical", layer_name="TCC"
)
Map.centerObject(aoi, 6)
Map
छद्म-अनुपस्थिति डेटा जनरेट करना
एसडीएम की प्रोसेस में, किसी प्रजाति के लिए इनपुट डेटा को मुख्य रूप से दो तरीकों से चुना जाता है:
प्रज़ेंस-बैकग्राउंड मेथड: इस तरीके में, किसी खास प्रजाति के देखे जाने (प्रज़ेंस) की जगहों की तुलना उन जगहों से की जाती है जहां उस प्रजाति को नहीं देखा गया है (बैकग्राउंड). यहां बैकग्राउंड डेटा का मतलब यह नहीं है कि प्रजाति उन इलाकों में मौजूद नहीं है. इसका मतलब यह है कि प्रजाति को अध्ययन क्षेत्र की कुल पर्यावरणीय स्थितियों को दिखाने के लिए सेट अप किया गया है. इसका इस्तेमाल, उन जगहों की पहचान करने के लिए किया जाता है जहां प्रजातियां रह सकती हैं. साथ ही, उन जगहों की पहचान करने के लिए भी किया जाता है जहां प्रजातियां नहीं रह सकती हैं.
मौजूदगी-गैरमौजूदगी का तरीका: इस तरीके में, उन जगहों की तुलना की जाती है जहां प्रजाति देखी गई है (मौजूदगी) और उन जगहों की तुलना की जाती है जहां प्रजाति नहीं देखी गई है (गैरमौजूदगी). यहां, प्रजाति के न होने का डेटा उन जगहों के बारे में बताता है जहां प्रजाति मौजूद नहीं है. इससे अध्ययन क्षेत्र की पर्यावरण से जुड़ी पूरी जानकारी नहीं मिलती है. इसके बजाय, यह उन जगहों के बारे में बताता है जहां प्रजाति के मौजूद न होने का अनुमान है.
असल में, किसी प्रजाति के न होने का सही डेटा इकट्ठा करना अक्सर मुश्किल होता है. इसलिए, आर्टिफ़िशियल तरीके से जनरेट किए गए छद्म-अनुपस्थिति डेटा का इस्तेमाल अक्सर किया जाता है. हालांकि, इस तरीके की सीमाओं और संभावित गड़बड़ियों को स्वीकार करना ज़रूरी है. ऐसा इसलिए, क्योंकि आर्टिफ़िशियल इंटेलिजेंस से जनरेट किए गए छद्म-अनुपस्थिति पॉइंट, अनुपस्थिति वाले असली क्षेत्रों को सटीक तरीके से नहीं दिखा सकते.
इन दोनों तरीकों में से किसी एक को चुनने का फ़ैसला, डेटा की उपलब्धता, रिसर्च के लक्ष्यों, मॉडल के सटीक और भरोसेमंद होने के साथ-साथ समय और संसाधनों पर निर्भर करता है. यहां हम "Presence-Absence" तरीके का इस्तेमाल करके मॉडल बनाने के लिए, GBIF से इकट्ठा किए गए डेटा और आर्टिफ़िशियल तरीके से जनरेट किए गए स्यूडो-ऐब्सेंस डेटा का इस्तेमाल करेंगे.
"एनवायरमेंटल प्रोफ़ाइलिंग अप्रोच" का इस्तेमाल करके, छद्म-अनुपस्थिति डेटा जनरेट किया जाएगा. इसके लिए, यह तरीका अपनाया जाएगा:
के-मीन्स क्लस्टरिंग का इस्तेमाल करके, पर्यावरण के हिसाब से कैटगरी तय करना: यूक्लिडियन दूरी पर आधारित के-मीन्स क्लस्टरिंग एल्गोरिदम का इस्तेमाल, स्टडी एरिया में मौजूद पिक्सल को दो क्लस्टर में बांटने के लिए किया जाएगा. एक क्लस्टर, उन इलाकों को दिखाएगा जहां पर्यावरण की विशेषताएं, रैंडम तरीके से चुनी गई 100 जगहों से मिलती-जुलती हैं. वहीं, दूसरा क्लस्टर उन इलाकों को दिखाएगा जहां पर्यावरण की विशेषताएं अलग हैं.
मिलते-जुलते क्लस्टर में छद्म-अनुपस्थिति डेटा जनरेट करना: पहले चरण में पहचाने गए दूसरे क्लस्टर में (जिसमें उपस्थिति डेटा से अलग पर्यावरणीय विशेषताएं हैं), छद्म-अनुपस्थिति पॉइंट को रैंडम तरीके से जनरेट किया जाएगा. ये छद्म-अनुपस्थिति पॉइंट, उन जगहों को दिखाएंगे जहां प्रजाति के मौजूद होने की उम्मीद नहीं है.
# Randomly select 100 locations for occurrence
pvals = predictors.sampleRegions(
collection=data.randomColumn().sort('random').limit(100),
properties=[],
scale=grain_size
)
# Perform k-means clustering
clusterer = ee.Clusterer.wekaKMeans(
nClusters=2,
distanceFunction="Euclidean"
).train(pvals)
cl_result = predictors.cluster(clusterer)
# Get cluster ID for locations similar to occurrence
cl_id = cl_result.sampleRegions(
collection=data.randomColumn().sort('random').limit(200),
properties=[],
scale=grain_size
)
# Define non-occurrence areas in dissimilar clusters
cl_id = ee.FeatureCollection(cl_id).reduceColumns(ee.Reducer.mode(),['cluster'])
cl_id = ee.Number(cl_id.get('mode')).subtract(1).abs()
cl_mask = cl_result.select(['cluster']).eq(cl_id)
# Presence location mask
presence_mask = data.reduceToImage(properties=['random'],
reducer=ee.Reducer.first()
).reproject('EPSG:4326', None,
grain_size).mask().neq(1).selfMask()
# Masking presence locations in non-occurrence areas and clipping to AOI
area_for_pa = presence_mask.updateMask(cl_mask).clip(aoi)
# Area for Pseudo-absence
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(area_for_pa, {'palette': 'black'}, 'AreaForPA')
Map.centerObject(aoi, 6)
Map
मॉडल फ़िटिंग और अनुमान
अब हम डेटा को ट्रेनिंग डेटा और टेस्ट डेटा में बांटेंगे. ट्रेनिंग डेटा का इस्तेमाल, मॉडल को ट्रेनिंग देकर सबसे सही पैरामीटर ढूंढने के लिए किया जाएगा. वहीं, टेस्ट डेटा का इस्तेमाल, पहले से ट्रेन किए गए मॉडल का आकलन करने के लिए किया जाएगा. इस संदर्भ में, एक अहम कॉन्सेप्ट पर ध्यान देना ज़रूरी है. यह कॉन्सेप्ट, स्पैटियल ऑटोकोरिलेशन है.
एसडीएम में स्पेशल ऑटोकोरिलेशन एक ज़रूरी एलिमेंट है. यह टॉबलर के नियम से जुड़ा है. यह इस कॉन्सेप्ट पर आधारित है कि "हर चीज़, किसी न किसी तरह से दूसरी चीज़ से जुड़ी होती है. हालांकि, जो चीज़ें एक-दूसरे के नज़दीक होती हैं वे दूर मौजूद चीज़ों के मुकाबले ज़्यादा जुड़ी होती हैं". स्पेशल ऑटोकोरिलेशन से पता चलता है कि किसी प्रजाति की जगह और पर्यावरण से जुड़े वैरिएबल के बीच क्या संबंध है. हालांकि, अगर ट्रेनिंग और टेस्ट डेटा के बीच स्पैटियल ऑटोकोरिलेशन मौजूद है, तो दोनों डेटा सेट के बीच इंडिपेंडेंस से समझौता किया जा सकता है. इससे मॉडल की सामान्यीकरण क्षमता का आकलन करने पर काफ़ी असर पड़ता है.
इस समस्या को हल करने का एक तरीका, स्पैटियल ब्लॉक क्रॉस-वैलिडेशन तकनीक है. इसमें डेटा को ट्रेनिंग और टेस्टिंग डेटासेट में बांटा जाता है. इस तकनीक में, डेटा को कई ब्लॉक में बांटा जाता है. साथ ही, हर ब्लॉक का इस्तेमाल ट्रेनिंग और टेस्ट डेटासेट के तौर पर अलग-अलग किया जाता है, ताकि स्पैटियल ऑटोकोरिलेशन के असर को कम किया जा सके. इससे डेटासेट के बीच निर्भरता कम हो जाती है. इससे मॉडल की सामान्यीकरण क्षमता का ज़्यादा सटीक आकलन किया जा सकता है.
इसके लिए, यह तरीका अपनाएं:
- स्पेशल ब्लॉक बनाना: पूरे डेटासेट को एक जैसे साइज़ के स्पेशल ब्लॉक में बांटें. उदाहरण के लिए, 50x50 कि॰मी॰).
- ट्रेनिंग और टेस्टिंग सेट असाइन करना: हर स्पैटियल ब्लॉक को ट्रेनिंग सेट (70%) या टेस्ट सेट (30%) में से किसी एक को रैंडम तरीके से असाइन किया जाता है. इससे मॉडल को किसी खास इलाके के डेटा के हिसाब से फ़िट होने से रोका जा सकता है. साथ ही, इसका मकसद ज़्यादा सामान्य नतीजे पाना है.
- बार-बार क्रॉस-वैलिडेशन: पूरी प्रोसेस को n बार दोहराया जाता है. उदाहरण के लिए, 10 बार). हर बार, ब्लॉक को ट्रेनिंग और टेस्ट सेट में फिर से बांटा जाता है. इससे मॉडल की स्थिरता और भरोसेमंदता को बेहतर बनाने में मदद मिलती है.
- स्यूडो-ऐब्सेंस डेटा जनरेट करना: हर इटरेशन में, मॉडल की परफ़ॉर्मेंस का आकलन करने के लिए, स्यूडो-ऐब्सेंस डेटा को रैंडम तरीके से जनरेट किया जाता है.
Scale = 50000
grid = watermask.reduceRegions(
collection=aoi.coveringGrid(scale=Scale, proj='EPSG:4326'),
reducer=ee.Reducer.mean()).filter(ee.Filter.neq('mean', None))
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(grid, {}, "Grid for spatial block cross validation")
Map.addLayer(outline, {'palette': 'FF0000'}, "Study Area")
Map.centerObject(aoi, 6)
Map
अब हम मॉडल को फ़िट कर सकते हैं. मॉडल को फ़िट करने का मतलब है कि डेटा में मौजूद पैटर्न को समझना और उसके हिसाब से मॉडल के पैरामीटर (वज़न और पूर्वाग्रह) को अडजस्ट करना. इस प्रोसेस की मदद से, मॉडल को नया डेटा मिलने पर ज़्यादा सटीक अनुमान लगाने में मदद मिलती है. इसके लिए, हमने SDM() नाम का एक फ़ंक्शन तय किया है, ताकि मॉडल को फ़िट किया जा सके.
हम रैंडम फ़ॉरेस्ट एल्गोरिदम का इस्तेमाल करेंगे.
def sdm(x):
seed = ee.Number(x)
# Random block division for training and validation
rand_blk = ee.FeatureCollection(grid).randomColumn(seed=seed).sort("random")
training_grid = rand_blk.filter(ee.Filter.lt("random", split)) # Grid for training
testing_grid = rand_blk.filter(ee.Filter.gte("random", split)) # Grid for testing
# Presence points
presence_points = ee.FeatureCollection(data)
presence_points = presence_points.map(lambda feature: feature.set("PresAbs", 1))
tr_presence_points = presence_points.filter(
ee.Filter.bounds(training_grid)
) # Presence points for training
te_presence_points = presence_points.filter(
ee.Filter.bounds(testing_grid)
) # Presence points for testing
# Pseudo-absence points for training
tr_pseudo_abs_points = area_for_pa.sample(
region=training_grid,
scale=grain_size,
numPixels=tr_presence_points.size().add(300),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for training
tr_pseudo_abs_points = (
tr_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(tr_presence_points.size()))
)
tr_pseudo_abs_points = tr_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
te_pseudo_abs_points = area_for_pa.sample(
region=testing_grid,
scale=grain_size,
numPixels=te_presence_points.size().add(100),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for testing
te_pseudo_abs_points = (
te_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(te_presence_points.size()))
)
te_pseudo_abs_points = te_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
# Merge training and pseudo-absence points
training_partition = tr_presence_points.merge(tr_pseudo_abs_points)
testing_partition = te_presence_points.merge(te_pseudo_abs_points)
# Extract predictor variable values at training points
train_pvals = predictors.sampleRegions(
collection=training_partition,
properties=["PresAbs"],
scale=grain_size,
geometries=True,
)
# Random Forest classifier
classifier = ee.Classifier.smileRandomForest(
numberOfTrees=500,
variablesPerSplit=None,
minLeafPopulation=10,
bagFraction=0.5,
maxNodes=None,
seed=seed,
)
# Presence probability: Habitat suitability map
classifier_pr = classifier.setOutputMode("PROBABILITY").train(
train_pvals, "PresAbs", bands
)
classified_img_pr = predictors.select(bands).classify(classifier_pr)
# Binary presence/absence map: Potential distribution map
classifier_bin = classifier.setOutputMode("CLASSIFICATION").train(
train_pvals, "PresAbs", bands
)
classified_img_bin = predictors.select(bands).classify(classifier_bin)
return [
classified_img_pr,
classified_img_bin,
training_partition,
testing_partition,
], classifier_pr
स्पेशल ब्लॉक को मॉडल ट्रेनिंग के लिए 70% और मॉडल टेस्टिंग के लिए 30% के हिसाब से बांटा जाता है. हर ट्रेनिंग और टेस्टिंग सेट में, हर बार छद्म-अनुपस्थिति डेटा को रैंडम तरीके से जनरेट किया जाता है. इस वजह से, हर बार मॉडल को ट्रेनिंग देने और उसकी जांच करने के लिए, अलग-अलग डेटा सेट मिलते हैं.
split = 0.7
numiter = 10
# Random Seed
runif = lambda length: [random.randint(1, 1000) for _ in range(length)]
items = runif(numiter)
# Fixed seed
# items = [287, 288, 553, 226, 151, 255, 902, 267, 419, 538]
results_list = [] # Initialize SDM results list
importances_list = [] # Initialize variable importance list
for item in items:
result, trained = sdm(item)
# Accumulate SDM results into the list
results_list.extend(result)
# Accumulate variable importance into the list
importance = ee.Dictionary(trained.explain()).get('importance')
importances_list.extend(importance.getInfo().items())
# Flatten the SDM results list
results = ee.List(results_list).flatten()
अब हम फ़ेयरी पिट्टा के लिए, हैबिटैट सूटेबिलिटी मैप और संभावित डिस्ट्रिब्यूशन मैप को विज़ुअलाइज़ कर सकते हैं. इस मामले में, mean() फ़ंक्शन का इस्तेमाल करके, हर पिक्सल की जगह के लिए सभी इमेज का औसत निकालकर, हैबिटैट के हिसाब से सही जगह का मैप बनाया जाता है. साथ ही, mode() फ़ंक्शन का इस्तेमाल करके, हर पिक्सल की जगह के लिए सभी इमेज में सबसे ज़्यादा बार दिखने वाली वैल्यू का पता लगाकर, संभावित डिस्ट्रिब्यूशन मैप जनरेट किया जाता है.
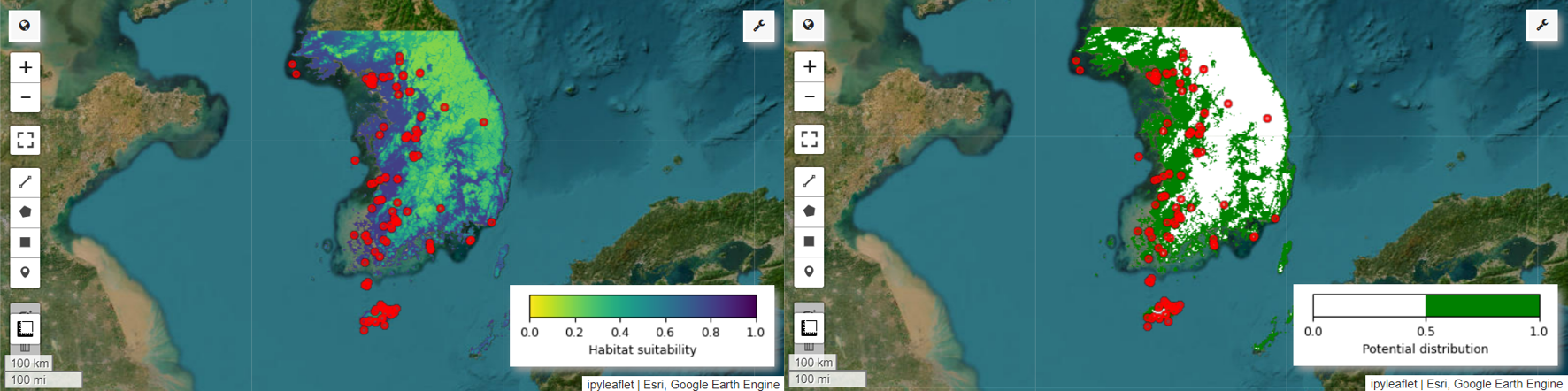
# Habitat suitability map
images = ee.List.sequence(
0, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x))
model_average = ee.ImageCollection.fromImages(images).mean()
Map = geemap.Map(layout={'height':'400px', 'width':'800px'}, basemap='Esri.WorldImagery')
vis_params = {
'min': 0,
'max': 1,
'palette': cm.palettes.viridis_r}
Map.addLayer(model_average, vis_params, 'Habitat suitability')
Map.add_colorbar(vis_params, label="Habitat suitability",
orientation="horizontal",
layer_name="Habitat suitability")
Map.addLayer(data, {'color':'red'}, 'Presence')
Map.centerObject(aoi, 6)
Map
# Potential distribution map
images2 = ee.List.sequence(1, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x)
)
distribution_map = ee.ImageCollection.fromImages(images2).mode()
Map = geemap.Map(
layout={"height": "400px", "width": "800px"}, basemap="Esri.WorldImagery"
)
vis_params = {"min": 0, "max": 1, "palette": ["white", "green"]}
Map.addLayer(distribution_map, vis_params, "Potential distribution")
Map.addLayer(data, {"color": "red"}, "Presence")
Map.add_colorbar(
vis_params,
label="Potential distribution",
discrete=True,
orientation="horizontal",
layer_name="Potential distribution",
)
Map.centerObject(data.geometry(), 6)
Map
वैरिएबल की अहमियत और सटीक आकलन
रैंडम फ़ॉरेस्ट (ee.Classifier.smileRandomForest), ग्रुप में सीखने के तरीकों में से एक है. यह अनुमान लगाने के लिए, कई डिसिज़न ट्री बनाता है. हर फ़ैसले का ट्री, डेटा के अलग-अलग सबसेट से स्वतंत्र रूप से सीखता है. साथ ही, ज़्यादा सटीक और स्थिर अनुमानों को चालू करने के लिए, उनके नतीजों को इकट्ठा किया जाता है.
वेरिएबल की अहमियत एक ऐसा मेज़रमेंट है जो Random Forest मॉडल में, अनुमानों पर हर वेरिएबल के असर का आकलन करता है. हम पहले से तय किए गए importances_list का इस्तेमाल करके, वैरिएबल की औसत अहमियत का हिसाब लगाएंगे और उसे प्रिंट करेंगे.
def plot_variable_importance(importances_list):
# Extract each variable importance value into a list
variables = [item[0] for item in importances_list]
importances = [item[1] for item in importances_list]
# Calculate the average importance for each variable
average_importances = {}
for variable in set(variables):
indices = [i for i, var in enumerate(variables) if var == variable]
average_importance = np.mean([importances[i] for i in indices])
average_importances[variable] = average_importance
# Sort the importances in descending order of importance
sorted_importances = sorted(average_importances.items(),
key=lambda x: x[1], reverse=False)
variables = [item[0] for item in sorted_importances]
avg_importances = [item[1] for item in sorted_importances]
# Adjust the graph size
plt.figure(figsize=(8, 4))
# Plot the average importance as a horizontal bar chart
plt.barh(variables, avg_importances)
plt.xlabel('Importance')
plt.ylabel('Variables')
plt.title('Average Variable Importance')
# Display values above the bars
for i, v in enumerate(avg_importances):
plt.text(v + 0.02, i, f"{v:.2f}", va='center')
# Adjust the x-axis range
plt.xlim(0, max(avg_importances) + 5) # Adjust to the desired range
plt.tight_layout()
plt.savefig('variable_importance.png')
plt.show()
plot_variable_importance(importances_list)
हम टेस्टिंग डेटासेट का इस्तेमाल करके, हर रन के लिए AUC-ROC और AUC-PR का हिसाब लगाते हैं. इसके बाद, हम n बार दोहराए गए प्रोसेस के आधार पर, औसत AUC-ROC और AUC-PR का हिसाब लगाते हैं.
एयूसी-आरओसी, 'सेंसिटिविटी (रीकॉल) बनाम 1-स्पेसिफ़िसिटी' ग्राफ़ के कर्व के नीचे के एरिया को दिखाता है. इससे थ्रेशोल्ड में बदलाव होने पर, सेंसिटिविटी और स्पेसिफ़िसिटी के बीच के संबंध के बारे में पता चलता है. खास जानकारी, उन सभी मामलों पर आधारित होती है जिनमें इवेंट नहीं हुआ. इसलिए, एयूसी-आरओसी में कन्फ़्यूज़न मैट्रिक्स के सभी क्वाड्रेंट शामिल होते हैं.
AUC-PR, 'प्रिसिज़न बनाम रीकॉल (सेंसिटिविटी)' ग्राफ़ के कर्व के नीचे का एरिया दिखाता है. इससे थ्रेशोल्ड में बदलाव होने पर, प्रिसिज़न और रीकॉल के बीच का संबंध पता चलता है. सटीकता, अनुमानित सभी इवेंट के आधार पर तय की जाती है. इसलिए, AUC-PR में ट्रू नेगेटिव (टीएन) शामिल नहीं होते.
def print_pres_abs_sizes(TestingDatasets, numiter):
# Check and print the sizes of presence and pseudo-absence coordinates
def get_pres_abs_size(x):
fc = ee.FeatureCollection(TestingDatasets.get(x))
presence_size = fc.filter(ee.Filter.eq("PresAbs", 1)).size()
pseudo_absence_size = fc.filter(ee.Filter.eq("PresAbs", 0)).size()
return ee.List([presence_size, pseudo_absence_size])
sizes_info = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(get_pres_abs_size)
.getInfo()
)
for i, sizes in enumerate(sizes_info):
presence_size = sizes[0]
pseudo_absence_size = sizes[1]
print(
f"Iteration {i + 1}: Presence Size = {presence_size}, Pseudo-absence Size = {pseudo_absence_size}"
)
# Extracting the Testing Datasets
testing_datasets = ee.List.sequence(
3, ee.Number(numiter).multiply(4).subtract(1), 4
).map(lambda x: results.get(x))
print_pres_abs_sizes(testing_datasets, numiter)
def get_acc(hsm, t_data, grain_size):
pr_prob_vals = hsm.sampleRegions(
collection=t_data, properties=["PresAbs"], scale=grain_size
)
seq = ee.List.sequence(start=0, end=1, count=25) # Divide 0 to 1 into 25 intervals
def calculate_metrics(cutoff):
# Each element of the seq list is passed as cutoff(threshold value)
# Observed present = TP + FN
pres = pr_prob_vals.filterMetadata("PresAbs", "equals", 1)
# TP (True Positive)
tp = ee.Number(
pres.filterMetadata("classification", "greater_than", cutoff).size()
)
# TPR (True Positive Rate) = Recall = Sensitivity = TP / (TP + FN) = TP / Observed present
tpr = tp.divide(pres.size())
# Observed absent = FP + TN
abs = pr_prob_vals.filterMetadata("PresAbs", "equals", 0)
# FN (False Negative)
fn = ee.Number(
pres.filterMetadata("classification", "less_than", cutoff).size()
)
# TNR (True Negative Rate) = Specificity = TN / (FP + TN) = TN / Observed absent
tn = ee.Number(abs.filterMetadata("classification", "less_than", cutoff).size())
tnr = tn.divide(abs.size())
# FP (False Positive)
fp = ee.Number(
abs.filterMetadata("classification", "greater_than", cutoff).size()
)
# FPR (False Positive Rate) = FP / (FP + TN) = FP / Observed absent
fpr = fp.divide(abs.size())
# Precision = TP / (TP + FP) = TP / Predicted present
precision = tp.divide(tp.add(fp))
# SUMSS = SUM of Sensitivity and Specificity
sumss = tpr.add(tnr)
return ee.Feature(
None,
{
"cutoff": cutoff,
"TP": tp,
"TN": tn,
"FP": fp,
"FN": fn,
"TPR": tpr,
"TNR": tnr,
"FPR": fpr,
"Precision": precision,
"SUMSS": sumss,
},
)
return ee.FeatureCollection(seq.map(calculate_metrics))
def calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter):
# Calculate AUC-ROC and AUC-PR
def calculate_auc_metrics(x):
hsm = ee.Image(images.get(x))
t_data = ee.FeatureCollection(testing_datasets.get(x))
acc = get_acc(hsm, t_data, grain_size)
# Calculate AUC-ROC
x = ee.Array(acc.aggregate_array("FPR"))
y = ee.Array(acc.aggregate_array("TPR"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_roc = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
# Calculate AUC-PR
x = ee.Array(acc.aggregate_array("TPR"))
y = ee.Array(acc.aggregate_array("Precision"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_pr = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
return (auc_roc, auc_pr)
auc_metrics = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(calculate_auc_metrics)
.getInfo()
)
# Print AUC-ROC and AUC-PR for each iteration
df = pd.DataFrame(auc_metrics, columns=["AUC-ROC", "AUC-PR"])
df.index = [f"Iteration {i + 1}" for i in range(len(df))]
df.to_csv("auc_metrics.csv", index_label="Iteration")
print(df)
# Calculate mean and standard deviation of AUC-ROC and AUC-PR
mean_auc_roc, std_auc_roc = df["AUC-ROC"].mean(), df["AUC-ROC"].std()
mean_auc_pr, std_auc_pr = df["AUC-PR"].mean(), df["AUC-PR"].std()
print(f"Mean AUC-ROC = {mean_auc_roc:.4f} ± {std_auc_roc:.4f}")
print(f"Mean AUC-PR = {mean_auc_pr:.4f} ± {std_auc_pr:.4f}")
%%time
# Calculate AUC-ROC and AUC-PR
calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter)
इस ट्यूटोरियल में, प्रजातियों के डिस्ट्रिब्यूशन की मॉडलिंग (एसडीएम) के लिए, Google Earth Engine (GEE) का इस्तेमाल करने का एक व्यावहारिक उदाहरण दिया गया है. इस रिसर्च से यह अहम जानकारी मिलती है कि एसडीएम के क्षेत्र में, GEE का इस्तेमाल कई तरह से किया जा सकता है और इसे आसानी से लागू किया जा सकता है. Earth Engine की जियोस्पेशल डेटा प्रोसेसिंग की बेहतरीन सुविधाओं का इस्तेमाल करके, रिसर्चर और संरक्षणवादियों को हमारे ग्रह पर जैव-विविधता को समझने और उसे सुरक्षित रखने के लिए कई अवसर मिलते हैं. इस ट्यूटोरियल से मिली जानकारी और सीखी गई बातों का इस्तेमाल करके, व्यक्ति इस दिलचस्प फ़ील्ड यानी कि इकोलॉजिकल रिसर्च के बारे में ज़्यादा जान सकते हैं और इसमें योगदान दे सकते हैं.
 Google Colab में चलाएं
Google Colab में चलाएं
 GitHub पर सोर्स देखें
GitHub पर सोर्स देखें