В этом уроке будет представлена методология моделирования распространения видов с использованием Google Earth Engine. Будет представлен краткий обзор моделирования распределения видов, за которым последует процесс прогнозирования и анализа среды обитания исчезающего вида птиц, известного как Фея питта (научное название: Питта нимфа ).
Сначала запусти меня
Запустите следующую ячейку, чтобы инициализировать API. Вывод будет содержать инструкции о том, как предоставить этому блокноту доступ к Earth Engine с помощью вашей учетной записи.
import ee
# Trigger the authentication flow.
ee.Authenticate()
# Initialize the library.
ee.Initialize(project='my-project')
Краткий обзор моделирования распространения видов
Давайте рассмотрим, что такое модели распространения видов, преимущества использования Google Earth Engine для их обработки, необходимые данные для моделей и как структурирован рабочий процесс.
Что такое моделирование распространения видов?
Моделирование распространения видов (ниже SDM) является наиболее распространенной методологией, используемой для оценки фактического или потенциального географического распространения вида. Он включает в себя характеристику условий окружающей среды, подходящих для конкретного вида, а затем определение географического распространения этих подходящих условий.
В последние годы SDM стал важнейшим компонентом природоохранного планирования, и для этой цели были разработаны различные методы моделирования. Реализация SDM в Google Earth Engine (GEE ниже) обеспечивает легкий доступ к крупномасштабным данным об окружающей среде, а также мощные вычислительные возможности и поддержку алгоритмов машинного обучения, что позволяет быстро моделировать.
Данные, необходимые для SDM
SDM обычно использует взаимосвязь между записями о встречаемости известных видов и переменными окружающей среды для определения условий, при которых популяция может существовать. Другими словами, требуются два типа входных данных модели:
- Записи о встречаемости известных видов
- Различные переменные окружающей среды
Эти данные вводятся в алгоритмы определения условий окружающей среды, связанных с присутствием видов.
Рабочий процесс SDM с использованием GEE
Рабочий процесс SDM с использованием GEE выглядит следующим образом:
- Сбор и предварительная обработка данных о встречаемости видов
- Определение области интересов
- Добавление переменных среды GEE
- Генерация данных о псевдоотсутствии
- Подбор модели и прогнозирование
- Оценка важности и точности переменных
Прогнозирование и анализ среды обитания с использованием GEE
Фея Питта ( Питта нимфа ) будет использоваться в качестве примера для демонстрации применения SDM на основе GEE. Хотя этот конкретный вид был выбран для одного примера, исследователи могут применить эту методологию к любому целевому виду, представляющему интерес, с небольшими изменениями в предоставленном исходном коде.
Сказочная питта — редкий летний мигрант и проходной мигрант в Южной Корее, ареал распространения которого расширяется в связи с недавним потеплением климата на Корейском полуострове. Он отнесен к редким видам, находящимся под угрозой исчезновения класса II дикой природы, памятнику природы № 204, оценен как регионально вымерший (RE) в Национальном Красном списке и уязвимый (VU) по категориям МСОП.
Проведение SDM для планирования сохранения питты Феи представляется весьма ценным. Теперь давайте продолжим прогнозирование и анализ среды обитания с помощью GEE.
Сначала импортируются библиотеки Python. Оператор import импортирует все содержимое модуля, а оператор from import позволяет импортировать определенные объекты из модуля.
# Import libraries
import geemap
import geemap.colormaps as cm
import pandas as pd, geopandas as gpd
import numpy as np, matplotlib.pyplot as plt
import os, requests, math, random
from ipyleaflet import TileLayer
from statsmodels.stats.outliers_influence import variance_inflation_factor
Сбор и предварительная обработка данных о встречаемости видов
Теперь давайте соберем данные о возникновении Питты Феи. Даже если в настоящее время у вас нет доступа к данным о встречаемости интересующих вас видов, вы можете получить данные наблюдений за конкретными видами через API GBIF. API GBIF — это интерфейс, который обеспечивает доступ к данным о распространении видов, предоставленным GBIF, позволяя пользователям искать, фильтровать и загружать данные, а также получать различную информацию, связанную с видами.
В приведенном ниже коде переменной species_name присвоено научное название вида (например, «Питта-нимфа» для «Феи-питты»), а переменной country_code присвоен код страны (например, KR для Южной Кореи). Переменная base_url хранит адрес GBIF API. params — это словарь, содержащий параметры, которые будут использоваться в запросе API:
-
scientificName: устанавливает научное название искомого вида. -
country: ограничивает поиск определенной страной. -
hasCoordinate: обеспечивает поиск только данных с координатами (true). -
basisOfRecord: выбирает только записи человеческого наблюдения (HUMAN_OBSERVATION). -
limit: устанавливает максимальное количество возвращаемых результатов, равное 10 000.
def get_gbif_species_data(species_name, country_code):
"""
Retrieves observational data for a specific species using the GBIF API and returns it as a pandas DataFrame.
Parameters:
species_name (str): The scientific name of the species to query.
country_code (str): The country code of the where the observation data will be queried.
Returns:
pd.DataFrame: A pandas DataFrame containing the observational data.
"""
base_url = "https://api.gbif.org/v1/occurrence/search"
params = {
"scientificName": species_name,
"country": country_code,
"hasCoordinate": "true",
"basisOfRecord": "HUMAN_OBSERVATION",
"limit": 10000,
}
try:
response = requests.get(base_url, params=params)
response.raise_for_status() # Raises an exception for a response error.
data = response.json()
occurrences = data.get("results", [])
if occurrences: # If data is present
df = pd.json_normalize(occurrences)
return df
else:
print("No data found for the given species and country code.")
return pd.DataFrame() # Returns an empty DataFrame
except requests.RequestException as e:
print(f"Request failed: {e}")
return pd.DataFrame() # Returns an empty DataFrame in case of an exception
Используя заданные ранее параметры, мы запрашиваем у GBIF API записи наблюдений Феи Питты ( Питта-нимфа ) и загружаем результаты в DataFrame, чтобы проверить первую строку. DataFrame — это структура данных для обработки данных в формате таблицы, состоящая из строк и столбцов. При необходимости DataFrame можно сохранить в виде файла CSV и прочитать обратно.
# Retrieve Fairy Pitta data
df = get_gbif_species_data("Pitta nympha", "KR")
"""
# Save DataFrame to CSV and read back in.
df.to_csv("pitta_nympha_data.csv", index=False)
df = pd.read_csv("pitta_nympha_data.csv")
"""
df.head(1) # Display the first row of the DataFrame
Затем мы преобразуем DataFrame в GeoDataFrame, который включает столбец для географической информации ( geometry ), и проверяем первую строку. GeoDataFrame можно сохранить как файл GeoPackage (*.gpkg) и прочитать обратно.
# Convert DataFrame to GeoDataFrame
gdf = gpd.GeoDataFrame(
df,
geometry=gpd.points_from_xy(df.decimalLongitude,
df.decimalLatitude),
crs="EPSG:4326"
)[["species", "year", "month", "geometry"]]
"""
# Convert GeoDataFrame to GeoPackage (requires pycrs module)
%pip install -U -q pycrs
gdf.to_file("pitta_nympha_data.gpkg", driver="GPKG")
gdf = gpd.read_file("pitta_nympha_data.gpkg")
"""
gdf.head(1) # Display the first row of the GeoDataFrame
На этот раз мы создали функцию для визуализации распределения данных по годам и месяцам из GeoDataFrame и отображения их в виде графика, который затем можно сохранить в виде файла изображения. Использование тепловой карты позволяет нам быстро определить частоту встречаемости видов по годам и месяцам, обеспечивая интуитивно понятную визуализацию временных изменений и закономерностей в данных. Это позволяет выявлять временные закономерности и сезонные вариации в данных о встречаемости видов, а также быстро выявлять выбросы или проблемы с качеством данных.
# Yearly and monthly data distribution heatmap
def plot_heatmap(gdf, h_size=8):
statistics = gdf.groupby(["month", "year"]).size().unstack(fill_value=0)
# Heatmap
plt.figure(figsize=(h_size, h_size - 6))
heatmap = plt.imshow(
statistics.values, cmap="YlOrBr", origin="upper", aspect="auto"
)
# Display values above each pixel
for i in range(len(statistics.index)):
for j in range(len(statistics.columns)):
plt.text(
j, i, statistics.values[i, j], ha="center", va="center", color="black"
)
plt.colorbar(heatmap, label="Count")
plt.title("Monthly Species Count by Year")
plt.xlabel("Year")
plt.ylabel("Month")
plt.xticks(range(len(statistics.columns)), statistics.columns)
plt.yticks(range(len(statistics.index)), statistics.index)
plt.tight_layout()
plt.savefig("heatmap_plot.png")
plt.show()
plot_heatmap(gdf)
Данные за 1995 год очень скудны, со значительными пробелами по сравнению с другими годами, а август и сентябрь также имеют ограниченные выборки и демонстрируют другие сезонные характеристики по сравнению с другими периодами. Исключение этих данных может способствовать повышению стабильности и прогнозирующей способности модели.
Однако важно отметить, что исключение данных может повысить способность модели к обобщению, но также может привести к потере ценной информации, имеющей отношение к целям исследования. Поэтому такие решения следует принимать взвешенно.
# Filtering data by year and month
filtered_gdf = gdf[
(~gdf['year'].eq(1995)) &
(~gdf['month'].between(8, 9))
]
Теперь отфильтрованный GeoDataFrame преобразуется в объект Google Earth Engine.
# Convert GeoDataFrame to Earth Engine object
data_raw = geemap.geopandas_to_ee(filtered_gdf)
Далее мы определим размер растрового пикселя результатов SDM как разрешение 1 км.
# Spatial resolution setting (meters)
grain_size = 1000
Когда в пределах одного и того же растрового пикселя с разрешением 1 км присутствует несколько точек возникновения, существует высокая вероятность того, что они находятся в одинаковых условиях окружающей среды в одном и том же географическом месте. Использование таких данных непосредственно в анализе может привести к смещению результатов.
Другими словами, нам необходимо ограничить потенциальное влияние систематической ошибки географической выборки. Для этого мы сохраним только одно местоположение в пределах каждого пикселя размером 1 км и удалим все остальные, что позволит модели более объективно отражать условия окружающей среды.
def remove_duplicates(data, grain_size):
# Select one occurrence record per pixel at the chosen spatial resolution
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
rand_point_vals = random_raster.sampleRegions(
collection=ee.FeatureCollection(data), geometries=True
)
return rand_point_vals.distinct("random")
data = remove_duplicates(data_raw, grain_size)
# Before selection and after selection
print("Original data size:", data_raw.size().getInfo())
print("Final data size:", data.size().getInfo())
Визуализация, сравнивающая смещение географической выборки до предварительной обработки (синий цвет) и после предварительной обработки (красный цвет), показана ниже. Для облегчения сравнения карта была сосредоточена на районе с высокой концентрацией координат появления Фейри-питты в национальном парке Халласан.
# Visualization of geographic sampling bias before (blue) and after (red) preprocessing
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
# Add the random raster layer
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
Map.addLayer(
random_raster,
{"min": 0, "max": 1, "palette": ["black", "white"], "opacity": 0.5},
"Random Raster",
)
# Add the original data layer in blue
Map.addLayer(data_raw, {"color": "blue"}, "Original data")
# Add the final data layer in red
Map.addLayer(data, {"color": "red"}, "Final data")
# Set the center of the map to the coordinates
Map.setCenter(126.712, 33.516, 14)
Map
Определение области интересов
Определение области интереса (AOI ниже) относится к термину, используемому исследователями для обозначения географической области, которую они хотят проанализировать. Оно имеет то же значение, что и термин «Область исследования».
В этом контексте мы получили ограничивающую рамку геометрии слоя точек возникновения и создали вокруг нее 50-километровый буфер (с максимальным допуском 1000 метров) для определения области интереса.
# Define the AOI
aoi = data.geometry().bounds().buffer(distance=50000, maxError=1000)
# Add the AOI to the map
outline = ee.Image().byte().paint(
featureCollection=aoi, color=1, width=3)
Map.remove_layer("Random Raster")
Map.addLayer(outline, {'palette': 'FF0000'}, "AOI")
Map.centerObject(aoi, 6)
Map
Добавление переменных среды GEE
Теперь давайте добавим в анализ переменные окружающей среды. GEE предоставляет широкий спектр наборов данных для переменных окружающей среды, таких как температура, осадки, высота, земной покров и рельеф. Эти наборы данных позволяют нам всесторонне проанализировать различные факторы, которые могут повлиять на предпочтения среды обитания Феи Питты.
Выбор переменных окружающей среды GEE в SDM должен отражать характеристики предпочтений вида в среде обитания. Для этого необходимо провести предварительное исследование и обзор литературы о предпочтениях среды обитания Феи-питты. В этом руководстве основное внимание уделяется рабочему процессу SDM с использованием GEE, поэтому некоторые подробные сведения опущены.
WorldClim V1 Bioclim : этот набор данных содержит 19 биоклиматических переменных, полученных на основе ежемесячных данных о температуре и осадках. Он охватывает период с 1960 по 1991 год и имеет разрешение 927,67 метра.
# WorldClim V1 Bioclim
bio = ee.Image("WORLDCLIM/V1/BIO")
NASA SRTM Digital Elevation 30m : этот набор данных содержит цифровые данные о высоте, полученные в ходе миссии по радиолокационной топографии шаттла (SRTM). Данные были собраны в основном примерно в 2000 году и предоставляются с разрешением примерно 30 метров (1 угловая секунда). Следующий код вычисляет слои высот, уклонов, экспозиций и отмывки на основе данных SRTM.
# NASA SRTM Digital Elevation 30m
terrain = ee.Algorithms.Terrain(ee.Image("USGS/SRTMGL1_003"))
Глобальное изменение лесного покрова (GFCC) Древесный покров, многолетний глобальный 30 м : Набор данных Vegetation Continuous Fields (VCF) от Landsat оценивает долю растительного покрова в вертикальной проекции, когда высота растительности превышает 5 метров. Этот набор данных предоставляется за четыре периода времени, примерно за 2000, 2005, 2010 и 2015 годы, с разрешением 30 метров. Здесь используются медианные значения за эти четыре периода времени.
# Global Forest Cover Change (GFCC) Tree Cover Multi-Year Global 30m
tcc = ee.ImageCollection("NASA/MEASURES/GFCC/TC/v3")
median_tcc = (
tcc.filterDate("2000-01-01", "2015-12-31")
.select(["tree_canopy_cover"], ["TCC"])
.median()
)
bio (биоклиматические переменные), terrain (топография) и median_tcc (покров деревьев) объединены в одно многоканальное изображение. Полоса elevation выбирается из terrain , а для мест, где elevation больше 0 создается watermask . Это маскирует области ниже уровня моря (например, океан) и подготавливает исследователя к всестороннему анализу различных факторов окружающей среды для AOI.
# Combine bands into a multi-band image
predictors = bio.addBands(terrain).addBands(median_tcc)
# Create a water mask
watermask = terrain.select('elevation').gt(0)
# Mask out ocean pixels and clip to the area of interest
predictors = predictors.updateMask(watermask).clip(aoi)
Когда в модель включены сильно коррелированные переменные-предикторы, могут возникнуть проблемы мультиколлинеарности. Мультиколлинеарность — это явление, которое возникает, когда между независимыми переменными в модели существуют сильные линейные связи, что приводит к нестабильности в оценке коэффициентов (весов) модели. Эта нестабильность может снизить надежность модели и затруднить прогнозирование или интерпретацию новых данных. Поэтому мы рассмотрим мультиколлинеарность и приступим к процессу выбора переменных-предикторов.
Сначала мы сгенерируем 5000 случайных точек, а затем извлечем значения переменных-предикторов одного многоканального изображения в этих точках.
# Generate 5,000 random points
data_cor = predictors.sample(scale=grain_size, numPixels=5000, geometries=True)
# Extract predictor variable values
pvals = predictors.sampleRegions(collection=data_cor, scale=grain_size)
Мы преобразуем извлеченные значения предикторов для каждой точки в DataFrame, а затем проверим первую строку.
# Converting predictor values from Earth Engine to a DataFrame
pvals_df = geemap.ee_to_df(pvals)
pvals_df.head(1)
# Displaying the columns
columns = pvals_df.columns
print(columns)
Расчет коэффициентов корреляции Спирмена между заданными переменными-предикторами и их визуализация на тепловой карте.
def plot_correlation_heatmap(dataframe, h_size=10, show_labels=False):
# Calculate Spearman correlation coefficients
correlation_matrix = dataframe.corr(method="spearman")
# Create a heatmap
plt.figure(figsize=(h_size, h_size-2))
plt.imshow(correlation_matrix, cmap='coolwarm', interpolation='nearest')
# Optionally display values on the heatmap
if show_labels:
for i in range(correlation_matrix.shape[0]):
for j in range(correlation_matrix.shape[1]):
plt.text(j, i, f"{correlation_matrix.iloc[i, j]:.2f}",
ha='center', va='center', color='white', fontsize=8)
columns = dataframe.columns.tolist()
plt.xticks(range(len(columns)), columns, rotation=90)
plt.yticks(range(len(columns)), columns)
plt.title("Variables Correlation Matrix")
plt.colorbar(label="Spearman Correlation")
plt.savefig('correlation_heatmap_plot.png')
plt.show()
# Plot the correlation heatmap of variables
plot_correlation_heatmap(pvals_df)
Коэффициент корреляции Спирмена полезен для понимания общих связей между переменными-предикторами, но не оценивает напрямую, как взаимодействуют несколько переменных, в частности, выявляя мультиколлинеарность.
Фактор инфляции дисперсии (VIF ниже) — это статистический показатель, используемый для оценки мультиколлинеарности и выбора переменных. Он указывает на степень линейной связи каждой независимой переменной с другими независимыми переменными, а высокие значения VIF могут свидетельствовать о мультиколлинеарности.
Обычно, когда значения VIF превышают 5 или 10, это предполагает, что переменная имеет сильную корреляцию с другими переменными, что потенциально ставит под угрозу стабильность и интерпретируемость модели. В этом уроке для выбора переменных использовался критерий значений VIF менее 10. Следующие 6 переменных были выбраны на основе VIF.
# Filter variables based on Variance Inflation Factor (VIF)
def filter_variables_by_vif(dataframe, threshold=10):
original_columns = dataframe.columns.tolist()
remaining_columns = original_columns[:]
while True:
vif_data = dataframe[remaining_columns]
vif_values = [
variance_inflation_factor(vif_data.values, i)
for i in range(vif_data.shape[1])
]
max_vif_index = vif_values.index(max(vif_values))
max_vif = max(vif_values)
if max_vif < threshold:
break
print(f"Removing '{remaining_columns[max_vif_index]}' with VIF {max_vif:.2f}")
del remaining_columns[max_vif_index]
filtered_data = dataframe[remaining_columns]
bands = filtered_data.columns.tolist()
print("Bands:", bands)
return filtered_data, bands
filtered_pvals_df, bands = filter_variables_by_vif(pvals_df)
# Variable Selection Based on VIF
predictors = predictors.select(bands)
# Plot the correlation heatmap of variables
plot_correlation_heatmap(filtered_pvals_df, h_size=6, show_labels=True)
Далее давайте визуализируем 6 выбранных переменных-предсказателей на карте. 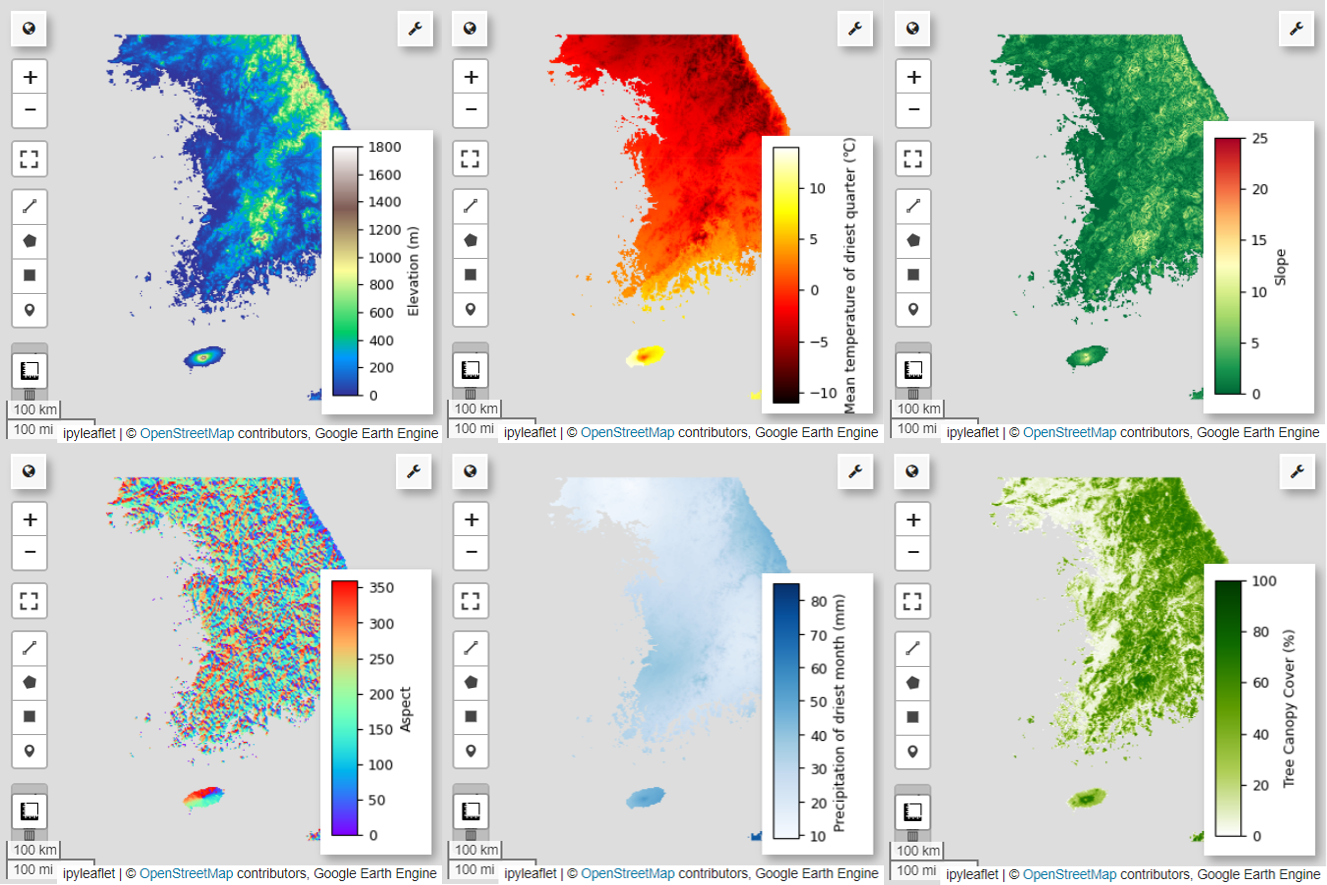
Вы можете изучить доступные палитры для визуализации карты, используя следующий код. Например, палитра terrain выглядит так. cm.plot_colormaps(width=8.0, height=0.2)
cm.plot_colormap('terrain', width=8.0, height=0.2, orientation='horizontal')
# Elevation layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['elevation'], 'min': 0, 'max': 1800, 'palette': cm.palettes.terrain}
Map.addLayer(predictors, vis_params, 'elevation')
Map.add_colorbar(vis_params, label="Elevation (m)", orientation="vertical", layer_name="elevation")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio09
min_max_val = (
predictors.select("bio09")
.multiply(0.1)
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio09 (Mean temperature of driest quarter) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"min": math.floor(min_max_val["bio09_min"]),
"max": math.ceil(min_max_val["bio09_max"]),
"palette": cm.palettes.hot,
}
Map.addLayer(predictors.select("bio09").multiply(0.1), vis_params, "bio09")
Map.add_colorbar(
vis_params,
label="Mean temperature of driest quarter (℃)",
orientation="vertical",
layer_name="bio09",
)
Map.centerObject(aoi, 6)
Map
# Slope layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['slope'], 'min': 0, 'max': 25, 'palette': cm.palettes.RdYlGn_r}
Map.addLayer(predictors, vis_params, 'slope')
Map.add_colorbar(vis_params, label="Slope", orientation="vertical", layer_name="slope")
Map.centerObject(aoi, 6)
Map
# Aspect layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['aspect'], 'min': 0, 'max': 360, 'palette': cm.palettes.rainbow}
Map.addLayer(predictors, vis_params, 'aspect')
Map.add_colorbar(vis_params, label="Aspect", orientation="vertical", layer_name="aspect")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio14
min_max_val = (
predictors.select("bio14")
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio14 (Precipitation of driest month) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["bio14"],
"min": math.floor(min_max_val["bio14_min"]),
"max": math.ceil(min_max_val["bio14_max"]),
"palette": cm.palettes.Blues,
}
Map.addLayer(predictors, vis_params, "bio14")
Map.add_colorbar(
vis_params,
label="Precipitation of driest month (mm)",
orientation="vertical",
layer_name="bio14",
)
Map.centerObject(aoi, 6)
Map
# TCC layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["TCC"],
"min": 0,
"max": 100,
"palette": ["ffffff", "afce56", "5f9c00", "0e6a00", "003800"],
}
Map.addLayer(predictors, vis_params, "TCC")
Map.add_colorbar(
vis_params, label="Tree Canopy Cover (%)", orientation="vertical", layer_name="TCC"
)
Map.centerObject(aoi, 6)
Map
Генерация данных о псевдоотсутствии
В процессе SDM к выбору входных данных по виду в основном подходят два метода:
Метод присутствия-фона : этот метод сравнивает места, где определенный вид наблюдался (присутствие), с другими местами, где этот вид не наблюдался (фон). Здесь фоновые данные не обязательно означают территории, где этот вид не существует, а скорее созданы с учетом общих условий окружающей среды исследуемой территории. Он используется для того, чтобы отличить подходящие среды, в которых виды могут существовать, от менее подходящих.
Метод присутствия-отсутствия : этот метод сравнивает места, где данный вид наблюдался (присутствие), с местами, где он окончательно не наблюдался (отсутствие). Здесь данные об отсутствии представляют собой конкретные места, где, как известно, этот вид не существует. Он не отражает общие экологические условия исследуемой территории, а скорее указывает на места, где, по оценкам, этот вид не существует.
На практике зачастую бывает сложно собрать истинные данные об отсутствии, поэтому часто используются данные о псевдоотсутствии, генерируемые искусственно. Однако важно осознавать ограничения и потенциальные ошибки этого метода, поскольку искусственно созданные точки псевдоотсутствия могут неточно отражать истинные области отсутствия.
Выбор между этими двумя методами зависит от доступности данных, целей исследования, точности и надежности модели, а также времени и ресурсов. Здесь мы будем использовать данные о встречах, собранные из GBIF, и искусственно сгенерированные данные о псевдоотсутствии для моделирования с использованием метода «Присутствие-Отсутствие».
Генерация данных о псевдоотсутствии будет осуществляться с помощью «подхода экологического профилирования», а конкретные шаги заключаются в следующем:
Классификация окружающей среды с использованием кластеризации k-средних: Алгоритм кластеризации k-средних, основанный на евклидовом расстоянии, будет использоваться для разделения пикселей в пределах исследуемой области на два кластера. Один кластер будет представлять территории с характеристиками окружающей среды, схожими со случайно выбранными 100 местами присутствия, в то время как другой кластер будет представлять территории с другими характеристиками.
Генерация данных псевдоотсутствия в разнородных кластерах: внутри второго кластера, определенного на первом этапе (который имеет характеристики окружающей среды, отличные от данных о присутствии), будут созданы случайно сгенерированные точки псевдоотсутствия. Эти точки псевдоотсутствия будут представлять собой места, где, как ожидается, этот вид не будет существовать.
# Randomly select 100 locations for occurrence
pvals = predictors.sampleRegions(
collection=data.randomColumn().sort('random').limit(100),
properties=[],
scale=grain_size
)
# Perform k-means clustering
clusterer = ee.Clusterer.wekaKMeans(
nClusters=2,
distanceFunction="Euclidean"
).train(pvals)
cl_result = predictors.cluster(clusterer)
# Get cluster ID for locations similar to occurrence
cl_id = cl_result.sampleRegions(
collection=data.randomColumn().sort('random').limit(200),
properties=[],
scale=grain_size
)
# Define non-occurrence areas in dissimilar clusters
cl_id = ee.FeatureCollection(cl_id).reduceColumns(ee.Reducer.mode(),['cluster'])
cl_id = ee.Number(cl_id.get('mode')).subtract(1).abs()
cl_mask = cl_result.select(['cluster']).eq(cl_id)
# Presence location mask
presence_mask = data.reduceToImage(properties=['random'],
reducer=ee.Reducer.first()
).reproject('EPSG:4326', None,
grain_size).mask().neq(1).selfMask()
# Masking presence locations in non-occurrence areas and clipping to AOI
area_for_pa = presence_mask.updateMask(cl_mask).clip(aoi)
# Area for Pseudo-absence
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(area_for_pa, {'palette': 'black'}, 'AreaForPA')
Map.centerObject(aoi, 6)
Map
Подбор модели и прогнозирование
Теперь мы разделим данные на обучающие и тестовые данные. Данные обучения будут использоваться для поиска оптимальных параметров путем обучения модели, а тестовые данные будут использоваться для оценки предварительно обученной модели. Важной концепцией, которую следует учитывать в этом контексте, является пространственная автокорреляция.
Пространственная автокорреляция является важным элементом SDM, связанным с законом Тоблера. Он воплощает в себе концепцию, согласно которой «все связано со всем остальным, но близкие вещи более связаны, чем отдаленные». Пространственная автокорреляция представляет собой значительную взаимосвязь между расположением видов и переменными окружающей среды. Однако если между обучающими и тестовыми данными существует пространственная автокорреляция, независимость между двумя наборами данных может быть нарушена. Это существенно влияет на оценку обобщающей способности модели.
Одним из методов решения этой проблемы является метод перекрестной проверки пространственных блоков, который включает в себя разделение данных на наборы обучающих и тестовых данных. Этот метод предполагает разделение данных на несколько блоков с использованием каждого блока независимо в качестве наборов обучающих и тестовых данных, чтобы уменьшить влияние пространственной автокорреляции. Это повышает независимость между наборами данных, позволяя более точно оценить способность модели к обобщению.
Конкретная процедура заключается в следующем:
- Создание пространственных блоков: разделите весь набор данных на пространственные блоки одинакового размера (например, 50x50 км).
- Назначение обучающего и тестового наборов. Каждый пространственный блок случайным образом назначается либо обучающему набору (70%), либо тестовому набору (30%). Это предотвращает переобучение модели данным из конкретных областей и направлено на достижение более обобщенных результатов.
- Итеративная перекрестная проверка: весь процесс повторяется n раз (например, 10 раз). На каждой итерации блоки снова случайным образом делятся на обучающий и тестовый наборы, что призвано повысить стабильность и надежность модели.
- Генерация данных псевдоотсутствия. На каждой итерации данные псевдоотсутствия генерируются случайным образом для оценки производительности модели.
Scale = 50000
grid = watermask.reduceRegions(
collection=aoi.coveringGrid(scale=Scale, proj='EPSG:4326'),
reducer=ee.Reducer.mean()).filter(ee.Filter.neq('mean', None))
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(grid, {}, "Grid for spatial block cross validation")
Map.addLayer(outline, {'palette': 'FF0000'}, "Study Area")
Map.centerObject(aoi, 6)
Map
Теперь мы можем подогнать модель. Подбор модели предполагает понимание закономерностей в данных и соответствующую корректировку параметров модели (весов и отклонений). Этот процесс позволяет модели делать более точные прогнозы при представлении новых данных. Для этой цели мы определили функцию SDM(), соответствующую модели.
Мы будем использовать алгоритм Random Forest .
def sdm(x):
seed = ee.Number(x)
# Random block division for training and validation
rand_blk = ee.FeatureCollection(grid).randomColumn(seed=seed).sort("random")
training_grid = rand_blk.filter(ee.Filter.lt("random", split)) # Grid for training
testing_grid = rand_blk.filter(ee.Filter.gte("random", split)) # Grid for testing
# Presence points
presence_points = ee.FeatureCollection(data)
presence_points = presence_points.map(lambda feature: feature.set("PresAbs", 1))
tr_presence_points = presence_points.filter(
ee.Filter.bounds(training_grid)
) # Presence points for training
te_presence_points = presence_points.filter(
ee.Filter.bounds(testing_grid)
) # Presence points for testing
# Pseudo-absence points for training
tr_pseudo_abs_points = area_for_pa.sample(
region=training_grid,
scale=grain_size,
numPixels=tr_presence_points.size().add(300),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for training
tr_pseudo_abs_points = (
tr_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(tr_presence_points.size()))
)
tr_pseudo_abs_points = tr_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
te_pseudo_abs_points = area_for_pa.sample(
region=testing_grid,
scale=grain_size,
numPixels=te_presence_points.size().add(100),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for testing
te_pseudo_abs_points = (
te_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(te_presence_points.size()))
)
te_pseudo_abs_points = te_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
# Merge training and pseudo-absence points
training_partition = tr_presence_points.merge(tr_pseudo_abs_points)
testing_partition = te_presence_points.merge(te_pseudo_abs_points)
# Extract predictor variable values at training points
train_pvals = predictors.sampleRegions(
collection=training_partition,
properties=["PresAbs"],
scale=grain_size,
geometries=True,
)
# Random Forest classifier
classifier = ee.Classifier.smileRandomForest(
numberOfTrees=500,
variablesPerSplit=None,
minLeafPopulation=10,
bagFraction=0.5,
maxNodes=None,
seed=seed,
)
# Presence probability: Habitat suitability map
classifier_pr = classifier.setOutputMode("PROBABILITY").train(
train_pvals, "PresAbs", bands
)
classified_img_pr = predictors.select(bands).classify(classifier_pr)
# Binary presence/absence map: Potential distribution map
classifier_bin = classifier.setOutputMode("CLASSIFICATION").train(
train_pvals, "PresAbs", bands
)
classified_img_bin = predictors.select(bands).classify(classifier_bin)
return [
classified_img_pr,
classified_img_bin,
training_partition,
testing_partition,
], classifier_pr
Пространственные блоки разделены на 70% для обучения модели и 30% для тестирования модели соответственно. Данные о псевдоотсутствии генерируются случайным образом в каждом наборе обучения и тестирования на каждой итерации. В результате каждое выполнение дает разные наборы данных о присутствии и псевдоотсутствии для обучения и тестирования модели.
split = 0.7
numiter = 10
# Random Seed
runif = lambda length: [random.randint(1, 1000) for _ in range(length)]
items = runif(numiter)
# Fixed seed
# items = [287, 288, 553, 226, 151, 255, 902, 267, 419, 538]
results_list = [] # Initialize SDM results list
importances_list = [] # Initialize variable importance list
for item in items:
result, trained = sdm(item)
# Accumulate SDM results into the list
results_list.extend(result)
# Accumulate variable importance into the list
importance = ee.Dictionary(trained.explain()).get('importance')
importances_list.extend(importance.getInfo().items())
# Flatten the SDM results list
results = ee.List(results_list).flatten()
Теперь мы можем визуализировать карту пригодности среды обитания и карту потенциального распространения питты Феи. В этом случае карта пригодности среды обитания создается с помощью mean() для расчета среднего значения для каждого местоположения пикселя по всем изображениям, а карта потенциального распределения создается с помощью функции mode() для определения наиболее часто встречающегося значения. в каждом пикселе на всех изображениях.
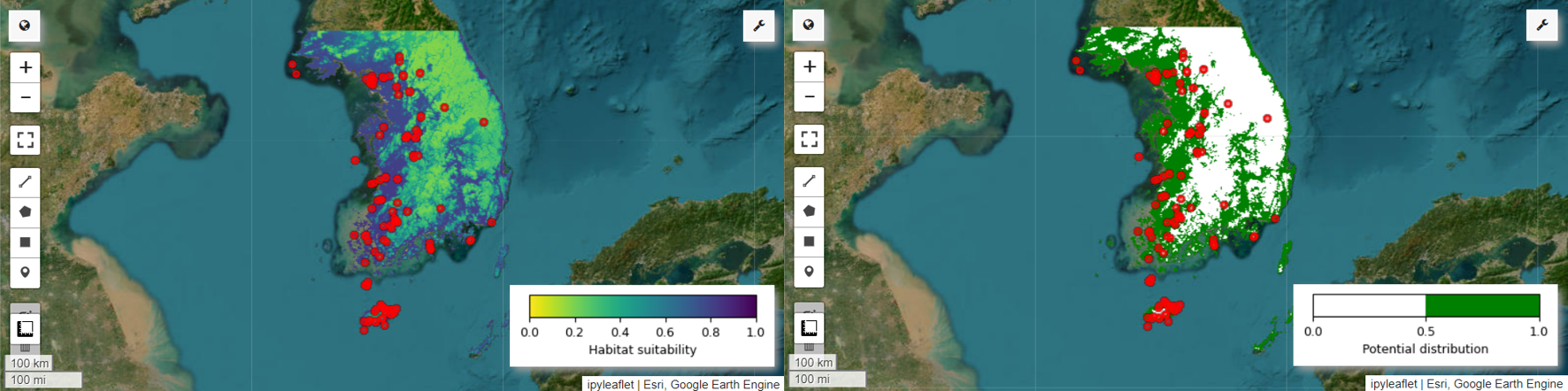
# Habitat suitability map
images = ee.List.sequence(
0, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x))
model_average = ee.ImageCollection.fromImages(images).mean()
Map = geemap.Map(layout={'height':'400px', 'width':'800px'}, basemap='Esri.WorldImagery')
vis_params = {
'min': 0,
'max': 1,
'palette': cm.palettes.viridis_r}
Map.addLayer(model_average, vis_params, 'Habitat suitability')
Map.add_colorbar(vis_params, label="Habitat suitability",
orientation="horizontal",
layer_name="Habitat suitability")
Map.addLayer(data, {'color':'red'}, 'Presence')
Map.centerObject(aoi, 6)
Map
# Potential distribution map
images2 = ee.List.sequence(1, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x)
)
distribution_map = ee.ImageCollection.fromImages(images2).mode()
Map = geemap.Map(
layout={"height": "400px", "width": "800px"}, basemap="Esri.WorldImagery"
)
vis_params = {"min": 0, "max": 1, "palette": ["white", "green"]}
Map.addLayer(distribution_map, vis_params, "Potential distribution")
Map.addLayer(data, {"color": "red"}, "Presence")
Map.add_colorbar(
vis_params,
label="Potential distribution",
discrete=True,
orientation="horizontal",
layer_name="Potential distribution",
)
Map.centerObject(data.geometry(), 6)
Map
Оценка важности и точности переменных
Случайный лес ( ee.Classifier.smileRandomForest ) — это один из методов ансамблевого обучения, который основан на построении нескольких деревьев решений для прогнозирования. Каждое дерево решений независимо обучается на основе различных подмножеств данных, а их результаты агрегируются для обеспечения более точных и стабильных прогнозов.
Важность переменной — это мера, которая оценивает влияние каждой переменной на прогнозы в модели случайного леса. Мы будем использовать ранее определенный importances_list для расчета и печати средней важности переменной.
def plot_variable_importance(importances_list):
# Extract each variable importance value into a list
variables = [item[0] for item in importances_list]
importances = [item[1] for item in importances_list]
# Calculate the average importance for each variable
average_importances = {}
for variable in set(variables):
indices = [i for i, var in enumerate(variables) if var == variable]
average_importance = np.mean([importances[i] for i in indices])
average_importances[variable] = average_importance
# Sort the importances in descending order of importance
sorted_importances = sorted(average_importances.items(),
key=lambda x: x[1], reverse=False)
variables = [item[0] for item in sorted_importances]
avg_importances = [item[1] for item in sorted_importances]
# Adjust the graph size
plt.figure(figsize=(8, 4))
# Plot the average importance as a horizontal bar chart
plt.barh(variables, avg_importances)
plt.xlabel('Importance')
plt.ylabel('Variables')
plt.title('Average Variable Importance')
# Display values above the bars
for i, v in enumerate(avg_importances):
plt.text(v + 0.02, i, f"{v:.2f}", va='center')
# Adjust the x-axis range
plt.xlim(0, max(avg_importances) + 5) # Adjust to the desired range
plt.tight_layout()
plt.savefig('variable_importance.png')
plt.show()
plot_variable_importance(importances_list)
Используя наборы тестовых данных, мы рассчитываем AUC-ROC и AUC-PR для каждого запуска. Затем мы вычисляем средние значения AUC-ROC и AUC-PR за n итераций.
AUC-ROC представляет собой площадь под кривой графика «Чувствительность (отзыв) против 1-специфичности», иллюстрируя взаимосвязь между чувствительностью и специфичностью при изменении порога. Специфичность основана на всех наблюдаемых ненаблюдениях. Таким образом, AUC-ROC охватывает все квадранты матрицы путаницы.
AUC-PR представляет собой область под кривой графика «Точность и полнота (чувствительность)», показывающую взаимосвязь между точностью и полнотой при изменении порогового значения. Точность основана на всех прогнозируемых событиях. Следовательно, AUC-PR не включает истинные отрицательные результаты (TN).
def print_pres_abs_sizes(TestingDatasets, numiter):
# Check and print the sizes of presence and pseudo-absence coordinates
def get_pres_abs_size(x):
fc = ee.FeatureCollection(TestingDatasets.get(x))
presence_size = fc.filter(ee.Filter.eq("PresAbs", 1)).size()
pseudo_absence_size = fc.filter(ee.Filter.eq("PresAbs", 0)).size()
return ee.List([presence_size, pseudo_absence_size])
sizes_info = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(get_pres_abs_size)
.getInfo()
)
for i, sizes in enumerate(sizes_info):
presence_size = sizes[0]
pseudo_absence_size = sizes[1]
print(
f"Iteration {i + 1}: Presence Size = {presence_size}, Pseudo-absence Size = {pseudo_absence_size}"
)
# Extracting the Testing Datasets
testing_datasets = ee.List.sequence(
3, ee.Number(numiter).multiply(4).subtract(1), 4
).map(lambda x: results.get(x))
print_pres_abs_sizes(testing_datasets, numiter)
def get_acc(hsm, t_data, grain_size):
pr_prob_vals = hsm.sampleRegions(
collection=t_data, properties=["PresAbs"], scale=grain_size
)
seq = ee.List.sequence(start=0, end=1, count=25) # Divide 0 to 1 into 25 intervals
def calculate_metrics(cutoff):
# Each element of the seq list is passed as cutoff(threshold value)
# Observed present = TP + FN
pres = pr_prob_vals.filterMetadata("PresAbs", "equals", 1)
# TP (True Positive)
tp = ee.Number(
pres.filterMetadata("classification", "greater_than", cutoff).size()
)
# TPR (True Positive Rate) = Recall = Sensitivity = TP / (TP + FN) = TP / Observed present
tpr = tp.divide(pres.size())
# Observed absent = FP + TN
abs = pr_prob_vals.filterMetadata("PresAbs", "equals", 0)
# FN (False Negative)
fn = ee.Number(
pres.filterMetadata("classification", "less_than", cutoff).size()
)
# TNR (True Negative Rate) = Specificity = TN / (FP + TN) = TN / Observed absent
tn = ee.Number(abs.filterMetadata("classification", "less_than", cutoff).size())
tnr = tn.divide(abs.size())
# FP (False Positive)
fp = ee.Number(
abs.filterMetadata("classification", "greater_than", cutoff).size()
)
# FPR (False Positive Rate) = FP / (FP + TN) = FP / Observed absent
fpr = fp.divide(abs.size())
# Precision = TP / (TP + FP) = TP / Predicted present
precision = tp.divide(tp.add(fp))
# SUMSS = SUM of Sensitivity and Specificity
sumss = tpr.add(tnr)
return ee.Feature(
None,
{
"cutoff": cutoff,
"TP": tp,
"TN": tn,
"FP": fp,
"FN": fn,
"TPR": tpr,
"TNR": tnr,
"FPR": fpr,
"Precision": precision,
"SUMSS": sumss,
},
)
return ee.FeatureCollection(seq.map(calculate_metrics))
def calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter):
# Calculate AUC-ROC and AUC-PR
def calculate_auc_metrics(x):
hsm = ee.Image(images.get(x))
t_data = ee.FeatureCollection(testing_datasets.get(x))
acc = get_acc(hsm, t_data, grain_size)
# Calculate AUC-ROC
x = ee.Array(acc.aggregate_array("FPR"))
y = ee.Array(acc.aggregate_array("TPR"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_roc = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
# Calculate AUC-PR
x = ee.Array(acc.aggregate_array("TPR"))
y = ee.Array(acc.aggregate_array("Precision"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_pr = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
return (auc_roc, auc_pr)
auc_metrics = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(calculate_auc_metrics)
.getInfo()
)
# Print AUC-ROC and AUC-PR for each iteration
df = pd.DataFrame(auc_metrics, columns=["AUC-ROC", "AUC-PR"])
df.index = [f"Iteration {i + 1}" for i in range(len(df))]
df.to_csv("auc_metrics.csv", index_label="Iteration")
print(df)
# Calculate mean and standard deviation of AUC-ROC and AUC-PR
mean_auc_roc, std_auc_roc = df["AUC-ROC"].mean(), df["AUC-ROC"].std()
mean_auc_pr, std_auc_pr = df["AUC-PR"].mean(), df["AUC-PR"].std()
print(f"Mean AUC-ROC = {mean_auc_roc:.4f} ± {std_auc_roc:.4f}")
print(f"Mean AUC-PR = {mean_auc_pr:.4f} ± {std_auc_pr:.4f}")
%%time
# Calculate AUC-ROC and AUC-PR
calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter)
В этом руководстве представлен практический пример использования Google Earth Engine (GEE) для моделирования распространения видов (SDM). Важным выводом является универсальность и гибкость GEE в области SDM. Использование мощных возможностей обработки геопространственных данных Earth Engine открывает безграничные возможности для исследователей и защитников природы для понимания и сохранения биоразнообразия на нашей планете. Применяя знания и навыки, полученные из этого руководства, люди могут исследовать и внести свой вклад в эту увлекательную область экологических исследований.

 Запустить в Google Colab
Запустить в Google Colab Посмотреть исходный код на GitHub
Посмотреть исходный код на GitHub