Trong hướng dẫn này, phương pháp lập mô hình phân bố loài bằng Google Earth Engine sẽ được giới thiệu. Chúng tôi sẽ cung cấp thông tin tổng quan ngắn gọn về Mô hình phân bố loài, sau đó là quy trình dự đoán và phân tích môi trường sống của một loài chim có nguy cơ tuyệt chủng, được gọi là Pitta tiên (tên khoa học: Pitta nympha).
Chạy tôi trước
Chạy ô sau để khởi chạy API. Kết quả sẽ có hướng dẫn về cách cấp cho sổ tay này quyền truy cập vào Earth Engine bằng tài khoản của bạn.
import ee
# Trigger the authentication flow.
ee.Authenticate()
# Initialize the library.
ee.Initialize(project='my-project')
Tổng quan ngắn gọn về mô hình phân bố loài
Hãy cùng tìm hiểu về mô hình phân bố loài, những lợi ích khi sử dụng Google Earth Engine để xử lý mô hình, dữ liệu cần thiết cho mô hình và cách cấu trúc quy trình làm việc.
Mô hình phân bố loài là gì?
Mô hình hoá sự phân bố loài (SDM bên dưới) là phương pháp phổ biến nhất được dùng để ước tính sự phân bố địa lý thực tế hoặc tiềm năng của một loài. Việc này bao gồm việc mô tả các điều kiện môi trường phù hợp với một loài cụ thể, sau đó xác định nơi phân bố các điều kiện phù hợp này theo địa lý.
SDM đã trở thành một thành phần quan trọng trong kế hoạch bảo tồn những năm gần đây và nhiều kỹ thuật mô hình hoá đã được phát triển cho mục đích này. Việc triển khai SDM trong Google Earth Engine (GEE bên dưới) giúp dễ dàng truy cập vào dữ liệu môi trường quy mô lớn, cùng với khả năng tính toán mạnh mẽ và hỗ trợ các thuật toán học máy, cho phép mô hình hoá nhanh chóng.
Dữ liệu bắt buộc đối với SDM
SDM thường sử dụng mối quan hệ giữa các bản ghi về sự xuất hiện của loài đã biết và các biến môi trường để xác định các điều kiện mà một quần thể có thể duy trì. Nói cách khác, bạn cần có 2 loại dữ liệu đầu vào cho mô hình:
- Bản ghi về sự xuất hiện của các loài đã biết
- Nhiều biến môi trường
Những dữ liệu này được đưa vào các thuật toán để xác định điều kiện môi trường liên quan đến sự hiện diện của các loài.
Quy trình làm việc của SDM bằng GEE
Quy trình làm việc cho SDM bằng GEE như sau:
- Thu thập và tiền xử lý dữ liệu về sự xuất hiện của loài
- Định nghĩa về Khu vực quan tâm
- Bổ sung các biến môi trường GEE
- Tạo dữ liệu vắng mặt giả
- Điều chỉnh mô hình và dự đoán
- Đánh giá độ chính xác và tầm quan trọng của biến
Dự đoán và phân tích môi trường sống bằng GEE
Chim Pitta tiên (Pitta nympha) sẽ được dùng làm nghiên cứu điển hình để minh hoạ việc áp dụng SDM dựa trên GEE. Mặc dù loài cụ thể này đã được chọn cho một ví dụ, nhưng các nhà nghiên cứu có thể áp dụng phương pháp này cho bất kỳ loài mục tiêu nào mà họ quan tâm bằng cách sửa đổi nhẹ mã nguồn được cung cấp.
Chim Pitta tiên là loài chim di cư hiếm gặp vào mùa hè và mùa đông ở Hàn Quốc. Phạm vi phân bố của loài chim này đang mở rộng do tình trạng ấm lên gần đây của khí hậu trên Bán đảo Triều Tiên. Đây là loài quý hiếm, động vật hoang dã thuộc nhóm II có nguy cơ tuyệt chủng, Di tích tự nhiên số 204, được đánh giá là Tuyệt chủng ở khu vực (RE) trong Sách đỏ quốc gia và Dễ bị tổn thương (VU) theo các danh mục của IUCN.
Việc tiến hành SDM cho kế hoạch bảo tồn loài Pitta nympha có vẻ khá có giá trị. Bây giờ, hãy tiến hành dự đoán và phân tích môi trường sống thông qua GEE.
Trước tiên, các thư viện Python sẽ được nhập.Câu lệnh import sẽ đưa toàn bộ nội dung của một mô-đun vào, trong khi câu lệnh from import cho phép nhập các đối tượng cụ thể từ một mô-đun.
# Import libraries
import geemap
import geemap.colormaps as cm
import pandas as pd, geopandas as gpd
import numpy as np, matplotlib.pyplot as plt
import os, requests, math, random
from ipyleaflet import TileLayer
from statsmodels.stats.outliers_influence import variance_inflation_factor
Thu thập và xử lý trước dữ liệu về sự xuất hiện của loài
Bây giờ, hãy thu thập dữ liệu về sự xuất hiện của chim Pitta tiên. Ngay cả khi hiện không có quyền truy cập vào dữ liệu về sự xuất hiện của loài mà bạn quan tâm, bạn vẫn có thể thu thập dữ liệu quan sát về các loài cụ thể thông qua GBIF API. GBIF API là một giao diện cho phép truy cập vào dữ liệu phân bố loài do GBIF cung cấp, cho phép người dùng tìm kiếm, lọc và tải dữ liệu xuống, cũng như thu thập nhiều thông tin liên quan đến loài.
Trong mã bên dưới, biến species_name được gán tên khoa học của loài (ví dụ: Pitta nympha cho Pitta tiên), và biến country_code được chỉ định mã quốc gia (ví dụ: KR cho Hàn Quốc). Biến base_url lưu trữ địa chỉ của API GBIF. params là một từ điển chứa các tham số sẽ được dùng trong yêu cầu API:
scientificName: Đặt tên khoa học của loài cần tìm kiếm.country: Giới hạn phạm vi tìm kiếm ở một quốc gia cụ thể.hasCoordinate: Đảm bảo chỉ tìm kiếm dữ liệu có toạ độ (true).basisOfRecord: Chỉ chọn bản ghi quan sát của con người (HUMAN_OBSERVATION).limit: Đặt số lượng kết quả tối đa được trả về thành 10000.
def get_gbif_species_data(species_name, country_code):
"""
Retrieves observational data for a specific species using the GBIF API and returns it as a pandas DataFrame.
Parameters:
species_name (str): The scientific name of the species to query.
country_code (str): The country code of the where the observation data will be queried.
Returns:
pd.DataFrame: A pandas DataFrame containing the observational data.
"""
base_url = "https://api.gbif.org/v1/occurrence/search"
params = {
"scientificName": species_name,
"country": country_code,
"hasCoordinate": "true",
"basisOfRecord": "HUMAN_OBSERVATION",
"limit": 10000,
}
try:
response = requests.get(base_url, params=params)
response.raise_for_status() # Raises an exception for a response error.
data = response.json()
occurrences = data.get("results", [])
if occurrences: # If data is present
df = pd.json_normalize(occurrences)
return df
else:
print("No data found for the given species and country code.")
return pd.DataFrame() # Returns an empty DataFrame
except requests.RequestException as e:
print(f"Request failed: {e}")
return pd.DataFrame() # Returns an empty DataFrame in case of an exception
Bằng cách sử dụng các tham số đã đặt trước đó, chúng ta sẽ truy vấn API GBIF để lấy các bản ghi quan sát về loài Chim pitta tiên (Pitta nympha) và tải kết quả vào DataFrame để kiểm tra hàng đầu tiên. DataFrame là một cấu trúc dữ liệu để xử lý dữ liệu ở định dạng bảng, bao gồm các hàng và cột. Nếu cần, bạn có thể lưu DataFrame dưới dạng tệp CSV và đọc lại.
# Retrieve Fairy Pitta data
df = get_gbif_species_data("Pitta nympha", "KR")
"""
# Save DataFrame to CSV and read back in.
df.to_csv("pitta_nympha_data.csv", index=False)
df = pd.read_csv("pitta_nympha_data.csv")
"""
df.head(1) # Display the first row of the DataFrame
Tiếp theo, chúng ta chuyển đổi DataFrame thành GeoDataFrame có chứa một cột cho thông tin địa lý (geometry) và kiểm tra hàng đầu tiên. Bạn có thể lưu GeoDataFrame dưới dạng tệp GeoPackage (*.gpkg) và đọc lại.
# Convert DataFrame to GeoDataFrame
gdf = gpd.GeoDataFrame(
df,
geometry=gpd.points_from_xy(df.decimalLongitude,
df.decimalLatitude),
crs="EPSG:4326"
)[["species", "year", "month", "geometry"]]
"""
# Convert GeoDataFrame to GeoPackage (requires pycrs module)
%pip install -U -q pycrs
gdf.to_file("pitta_nympha_data.gpkg", driver="GPKG")
gdf = gpd.read_file("pitta_nympha_data.gpkg")
"""
gdf.head(1) # Display the first row of the GeoDataFrame
Lần này, chúng ta đã tạo một hàm để trực quan hoá việc phân phối dữ liệu theo năm và tháng từ GeoDataFrame, đồng thời hiển thị dữ liệu đó dưới dạng biểu đồ. Sau đó, bạn có thể lưu biểu đồ này dưới dạng tệp hình ảnh. Việc sử dụng bản đồ nhiệt giúp chúng ta nhanh chóng nắm bắt tần suất xuất hiện của các loài theo năm và tháng, mang đến hình ảnh trực quan về những thay đổi và mẫu hình theo thời gian trong dữ liệu. Điều này cho phép xác định các mẫu tạm thời và biến động theo mùa trong dữ liệu về sự xuất hiện của loài, cũng như nhanh chóng phát hiện các giá trị ngoại lệ hoặc vấn đề về chất lượng trong dữ liệu.
# Yearly and monthly data distribution heatmap
def plot_heatmap(gdf, h_size=8):
statistics = gdf.groupby(["month", "year"]).size().unstack(fill_value=0)
# Heatmap
plt.figure(figsize=(h_size, h_size - 6))
heatmap = plt.imshow(
statistics.values, cmap="YlOrBr", origin="upper", aspect="auto"
)
# Display values above each pixel
for i in range(len(statistics.index)):
for j in range(len(statistics.columns)):
plt.text(
j, i, statistics.values[i, j], ha="center", va="center", color="black"
)
plt.colorbar(heatmap, label="Count")
plt.title("Monthly Species Count by Year")
plt.xlabel("Year")
plt.ylabel("Month")
plt.xticks(range(len(statistics.columns)), statistics.columns)
plt.yticks(range(len(statistics.index)), statistics.index)
plt.tight_layout()
plt.savefig("heatmap_plot.png")
plt.show()
plot_heatmap(gdf)
Dữ liệu từ năm 1995 rất thưa thớt, có những khoảng trống đáng kể so với các năm khác, đồng thời tháng 8 và tháng 9 cũng có ít mẫu và thể hiện các đặc điểm theo mùa khác so với các khoảng thời gian khác. Việc loại trừ dữ liệu này có thể góp phần cải thiện độ ổn định và khả năng dự đoán của mô hình.
Tuy nhiên, bạn cần lưu ý rằng việc loại trừ dữ liệu có thể nâng cao khả năng tổng quát hoá của mô hình, nhưng cũng có thể dẫn đến việc mất thông tin có giá trị liên quan đến mục tiêu nghiên cứu. Do đó, bạn nên cân nhắc kỹ lưỡng trước khi đưa ra những quyết định như vậy.
# Filtering data by year and month
filtered_gdf = gdf[
(~gdf['year'].eq(1995)) &
(~gdf['month'].between(8, 9))
]
Giờ đây, GeoDataFrame đã được lọc sẽ được chuyển đổi thành một đối tượng Google Earth Engine.
# Convert GeoDataFrame to Earth Engine object
data_raw = geemap.geopandas_to_ee(filtered_gdf)
Tiếp theo, chúng ta sẽ xác định kích thước pixel raster của kết quả SDM là độ phân giải 1 km.
# Spatial resolution setting (meters)
grain_size = 1000
Khi có nhiều điểm xuất hiện trong cùng một pixel raster có độ phân giải 1 km, thì có nhiều khả năng chúng có cùng điều kiện môi trường tại cùng một vị trí địa lý. Việc sử dụng trực tiếp dữ liệu như vậy trong quá trình phân tích có thể đưa ra kết quả thiên vị.
Nói cách khác, chúng ta cần hạn chế tác động tiềm ẩn của sai lệch do lấy mẫu theo địa lý. Để đạt được điều này, chúng tôi sẽ chỉ giữ lại một vị trí trong mỗi ô vuông 1 km và xoá tất cả các vị trí khác, cho phép mô hình phản ánh các điều kiện môi trường một cách khách quan hơn.
def remove_duplicates(data, grain_size):
# Select one occurrence record per pixel at the chosen spatial resolution
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
rand_point_vals = random_raster.sampleRegions(
collection=ee.FeatureCollection(data), geometries=True
)
return rand_point_vals.distinct("random")
data = remove_duplicates(data_raw, grain_size)
# Before selection and after selection
print("Original data size:", data_raw.size().getInfo())
print("Final data size:", data.size().getInfo())
Hình ảnh trực quan so sánh độ lệch lấy mẫu theo địa lý trước khi xử lý trước (màu xanh dương) và sau khi xử lý trước (màu đỏ) được minh hoạ bên dưới. Để so sánh dễ dàng hơn, bản đồ đã được đặt ở khu vực có nhiều toạ độ xuất hiện của loài chim Pitta nympha trong Vườn quốc gia Hallasan.
# Visualization of geographic sampling bias before (blue) and after (red) preprocessing
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
# Add the random raster layer
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
Map.addLayer(
random_raster,
{"min": 0, "max": 1, "palette": ["black", "white"], "opacity": 0.5},
"Random Raster",
)
# Add the original data layer in blue
Map.addLayer(data_raw, {"color": "blue"}, "Original data")
# Add the final data layer in red
Map.addLayer(data, {"color": "red"}, "Final data")
# Set the center of the map to the coordinates
Map.setCenter(126.712, 33.516, 14)
Map
Định nghĩa về Khu vực quan tâm
Xác định Khu vực quan tâm (AOI bên dưới) là thuật ngữ mà các nhà nghiên cứu sử dụng để biểu thị khu vực địa lý mà họ muốn phân tích. Nó có ý nghĩa tương tự như thuật ngữ Khu vực nghiên cứu.
Trong bối cảnh này, chúng tôi đã lấy được hộp giới hạn của hình học lớp điểm xuất hiện và tạo vùng đệm 50 km xung quanh (với dung sai tối đa là 1.000 mét) để xác định AOI.
# Define the AOI
aoi = data.geometry().bounds().buffer(distance=50000, maxError=1000)
# Add the AOI to the map
outline = ee.Image().byte().paint(
featureCollection=aoi, color=1, width=3)
Map.remove_layer("Random Raster")
Map.addLayer(outline, {'palette': 'FF0000'}, "AOI")
Map.centerObject(aoi, 6)
Map
Bổ sung các biến môi trường GEE
Bây giờ, hãy thêm các biến môi trường vào bản phân tích. GEE cung cấp nhiều loại tập dữ liệu cho các biến môi trường như nhiệt độ, lượng mưa, độ cao, độ che phủ đất và địa hình. Những bộ dữ liệu này giúp chúng tôi phân tích toàn diện nhiều yếu tố có thể ảnh hưởng đến lựa chọn môi trường sống của loài chim Pitta tiên.
Việc lựa chọn các biến môi trường GEE trong SDM phải phản ánh đặc điểm về môi trường sống ưa thích của loài. Để làm được điều này, bạn nên tiến hành nghiên cứu trước và xem xét tài liệu về các đặc điểm môi trường sống ưa thích của chim Pitta tiên. Hướng dẫn này chủ yếu tập trung vào quy trình làm việc của SDM bằng GEE, vì vậy, một số thông tin chi tiết chuyên sâu sẽ bị bỏ qua.
WorldClim V1 Bioclim: Tập dữ liệu này cung cấp 19 biến số sinh khí hậu được lấy từ dữ liệu về nhiệt độ và lượng mưa hằng tháng. Tập dữ liệu này bao gồm khoảng thời gian từ năm 1960 đến năm 1991 và có độ phân giải là 927,67 mét.
# WorldClim V1 Bioclim
bio = ee.Image("WORLDCLIM/V1/BIO")
Dữ liệu độ cao kỹ thuật số 30 m của NASA SRTM: Tập dữ liệu này chứa dữ liệu độ cao kỹ thuật số từ Dự án lập bản đồ địa hình bằng radar trên tàu con thoi (SRTM). Dữ liệu này chủ yếu được thu thập vào khoảng năm 2000 và được cung cấp ở độ phân giải khoảng 30 mét (1 giây cung). Đoạn mã sau đây tính toán các lớp độ cao, độ dốc, hướng dốc và bóng đồi từ dữ liệu SRTM.
# NASA SRTM Digital Elevation 30m
terrain = ee.Algorithms.Terrain(ee.Image("USGS/SRTMGL1_003"))
Thay đổi về độ che phủ rừng trên toàn cầu (GFCC) Độ che phủ cây xanh trên toàn cầu trong nhiều năm ở độ phân giải 30 m: Tập dữ liệu Các trường thảm thực vật liên tục (VCF) của Landsat ước tính tỷ lệ độ che phủ thảm thực vật được chiếu theo chiều dọc khi chiều cao của thảm thực vật lớn hơn 5 mét. Tập dữ liệu này được cung cấp cho 4 khoảng thời gian tập trung vào các năm 2000, 2005, 2010 và 2015, với độ phân giải 30 mét. Ở đây, các giá trị trung vị của 4 khoảng thời gian này được sử dụng.
# Global Forest Cover Change (GFCC) Tree Cover Multi-Year Global 30m
tcc = ee.ImageCollection("NASA/MEASURES/GFCC/TC/v3")
median_tcc = (
tcc.filterDate("2000-01-01", "2015-12-31")
.select(["tree_canopy_cover"], ["TCC"])
.median()
)
bio (Các biến số khí hậu sinh học), terrain (địa hình) và median_tcc (độ che phủ của tán cây) được kết hợp thành một hình ảnh nhiều dải tần. Dải elevation được chọn từ terrain và watermask được tạo cho những vị trí mà elevation lớn hơn 0. Điều này sẽ che các khu vực dưới mực nước biển (ví dụ: đại dương) và giúp nhà nghiên cứu chuẩn bị phân tích toàn diện nhiều yếu tố môi trường cho AOI.
# Combine bands into a multi-band image
predictors = bio.addBands(terrain).addBands(median_tcc)
# Create a water mask
watermask = terrain.select('elevation').gt(0)
# Mask out ocean pixels and clip to the area of interest
predictors = predictors.updateMask(watermask).clip(aoi)
Khi các biến dự đoán có mối tương quan cao được đưa vào cùng một mô hình, vấn đề đa cộng tuyến có thể xảy ra. Đa cộng tuyến là một hiện tượng xảy ra khi có mối quan hệ tuyến tính chặt chẽ giữa các biến độc lập trong một mô hình, dẫn đến sự không ổn định trong việc ước tính các hệ số (trọng số) của mô hình. Sự không ổn định này có thể làm giảm độ tin cậy của mô hình và gây khó khăn cho việc dự đoán hoặc diễn giải dữ liệu mới. Do đó, chúng ta sẽ xem xét hiện tượng tương quan đa biến và tiến hành quy trình chọn biến dự đoán.
Trước tiên, chúng ta sẽ tạo 5.000 điểm ngẫu nhiên,sau đó trích xuất các giá trị biến dự đoán của hình ảnh nhiều dải tần số tại những điểm đó.
# Generate 5,000 random points
data_cor = predictors.sample(scale=grain_size, numPixels=5000, geometries=True)
# Extract predictor variable values
pvals = predictors.sampleRegions(collection=data_cor, scale=grain_size)
Chúng ta sẽ chuyển đổi các giá trị dự đoán đã trích xuất cho từng điểm thành DataFrame, sau đó kiểm tra hàng đầu tiên.
# Converting predictor values from Earth Engine to a DataFrame
pvals_df = geemap.ee_to_df(pvals)
pvals_df.head(1)
# Displaying the columns
columns = pvals_df.columns
print(columns)
Tính toán hệ số tương quan Spearman giữa các biến dự đoán đã cho và trực quan hoá chúng trong một bản đồ nhiệt.
def plot_correlation_heatmap(dataframe, h_size=10, show_labels=False):
# Calculate Spearman correlation coefficients
correlation_matrix = dataframe.corr(method="spearman")
# Create a heatmap
plt.figure(figsize=(h_size, h_size-2))
plt.imshow(correlation_matrix, cmap='coolwarm', interpolation='nearest')
# Optionally display values on the heatmap
if show_labels:
for i in range(correlation_matrix.shape[0]):
for j in range(correlation_matrix.shape[1]):
plt.text(j, i, f"{correlation_matrix.iloc[i, j]:.2f}",
ha='center', va='center', color='white', fontsize=8)
columns = dataframe.columns.tolist()
plt.xticks(range(len(columns)), columns, rotation=90)
plt.yticks(range(len(columns)), columns)
plt.title("Variables Correlation Matrix")
plt.colorbar(label="Spearman Correlation")
plt.savefig('correlation_heatmap_plot.png')
plt.show()
# Plot the correlation heatmap of variables
plot_correlation_heatmap(pvals_df)
Hệ số tương quan Spearman rất hữu ích để hiểu được các mối liên kết chung giữa các biến dự đoán nhưng không đánh giá trực tiếp cách nhiều biến tương tác, cụ thể là phát hiện hiện tượng đa cộng tuyến.
Hệ số phóng đại phương sai (VIF bên dưới) là một chỉ số thống kê được dùng để đánh giá hiện tượng đa cộng tuyến và hướng dẫn lựa chọn biến. Hệ số này cho biết mức độ mối quan hệ tuyến tính của từng biến độc lập với các biến độc lập khác và giá trị VIF cao có thể là bằng chứng về hiện tượng đa cộng tuyến.
Thông thường, khi giá trị VIF vượt quá 5 hoặc 10, điều này cho thấy biến có mối tương quan chặt chẽ với các biến khác, có thể ảnh hưởng đến tính ổn định và khả năng diễn giải của mô hình. Trong hướng dẫn này, tiêu chí về giá trị VIF nhỏ hơn 10 đã được dùng để chọn biến. 6 biến sau đây được chọn dựa trên VIF.
# Filter variables based on Variance Inflation Factor (VIF)
def filter_variables_by_vif(dataframe, threshold=10):
original_columns = dataframe.columns.tolist()
remaining_columns = original_columns[:]
while True:
vif_data = dataframe[remaining_columns]
vif_values = [
variance_inflation_factor(vif_data.values, i)
for i in range(vif_data.shape[1])
]
max_vif_index = vif_values.index(max(vif_values))
max_vif = max(vif_values)
if max_vif < threshold:
break
print(f"Removing '{remaining_columns[max_vif_index]}' with VIF {max_vif:.2f}")
del remaining_columns[max_vif_index]
filtered_data = dataframe[remaining_columns]
bands = filtered_data.columns.tolist()
print("Bands:", bands)
return filtered_data, bands
filtered_pvals_df, bands = filter_variables_by_vif(pvals_df)
# Variable Selection Based on VIF
predictors = predictors.select(bands)
# Plot the correlation heatmap of variables
plot_correlation_heatmap(filtered_pvals_df, h_size=6, show_labels=True)
Tiếp theo, hãy trực quan hoá 6 biến dự đoán đã chọn trên bản đồ.
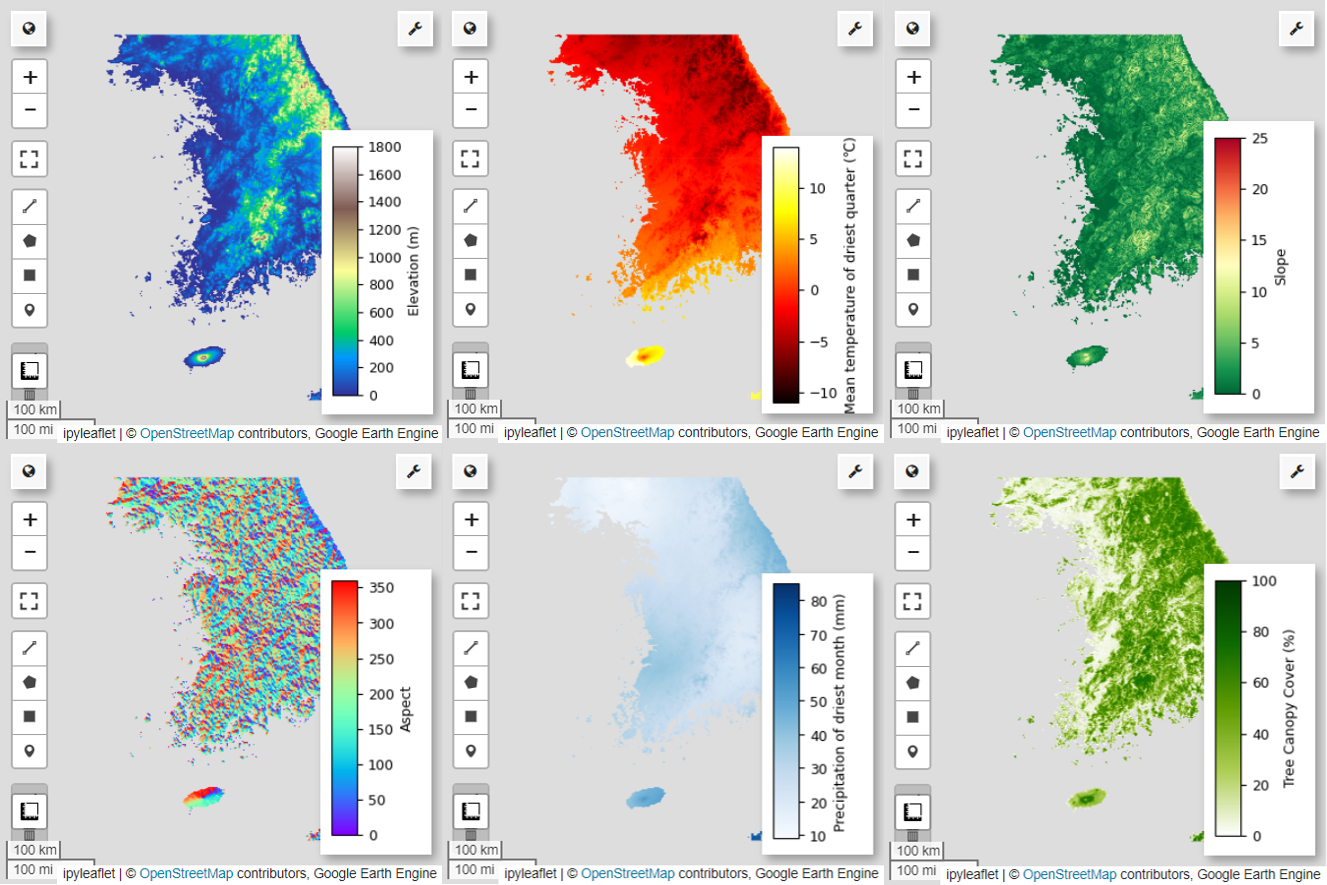
Bạn có thể khám phá các bảng màu có sẵn để trực quan hoá bản đồ bằng mã sau. Ví dụ: bảng terrain có dạng như sau.
cm.plot_colormaps(width=8.0, height=0.2)
cm.plot_colormap('terrain', width=8.0, height=0.2, orientation='horizontal')
# Elevation layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['elevation'], 'min': 0, 'max': 1800, 'palette': cm.palettes.terrain}
Map.addLayer(predictors, vis_params, 'elevation')
Map.add_colorbar(vis_params, label="Elevation (m)", orientation="vertical", layer_name="elevation")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio09
min_max_val = (
predictors.select("bio09")
.multiply(0.1)
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio09 (Mean temperature of driest quarter) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"min": math.floor(min_max_val["bio09_min"]),
"max": math.ceil(min_max_val["bio09_max"]),
"palette": cm.palettes.hot,
}
Map.addLayer(predictors.select("bio09").multiply(0.1), vis_params, "bio09")
Map.add_colorbar(
vis_params,
label="Mean temperature of driest quarter (℃)",
orientation="vertical",
layer_name="bio09",
)
Map.centerObject(aoi, 6)
Map
# Slope layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['slope'], 'min': 0, 'max': 25, 'palette': cm.palettes.RdYlGn_r}
Map.addLayer(predictors, vis_params, 'slope')
Map.add_colorbar(vis_params, label="Slope", orientation="vertical", layer_name="slope")
Map.centerObject(aoi, 6)
Map
# Aspect layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['aspect'], 'min': 0, 'max': 360, 'palette': cm.palettes.rainbow}
Map.addLayer(predictors, vis_params, 'aspect')
Map.add_colorbar(vis_params, label="Aspect", orientation="vertical", layer_name="aspect")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio14
min_max_val = (
predictors.select("bio14")
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio14 (Precipitation of driest month) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["bio14"],
"min": math.floor(min_max_val["bio14_min"]),
"max": math.ceil(min_max_val["bio14_max"]),
"palette": cm.palettes.Blues,
}
Map.addLayer(predictors, vis_params, "bio14")
Map.add_colorbar(
vis_params,
label="Precipitation of driest month (mm)",
orientation="vertical",
layer_name="bio14",
)
Map.centerObject(aoi, 6)
Map
# TCC layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["TCC"],
"min": 0,
"max": 100,
"palette": ["ffffff", "afce56", "5f9c00", "0e6a00", "003800"],
}
Map.addLayer(predictors, vis_params, "TCC")
Map.add_colorbar(
vis_params, label="Tree Canopy Cover (%)", orientation="vertical", layer_name="TCC"
)
Map.centerObject(aoi, 6)
Map
Tạo dữ liệu vắng mặt giả
Trong quy trình SDM, việc lựa chọn dữ liệu đầu vào cho một loài chủ yếu được thực hiện bằng hai phương pháp:
Phương pháp Hiện diện – Nền: Phương pháp này so sánh những vị trí mà một loài cụ thể đã được quan sát (hiện diện) với những vị trí khác mà loài đó chưa được quan sát (nền). Ở đây, dữ liệu nền không nhất thiết có nghĩa là những khu vực không có loài này, mà được thiết lập để phản ánh các điều kiện môi trường tổng thể của khu vực nghiên cứu. Chỉ số này được dùng để phân biệt những môi trường phù hợp với loài này với những môi trường ít phù hợp hơn.
Phương pháp có mặt và không có mặt: Phương pháp này so sánh những vị trí mà loài này đã được quan sát thấy (có mặt) với những vị trí mà loài này chắc chắn không được quan sát thấy (không có mặt). Ở đây, dữ liệu về sự vắng mặt đại diện cho những vị trí cụ thể mà loài này không tồn tại. Nó không phản ánh điều kiện môi trường tổng thể của khu vực nghiên cứu mà chỉ ra những vị trí mà loài này được ước tính là không tồn tại.
Trên thực tế, thường rất khó thu thập dữ liệu vắng mặt thực tế, vì vậy, dữ liệu vắng mặt giả được tạo ra một cách nhân tạo thường được sử dụng. Tuy nhiên, điều quan trọng là phải thừa nhận những hạn chế và lỗi tiềm ẩn của phương pháp này, vì các điểm giả định không có sự xuất hiện được tạo nhân tạo có thể không phản ánh chính xác các khu vực thực sự không có sự xuất hiện.
Việc lựa chọn giữa hai phương pháp này phụ thuộc vào tính sẵn có của dữ liệu, mục tiêu nghiên cứu, độ chính xác và độ tin cậy của mô hình, cũng như thời gian và nguồn lực. Ở đây, chúng ta sẽ sử dụng dữ liệu về sự xuất hiện được thu thập từ GBIF và dữ liệu giả về sự vắng mặt được tạo nhân tạo để mô hình hoá bằng phương pháp "Có mặt – Vắng mặt".
Việc tạo dữ liệu vắng mặt giả sẽ được thực hiện thông qua "phương pháp lập hồ sơ môi trường" và các bước cụ thể như sau:
Phân loại môi trường bằng phương pháp phân cụm k-means: Thuật toán phân cụm k-means, dựa trên khoảng cách Euclid, sẽ được dùng để chia các pixel trong khu vực nghiên cứu thành 2 cụm. Một cụm sẽ đại diện cho những khu vực có đặc điểm môi trường tương tự như 100 vị trí có sự hiện diện được chọn ngẫu nhiên, trong khi cụm còn lại sẽ đại diện cho những khu vực có đặc điểm khác.
Tạo dữ liệu giả định về sự vắng mặt trong các cụm không giống nhau: Trong cụm thứ hai được xác định ở bước đầu tiên (có các đặc điểm môi trường khác với dữ liệu về sự hiện diện), các điểm giả định về sự vắng mặt được tạo ngẫu nhiên sẽ được tạo. Những điểm giả không có mặt này sẽ đại diện cho những vị trí mà loài này không được dự kiến sẽ tồn tại.
# Randomly select 100 locations for occurrence
pvals = predictors.sampleRegions(
collection=data.randomColumn().sort('random').limit(100),
properties=[],
scale=grain_size
)
# Perform k-means clustering
clusterer = ee.Clusterer.wekaKMeans(
nClusters=2,
distanceFunction="Euclidean"
).train(pvals)
cl_result = predictors.cluster(clusterer)
# Get cluster ID for locations similar to occurrence
cl_id = cl_result.sampleRegions(
collection=data.randomColumn().sort('random').limit(200),
properties=[],
scale=grain_size
)
# Define non-occurrence areas in dissimilar clusters
cl_id = ee.FeatureCollection(cl_id).reduceColumns(ee.Reducer.mode(),['cluster'])
cl_id = ee.Number(cl_id.get('mode')).subtract(1).abs()
cl_mask = cl_result.select(['cluster']).eq(cl_id)
# Presence location mask
presence_mask = data.reduceToImage(properties=['random'],
reducer=ee.Reducer.first()
).reproject('EPSG:4326', None,
grain_size).mask().neq(1).selfMask()
# Masking presence locations in non-occurrence areas and clipping to AOI
area_for_pa = presence_mask.updateMask(cl_mask).clip(aoi)
# Area for Pseudo-absence
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(area_for_pa, {'palette': 'black'}, 'AreaForPA')
Map.centerObject(aoi, 6)
Map
Điều chỉnh mô hình và dự đoán
Bây giờ, chúng ta sẽ chia dữ liệu thành dữ liệu huấn luyện và dữ liệu kiểm thử. Dữ liệu huấn luyện sẽ được dùng để tìm các thông số tối ưu bằng cách huấn luyện mô hình, trong khi dữ liệu kiểm thử sẽ được dùng để đánh giá mô hình đã được huấn luyện trước đó. Một khái niệm quan trọng cần xem xét trong bối cảnh này là tự tương quan không gian.
Tự tương quan không gian là một yếu tố thiết yếu trong SDM, liên quan đến quy luật của Tobler. Nó thể hiện khái niệm "mọi thứ đều liên quan đến mọi thứ khác, nhưng những thứ gần nhau có liên quan nhiều hơn những thứ ở xa". Tự tương quan không gian thể hiện mối quan hệ đáng kể giữa vị trí của các loài và các biến môi trường. Tuy nhiên, nếu có tương quan không gian giữa dữ liệu huấn luyện và dữ liệu kiểm thử, thì tính độc lập giữa hai tập dữ liệu có thể bị ảnh hưởng. Điều này ảnh hưởng đáng kể đến việc đánh giá khả năng tổng quát hoá của mô hình.
Một phương pháp để giải quyết vấn đề này là kỹ thuật xác thực chéo khối không gian, bao gồm việc chia dữ liệu thành các tập dữ liệu huấn luyện và kiểm thử. Kỹ thuật này bao gồm việc chia dữ liệu thành nhiều khối, sử dụng độc lập từng khối làm tập dữ liệu huấn luyện và kiểm thử để giảm tác động của tự tương quan không gian. Điều này giúp tăng cường tính độc lập giữa các tập dữ liệu, cho phép đánh giá chính xác hơn khả năng tổng quát hoá của mô hình.
Quy trình cụ thể như sau:
- Tạo các khối không gian: Chia toàn bộ tập dữ liệu thành các khối không gian có kích thước bằng nhau (ví dụ: 50x50 km).
- Phân công tập hợp huấn luyện và tập hợp kiểm thử: Mỗi khối không gian được chỉ định ngẫu nhiên cho tập hợp huấn luyện (70%) hoặc tập hợp kiểm thử (30%). Điều này giúp mô hình không bị khớp quá mức với dữ liệu từ các khu vực cụ thể và nhằm mục đích đạt được kết quả tổng quát hơn.
- Xác thực chéo lặp đi lặp lại: Toàn bộ quy trình được lặp lại n lần (ví dụ: 10 lần). Trong mỗi lần lặp lại, các khối sẽ được chia ngẫu nhiên thành các tập hợp huấn luyện và kiểm thử một lần nữa, nhằm mục đích cải thiện tính ổn định và độ tin cậy của mô hình.
- Tạo dữ liệu giả định về sự vắng mặt: Trong mỗi lần lặp lại, dữ liệu giả định về sự vắng mặt được tạo ngẫu nhiên để đánh giá hiệu suất của mô hình.
Scale = 50000
grid = watermask.reduceRegions(
collection=aoi.coveringGrid(scale=Scale, proj='EPSG:4326'),
reducer=ee.Reducer.mean()).filter(ee.Filter.neq('mean', None))
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(grid, {}, "Grid for spatial block cross validation")
Map.addLayer(outline, {'palette': 'FF0000'}, "Study Area")
Map.centerObject(aoi, 6)
Map
Giờ đây, chúng ta có thể điều chỉnh mô hình. Việc điều chỉnh một mô hình bao gồm việc tìm hiểu các mẫu trong dữ liệu và điều chỉnh các tham số (trọng số và độ lệch) của mô hình cho phù hợp. Quy trình này cho phép mô hình đưa ra thông tin dự đoán chính xác hơn khi có dữ liệu mới. Vì mục đích này, chúng ta đã xác định một hàm có tên là SDM() để điều chỉnh mô hình.
Chúng ta sẽ sử dụng thuật toán Rừng ngẫu nhiên.
def sdm(x):
seed = ee.Number(x)
# Random block division for training and validation
rand_blk = ee.FeatureCollection(grid).randomColumn(seed=seed).sort("random")
training_grid = rand_blk.filter(ee.Filter.lt("random", split)) # Grid for training
testing_grid = rand_blk.filter(ee.Filter.gte("random", split)) # Grid for testing
# Presence points
presence_points = ee.FeatureCollection(data)
presence_points = presence_points.map(lambda feature: feature.set("PresAbs", 1))
tr_presence_points = presence_points.filter(
ee.Filter.bounds(training_grid)
) # Presence points for training
te_presence_points = presence_points.filter(
ee.Filter.bounds(testing_grid)
) # Presence points for testing
# Pseudo-absence points for training
tr_pseudo_abs_points = area_for_pa.sample(
region=training_grid,
scale=grain_size,
numPixels=tr_presence_points.size().add(300),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for training
tr_pseudo_abs_points = (
tr_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(tr_presence_points.size()))
)
tr_pseudo_abs_points = tr_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
te_pseudo_abs_points = area_for_pa.sample(
region=testing_grid,
scale=grain_size,
numPixels=te_presence_points.size().add(100),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for testing
te_pseudo_abs_points = (
te_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(te_presence_points.size()))
)
te_pseudo_abs_points = te_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
# Merge training and pseudo-absence points
training_partition = tr_presence_points.merge(tr_pseudo_abs_points)
testing_partition = te_presence_points.merge(te_pseudo_abs_points)
# Extract predictor variable values at training points
train_pvals = predictors.sampleRegions(
collection=training_partition,
properties=["PresAbs"],
scale=grain_size,
geometries=True,
)
# Random Forest classifier
classifier = ee.Classifier.smileRandomForest(
numberOfTrees=500,
variablesPerSplit=None,
minLeafPopulation=10,
bagFraction=0.5,
maxNodes=None,
seed=seed,
)
# Presence probability: Habitat suitability map
classifier_pr = classifier.setOutputMode("PROBABILITY").train(
train_pvals, "PresAbs", bands
)
classified_img_pr = predictors.select(bands).classify(classifier_pr)
# Binary presence/absence map: Potential distribution map
classifier_bin = classifier.setOutputMode("CLASSIFICATION").train(
train_pvals, "PresAbs", bands
)
classified_img_bin = predictors.select(bands).classify(classifier_bin)
return [
classified_img_pr,
classified_img_bin,
training_partition,
testing_partition,
], classifier_pr
Các khối không gian được chia thành 70% để huấn luyện mô hình và 30% để kiểm thử mô hình. Dữ liệu vắng mặt giả được tạo ngẫu nhiên trong mỗi tập hợp huấn luyện và kiểm thử ở mọi lần lặp. Do đó, mỗi lần thực thi sẽ tạo ra các tập dữ liệu về sự hiện diện và sự vắng mặt giả tạo khác nhau để huấn luyện và kiểm thử mô hình.
split = 0.7
numiter = 10
# Random Seed
runif = lambda length: [random.randint(1, 1000) for _ in range(length)]
items = runif(numiter)
# Fixed seed
# items = [287, 288, 553, 226, 151, 255, 902, 267, 419, 538]
results_list = [] # Initialize SDM results list
importances_list = [] # Initialize variable importance list
for item in items:
result, trained = sdm(item)
# Accumulate SDM results into the list
results_list.extend(result)
# Accumulate variable importance into the list
importance = ee.Dictionary(trained.explain()).get('importance')
importances_list.extend(importance.getInfo().items())
# Flatten the SDM results list
results = ee.List(results_list).flatten()
Giờ đây, chúng ta có thể hình dung bản đồ mức độ phù hợp của môi trường sống và bản đồ phân bố tiềm năng cho loài Pitta nympha. Trong trường hợp này, bản đồ mức độ phù hợp của môi trường sống được tạo bằng cách sử dụng hàm mean() để tính giá trị trung bình cho từng vị trí pixel trên tất cả các hình ảnh, còn bản đồ phân bố tiềm năng được tạo bằng cách sử dụng hàm mode() để xác định giá trị xuất hiện thường xuyên nhất tại từng vị trí pixel trên tất cả các hình ảnh.
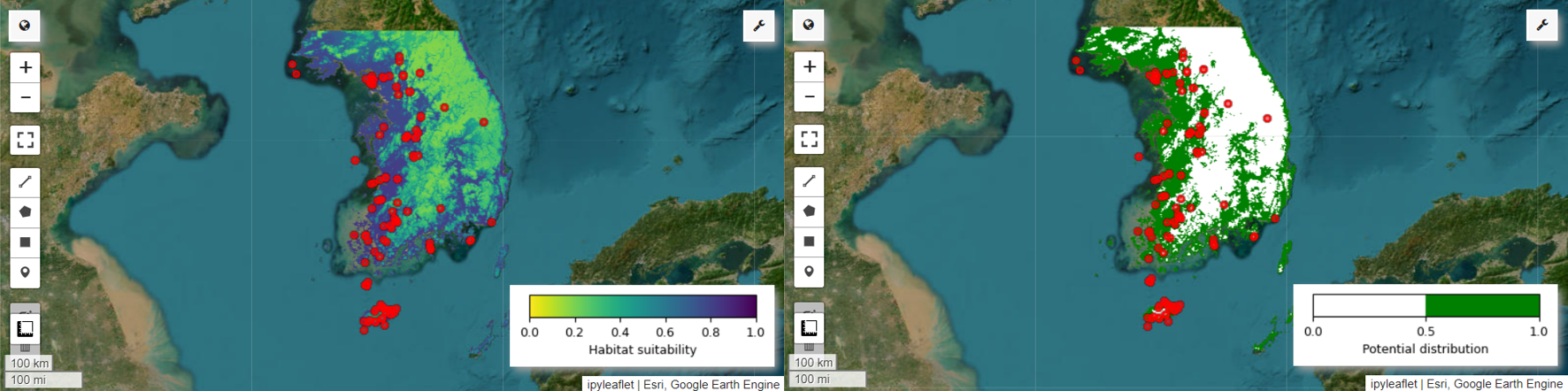
# Habitat suitability map
images = ee.List.sequence(
0, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x))
model_average = ee.ImageCollection.fromImages(images).mean()
Map = geemap.Map(layout={'height':'400px', 'width':'800px'}, basemap='Esri.WorldImagery')
vis_params = {
'min': 0,
'max': 1,
'palette': cm.palettes.viridis_r}
Map.addLayer(model_average, vis_params, 'Habitat suitability')
Map.add_colorbar(vis_params, label="Habitat suitability",
orientation="horizontal",
layer_name="Habitat suitability")
Map.addLayer(data, {'color':'red'}, 'Presence')
Map.centerObject(aoi, 6)
Map
# Potential distribution map
images2 = ee.List.sequence(1, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x)
)
distribution_map = ee.ImageCollection.fromImages(images2).mode()
Map = geemap.Map(
layout={"height": "400px", "width": "800px"}, basemap="Esri.WorldImagery"
)
vis_params = {"min": 0, "max": 1, "palette": ["white", "green"]}
Map.addLayer(distribution_map, vis_params, "Potential distribution")
Map.addLayer(data, {"color": "red"}, "Presence")
Map.add_colorbar(
vis_params,
label="Potential distribution",
discrete=True,
orientation="horizontal",
layer_name="Potential distribution",
)
Map.centerObject(data.geometry(), 6)
Map
Đánh giá độ chính xác và tầm quan trọng của biến
Rừng ngẫu nhiên (ee.Classifier.smileRandomForest) là một trong những phương pháp học tập kết hợp, hoạt động bằng cách xây dựng nhiều cây quyết định để đưa ra dự đoán. Mỗi cây quyết định học độc lập từ các tập hợp con khác nhau của dữ liệu và kết quả của chúng được tổng hợp để đưa ra dự đoán chính xác và ổn định hơn.
Mức độ quan trọng của biến là một chỉ số đánh giá mức độ tác động của từng biến đối với các dự đoán trong mô hình Rừng ngẫu nhiên. Chúng ta sẽ sử dụng importances_list đã xác định trước đó để tính toán và in tầm quan trọng trung bình của biến.
def plot_variable_importance(importances_list):
# Extract each variable importance value into a list
variables = [item[0] for item in importances_list]
importances = [item[1] for item in importances_list]
# Calculate the average importance for each variable
average_importances = {}
for variable in set(variables):
indices = [i for i, var in enumerate(variables) if var == variable]
average_importance = np.mean([importances[i] for i in indices])
average_importances[variable] = average_importance
# Sort the importances in descending order of importance
sorted_importances = sorted(average_importances.items(),
key=lambda x: x[1], reverse=False)
variables = [item[0] for item in sorted_importances]
avg_importances = [item[1] for item in sorted_importances]
# Adjust the graph size
plt.figure(figsize=(8, 4))
# Plot the average importance as a horizontal bar chart
plt.barh(variables, avg_importances)
plt.xlabel('Importance')
plt.ylabel('Variables')
plt.title('Average Variable Importance')
# Display values above the bars
for i, v in enumerate(avg_importances):
plt.text(v + 0.02, i, f"{v:.2f}", va='center')
# Adjust the x-axis range
plt.xlim(0, max(avg_importances) + 5) # Adjust to the desired range
plt.tight_layout()
plt.savefig('variable_importance.png')
plt.show()
plot_variable_importance(importances_list)
Khi sử dụng Tập dữ liệu kiểm thử, chúng tôi tính toán AUC-ROC và AUC-PR cho mỗi lần chạy. Sau đó, chúng tôi tính toán AUC-ROC và AUC-PR trung bình qua n lần lặp.
AUC-ROC biểu thị diện tích dưới đường cong của biểu đồ "Độ nhạy (Khả năng thu hồi) so với 1-Độ đặc hiệu", minh hoạ mối quan hệ giữa độ nhạy và độ đặc hiệu khi ngưỡng thay đổi. Độ đặc hiệu dựa trên tất cả các trường hợp không xảy ra được quan sát. Do đó, AUC-ROC bao gồm tất cả các góc phần tư của ma trận nhầm lẫn.
AUC-PR biểu thị diện tích dưới đường cong của biểu đồ "Độ chính xác so với độ thu hồi (độ nhạy)", cho thấy mối quan hệ giữa độ chính xác và độ thu hồi khi ngưỡng thay đổi. Độ chính xác dựa trên tất cả các lần xuất hiện được dự đoán. Do đó, AUC-PR không bao gồm các giá trị âm tính thực (TN).
def print_pres_abs_sizes(TestingDatasets, numiter):
# Check and print the sizes of presence and pseudo-absence coordinates
def get_pres_abs_size(x):
fc = ee.FeatureCollection(TestingDatasets.get(x))
presence_size = fc.filter(ee.Filter.eq("PresAbs", 1)).size()
pseudo_absence_size = fc.filter(ee.Filter.eq("PresAbs", 0)).size()
return ee.List([presence_size, pseudo_absence_size])
sizes_info = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(get_pres_abs_size)
.getInfo()
)
for i, sizes in enumerate(sizes_info):
presence_size = sizes[0]
pseudo_absence_size = sizes[1]
print(
f"Iteration {i + 1}: Presence Size = {presence_size}, Pseudo-absence Size = {pseudo_absence_size}"
)
# Extracting the Testing Datasets
testing_datasets = ee.List.sequence(
3, ee.Number(numiter).multiply(4).subtract(1), 4
).map(lambda x: results.get(x))
print_pres_abs_sizes(testing_datasets, numiter)
def get_acc(hsm, t_data, grain_size):
pr_prob_vals = hsm.sampleRegions(
collection=t_data, properties=["PresAbs"], scale=grain_size
)
seq = ee.List.sequence(start=0, end=1, count=25) # Divide 0 to 1 into 25 intervals
def calculate_metrics(cutoff):
# Each element of the seq list is passed as cutoff(threshold value)
# Observed present = TP + FN
pres = pr_prob_vals.filterMetadata("PresAbs", "equals", 1)
# TP (True Positive)
tp = ee.Number(
pres.filterMetadata("classification", "greater_than", cutoff).size()
)
# TPR (True Positive Rate) = Recall = Sensitivity = TP / (TP + FN) = TP / Observed present
tpr = tp.divide(pres.size())
# Observed absent = FP + TN
abs = pr_prob_vals.filterMetadata("PresAbs", "equals", 0)
# FN (False Negative)
fn = ee.Number(
pres.filterMetadata("classification", "less_than", cutoff).size()
)
# TNR (True Negative Rate) = Specificity = TN / (FP + TN) = TN / Observed absent
tn = ee.Number(abs.filterMetadata("classification", "less_than", cutoff).size())
tnr = tn.divide(abs.size())
# FP (False Positive)
fp = ee.Number(
abs.filterMetadata("classification", "greater_than", cutoff).size()
)
# FPR (False Positive Rate) = FP / (FP + TN) = FP / Observed absent
fpr = fp.divide(abs.size())
# Precision = TP / (TP + FP) = TP / Predicted present
precision = tp.divide(tp.add(fp))
# SUMSS = SUM of Sensitivity and Specificity
sumss = tpr.add(tnr)
return ee.Feature(
None,
{
"cutoff": cutoff,
"TP": tp,
"TN": tn,
"FP": fp,
"FN": fn,
"TPR": tpr,
"TNR": tnr,
"FPR": fpr,
"Precision": precision,
"SUMSS": sumss,
},
)
return ee.FeatureCollection(seq.map(calculate_metrics))
def calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter):
# Calculate AUC-ROC and AUC-PR
def calculate_auc_metrics(x):
hsm = ee.Image(images.get(x))
t_data = ee.FeatureCollection(testing_datasets.get(x))
acc = get_acc(hsm, t_data, grain_size)
# Calculate AUC-ROC
x = ee.Array(acc.aggregate_array("FPR"))
y = ee.Array(acc.aggregate_array("TPR"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_roc = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
# Calculate AUC-PR
x = ee.Array(acc.aggregate_array("TPR"))
y = ee.Array(acc.aggregate_array("Precision"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_pr = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
return (auc_roc, auc_pr)
auc_metrics = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(calculate_auc_metrics)
.getInfo()
)
# Print AUC-ROC and AUC-PR for each iteration
df = pd.DataFrame(auc_metrics, columns=["AUC-ROC", "AUC-PR"])
df.index = [f"Iteration {i + 1}" for i in range(len(df))]
df.to_csv("auc_metrics.csv", index_label="Iteration")
print(df)
# Calculate mean and standard deviation of AUC-ROC and AUC-PR
mean_auc_roc, std_auc_roc = df["AUC-ROC"].mean(), df["AUC-ROC"].std()
mean_auc_pr, std_auc_pr = df["AUC-PR"].mean(), df["AUC-PR"].std()
print(f"Mean AUC-ROC = {mean_auc_roc:.4f} ± {std_auc_roc:.4f}")
print(f"Mean AUC-PR = {mean_auc_pr:.4f} ± {std_auc_pr:.4f}")
%%time
# Calculate AUC-ROC and AUC-PR
calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter)
Hướng dẫn này đã cung cấp một ví dụ thực tế về cách sử dụng Google Earth Engine (GEE) cho Mô hình phân bố loài (SDM). Một điểm quan trọng cần lưu ý là tính linh hoạt và đa năng của GEE trong lĩnh vực SDM. Việc khai thác các chức năng xử lý dữ liệu không gian địa lý mạnh mẽ của Earth Engine mở ra vô vàn khả năng cho các nhà nghiên cứu và nhà bảo tồn để hiểu và bảo tồn sự đa dạng sinh học trên hành tinh của chúng ta. Bằng cách áp dụng kiến thức và kỹ năng thu được từ hướng dẫn này, các cá nhân có thể khám phá và đóng góp cho lĩnh vực nghiên cứu sinh thái hấp dẫn này.
 Chạy trong Google Colab
Chạy trong Google Colab
 Xem nguồn trên GitHub
Xem nguồn trên GitHub