في هذا البرنامج التعليمي، سيتم تقديم منهجية "نمذجة توزيع الأنواع" باستخدام Google Earth Engine. سيتم تقديم نظرة عامة موجزة حول "نمذجة توزيع الأنواع"، يليها عملية التنبؤ بموطن نوع من الطيور المهددة بالانقراض والمعروف باسم "البيتا الجنية" (الاسم العلمي: Pitta nympha) وتحليله.
تشغيل هذا الإجراء أولاً
نفِّذ الخلية التالية لتهيئة واجهة برمجة التطبيقات. سيتضمّن الناتج تعليمات حول كيفية منح دفتر الملاحظات هذا إذن الوصول إلى Earth Engine باستخدام حسابك.
import ee
# Trigger the authentication flow.
ee.Authenticate()
# Initialize the library.
ee.Initialize(project='my-project')
لمحة موجزة عن "نمذجة توزيع الأنواع"
لنستكشف ما هي نماذج توزيع الأنواع، ومزايا استخدام Google Earth Engine لمعالجتها، والبيانات المطلوبة للنماذج، وكيفية تنظيم سير العمل.
ما هي نمذجة توزيع الأنواع؟
نموذجة توزيع الأنواع (SDM أدناه) هي المنهجية الأكثر شيوعًا المستخدمة لتقدير التوزيع الجغرافي الفعلي أو المحتمل للأنواع. ويشمل ذلك تحديد الظروف البيئية المناسبة لنوع معيّن من الكائنات الحية، ثم تحديد الأماكن التي تتوفّر فيها هذه الظروف المناسبة جغرافيًا.
لقد برزت نماذج توزيع الأنواع كعنصر أساسي في تخطيط الحفاظ على البيئة في السنوات الأخيرة، وتم تطوير تقنيات نمذجة مختلفة لهذا الغرض. يتيح تنفيذ نموذج التوزيع المكاني للأنواع (SDM) في Google Earth Engine (المشار إليه أدناه باسم GEE) الوصول بسهولة إلى البيانات البيئية الواسعة النطاق، بالإضافة إلى إمكانات الحوسبة الفعّالة ودعم خوارزميات تعلُّم الآلة، ما يسمح بإعداد النماذج بسرعة.
البيانات المطلوبة لنموذج تحديد المصدر بالاستناد إلى البيانات
تستفيد نماذج توزيع الأنواع عادةً من العلاقة بين سجلّات رصد الأنواع المعروفة والمتغيّرات البيئية لتحديد الظروف التي يمكن أن يستمر فيها أحد التجمّعات. بعبارة أخرى، يجب توفير نوعَين من بيانات إدخال النموذج:
- سجلات رصد الأنواع المعروفة
- متغيرات بيئية مختلفة
يتم إدخال هذه البيانات في الخوارزميات لتحديد الظروف البيئية المرتبطة بوجود أنواع معيّنة من الكائنات الحية.
سير عمل SDM باستخدام GEE
في ما يلي سير عمل "نموذج بيانات الموقع المكاني" باستخدام "محرك Google Earth":
- جمع بيانات رصد الأنواع ومعالجتها مسبقًا
- تعريف المنطقة محط الاهتمام
- إضافة متغيرات بيئية في "محرك بحث Google Earth"
- إنشاء بيانات الغياب الزائف
- ملاءمة النموذج والتوقّع
- أهمية المتغيرات وتقييم الدقة
توقّع الموائل وتحليلها باستخدام "محرك بحث Google Earth"
سيتم استخدام طائر البيتا الجنية (Pitta nympha) كدراسة حالة لتوضيح تطبيق نموذج توزيع الأنواع المستند إلى GEE. على الرغم من أنّه تم اختيار هذا النوع تحديدًا في أحد الأمثلة، يمكن للباحثين تطبيق المنهجية على أي نوع مستهدف من الأنواع التي تهمّهم مع إجراء تعديلات طفيفة على رمز المصدر المقدَّم.
طائر البيتا الجنية هو طائر مهاجر نادر في الصيف وفي أثناء المرور في كوريا الجنوبية، ويتوسّع نطاق توزيعه بسبب ارتفاع درجة الحرارة مؤخرًا في شبه الجزيرة الكورية. يُصنّف هذا النوع على أنّه من الأنواع النادرة، والحيوانات البرية المهدّدة بالانقراض من الفئة الثانية، والمعالم الطبيعية رقم 204، ويُقيّم على أنّه منقرض إقليميًا (RE) في القائمة الحمراء الوطنية، ومعرّض للخطر (VU) وفقًا لتصنيفات الاتحاد الدولي لحفظ الطبيعة.
يبدو أنّ إجراء نمذجة توزيع الأنواع (SDM) لتخطيط الحفاظ على طائر "البيتا الجنية" له قيمة كبيرة. لننتقل الآن إلى توقّع الموائل وتحليلها من خلال GEE.
أولاً، يتم استيراد مكتبات Python.يجلب الأمر import المحتوى الكامل لوحدة، بينما يسمح الأمر from import باستيراد عناصر معيّنة من وحدة.
# Import libraries
import geemap
import geemap.colormaps as cm
import pandas as pd, geopandas as gpd
import numpy as np, matplotlib.pyplot as plt
import os, requests, math, random
from ipyleaflet import TileLayer
from statsmodels.stats.outliers_influence import variance_inflation_factor
جمع بيانات رصد الأنواع ومعالجتها مسبقًا
لنجمع الآن بيانات حول ظهور طائر البيتا الخيالي. حتى إذا لم يكن بإمكانك حاليًا الوصول إلى بيانات حدوث الأنواع التي تهمّك، يمكنك الحصول على بيانات الملاحظات حول أنواع معيّنة من خلال GBIF API. واجهة برمجة التطبيقات GBIF API هي واجهة تتيح الوصول إلى بيانات توزيع الأنواع التي يوفّرها GBIF، ما يتيح للمستخدمين البحث عن البيانات وفلترتها وتنزيلها، بالإضافة إلى الحصول على معلومات مختلفة متعلقة بالأنواع.
في الرمز أدناه، يتم تعيين الاسم العلمي للنوع (مثلاًspecies_name Pitta nympha لـ Fairy pitta)، ويتم تعيين رمز البلد للمتغير country_code (مثلاً KR لكوريا الجنوبية). يخزّن المتغيّر base_url عنوان GBIF API. params هو قاموس يحتوي على مَعلمات سيتم استخدامها في طلب البيانات من واجهة برمجة التطبيقات:
scientificName: تضبط هذه السمة الاسم العلمي للنوع المطلوب البحث عنه.-
country: تحصر عملية البحث ببلد معيّن. hasCoordinate: يضمن البحث عن البيانات التي تتضمّن إحداثيات فقط (صحيح).basisOfRecord: يختار هذا الخيار السجلات التي تم جمعها من خلال الملاحظة البشرية فقط (HUMAN_OBSERVATION).limit: يضبط الحد الأقصى لعدد النتائج المعروضة على 10,000.
def get_gbif_species_data(species_name, country_code):
"""
Retrieves observational data for a specific species using the GBIF API and returns it as a pandas DataFrame.
Parameters:
species_name (str): The scientific name of the species to query.
country_code (str): The country code of the where the observation data will be queried.
Returns:
pd.DataFrame: A pandas DataFrame containing the observational data.
"""
base_url = "https://api.gbif.org/v1/occurrence/search"
params = {
"scientificName": species_name,
"country": country_code,
"hasCoordinate": "true",
"basisOfRecord": "HUMAN_OBSERVATION",
"limit": 10000,
}
try:
response = requests.get(base_url, params=params)
response.raise_for_status() # Raises an exception for a response error.
data = response.json()
occurrences = data.get("results", [])
if occurrences: # If data is present
df = pd.json_normalize(occurrences)
return df
else:
print("No data found for the given species and country code.")
return pd.DataFrame() # Returns an empty DataFrame
except requests.RequestException as e:
print(f"Request failed: {e}")
return pd.DataFrame() # Returns an empty DataFrame in case of an exception
باستخدام المَعلمات التي تم ضبطها سابقًا، نطلب من GBIF API الحصول على سجلّات الملاحظات الخاصة بطائر البيتا الجنية (Pitta nympha)، ونحمّل النتائج في DataFrame للتحقّق من الصف الأول. DataFrame هو بنية بيانات للتعامل مع البيانات المنسَّقة في جدول، ويتألف من صفوف وأعمدة. إذا لزم الأمر، يمكن حفظ DataFrame كملف CSV وإعادة قراءته.
# Retrieve Fairy Pitta data
df = get_gbif_species_data("Pitta nympha", "KR")
"""
# Save DataFrame to CSV and read back in.
df.to_csv("pitta_nympha_data.csv", index=False)
df = pd.read_csv("pitta_nympha_data.csv")
"""
df.head(1) # Display the first row of the DataFrame
بعد ذلك، نحول DataFrame إلى GeoDataFrame يتضمّن عمودًا للمعلومات الجغرافية (geometry) ونتحقّق من الصف الأول. يمكن حفظ GeoDataFrame كملف GeoPackage (*.gpkg) وإعادة قراءته.
# Convert DataFrame to GeoDataFrame
gdf = gpd.GeoDataFrame(
df,
geometry=gpd.points_from_xy(df.decimalLongitude,
df.decimalLatitude),
crs="EPSG:4326"
)[["species", "year", "month", "geometry"]]
"""
# Convert GeoDataFrame to GeoPackage (requires pycrs module)
%pip install -U -q pycrs
gdf.to_file("pitta_nympha_data.gpkg", driver="GPKG")
gdf = gpd.read_file("pitta_nympha_data.gpkg")
"""
gdf.head(1) # Display the first row of the GeoDataFrame
في هذه المرة، أنشأنا دالة لتصوّر توزيع البيانات حسب السنة والشهر من GeoDataFrame وعرضها كرسم بياني، ويمكن بعد ذلك حفظها كملف صورة. يتيح لنا استخدام خريطة الحرارة فهم معدّل تكرار ظهور الأنواع بسرعة حسب السنة والشهر، ما يوفّر تصورًا بديهيًا للتغييرات والأنماط الزمنية ضمن البيانات. يتيح ذلك تحديد الأنماط الزمنية والاختلافات الموسمية في بيانات ظهور الأنواع، بالإضافة إلى الرصد السريع للقيم الشاذة أو مشاكل الجودة في البيانات.
# Yearly and monthly data distribution heatmap
def plot_heatmap(gdf, h_size=8):
statistics = gdf.groupby(["month", "year"]).size().unstack(fill_value=0)
# Heatmap
plt.figure(figsize=(h_size, h_size - 6))
heatmap = plt.imshow(
statistics.values, cmap="YlOrBr", origin="upper", aspect="auto"
)
# Display values above each pixel
for i in range(len(statistics.index)):
for j in range(len(statistics.columns)):
plt.text(
j, i, statistics.values[i, j], ha="center", va="center", color="black"
)
plt.colorbar(heatmap, label="Count")
plt.title("Monthly Species Count by Year")
plt.xlabel("Year")
plt.ylabel("Month")
plt.xticks(range(len(statistics.columns)), statistics.columns)
plt.yticks(range(len(statistics.index)), statistics.index)
plt.tight_layout()
plt.savefig("heatmap_plot.png")
plt.show()
plot_heatmap(gdf)
البيانات المتوفّرة من عام 1995 قليلة جدًا، مع وجود فجوات كبيرة مقارنةً بالسنوات الأخرى، كما أنّ عيّنات البيانات المتوفّرة من شهرَي آب (أغسطس) وأيلول (سبتمبر) محدودة أيضًا وتُظهر خصائص موسمية مختلفة مقارنةً بالفترات الأخرى. قد يساهم استبعاد هذه البيانات في تحسين ثبات النموذج وقدرته التنبؤية.
ومع ذلك، من المهم ملاحظة أنّ استبعاد البيانات قد يعزّز قدرة النموذج على التعميم، ولكنّه قد يؤدي أيضًا إلى فقدان معلومات قيّمة ذات صلة بأهداف البحث. لذلك، يجب اتخاذ هذه القرارات بعد دراسة متأنية.
# Filtering data by year and month
filtered_gdf = gdf[
(~gdf['year'].eq(1995)) &
(~gdf['month'].between(8, 9))
]
الآن، يتم تحويل GeoDataFrame الذي تمّت فلترته إلى عنصر Google Earth Engine.
# Convert GeoDataFrame to Earth Engine object
data_raw = geemap.geopandas_to_ee(filtered_gdf)
بعد ذلك، سنحدّد حجم بكسل النقطة في نتائج نموذج التوزيع المكاني على أنّه بدقة 1 كيلومتر.
# Spatial resolution setting (meters)
grain_size = 1000
عندما تتوفّر نقاط ظهور متعدّدة ضمن بكسل نقطي بدقة 1 كيلومتر نفسه، يرجّح أن تتشارك الظروف البيئية نفسها في الموقع الجغرافي نفسه. يمكن أن يؤدي استخدام هذه البيانات مباشرةً في التحليل إلى إدخال تحيّز في النتائج.
بعبارة أخرى، علينا الحدّ من التأثير المحتمل لتحيّز أخذ العينات الجغرافية. ولتحقيق ذلك، سنحتفظ بموقع جغرافي واحد فقط ضمن كل بكسل يبلغ طول ضلعه كيلومترًا واحدًا، وسنزيل جميع المواقع الجغرافية الأخرى، ما يسمح للنموذج بعكس الظروف البيئية بشكل أكثر موضوعية.
def remove_duplicates(data, grain_size):
# Select one occurrence record per pixel at the chosen spatial resolution
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
rand_point_vals = random_raster.sampleRegions(
collection=ee.FeatureCollection(data), geometries=True
)
return rand_point_vals.distinct("random")
data = remove_duplicates(data_raw, grain_size)
# Before selection and after selection
print("Original data size:", data_raw.size().getInfo())
print("Final data size:", data.size().getInfo())
يظهر أدناه التمثيل المرئي الذي يقارن بين تحيّز أخذ العيّنات الجغرافية قبل المعالجة المسبقة (باللون الأزرق) وبعدها (باللون الأحمر). لتسهيل المقارنة، تم توسيط الخريطة على المنطقة التي تضم عددًا كبيرًا من إحداثيات ظهور طائر "بيتا الجنية" في حديقة "هالاسان الوطنية".
# Visualization of geographic sampling bias before (blue) and after (red) preprocessing
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
# Add the random raster layer
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
Map.addLayer(
random_raster,
{"min": 0, "max": 1, "palette": ["black", "white"], "opacity": 0.5},
"Random Raster",
)
# Add the original data layer in blue
Map.addLayer(data_raw, {"color": "blue"}, "Original data")
# Add the final data layer in red
Map.addLayer(data, {"color": "red"}, "Final data")
# Set the center of the map to the coordinates
Map.setCenter(126.712, 33.516, 14)
Map
تعريف المنطقة محط الاهتمام
يشير تحديد "منطقة الاهتمام" (AOI أدناه) إلى المصطلح الذي يستخدمه الباحثون للإشارة إلى المنطقة الجغرافية التي يريدون تحليلها. وهي تشبه في معناها مصطلح "منطقة الدراسة".
في هذا السياق، حصلنا على المربع المحيط بهندسة طبقة نقطة الوقوع وأنشأنا مخزنًا مؤقتًا يبلغ طوله 50 كيلومترًا حوله (مع حد أقصى للتفاوت يبلغ 1,000 متر) لتحديد منطقة الاهتمام.
# Define the AOI
aoi = data.geometry().bounds().buffer(distance=50000, maxError=1000)
# Add the AOI to the map
outline = ee.Image().byte().paint(
featureCollection=aoi, color=1, width=3)
Map.remove_layer("Random Raster")
Map.addLayer(outline, {'palette': 'FF0000'}, "AOI")
Map.centerObject(aoi, 6)
Map
إضافة متغيرات بيئية في "محرك بحث Google Earth"
الآن، لنضِف متغيّرات بيئية إلى التحليل. توفّر منصة GEE مجموعة كبيرة من مجموعات البيانات للمتغيرات البيئية، مثل درجة الحرارة وهطول الأمطار والارتفاع والغطاء الأرضي والتضاريس. تتيح لنا مجموعات البيانات هذه تحليلًا شاملاً لمختلف العوامل التي قد تؤثّر في البيئات المفضّلة لطائر "بيتا الجنية".
يجب أن يعكس اختيار المتغيرات البيئية في GEE في نموذج توزيع الأنواع خصائص تفضيل الموائل للأنواع. لإجراء ذلك، يجب إجراء بحث مسبق ومراجعة الأدبيات حول البيئات المفضّلة لطائر البيتا الجنية. تركّز هذه الدورة التعليمية بشكل أساسي على سير عمل SDM باستخدام GEE، لذا تم حذف بعض التفاصيل المتعمقة.
WorldClim V1 Bioclim: توفّر مجموعة البيانات هذه 19 متغيّرًا مناخيًا حيويًا مستمدًا من بيانات درجة الحرارة وهطول الأمطار الشهرية. وتغطي هذه المجموعة الفترة من 1960 إلى 1991 وتبلغ دقتها 927.67 مترًا.
# WorldClim V1 Bioclim
bio = ee.Image("WORLDCLIM/V1/BIO")
بيانات الارتفاع الرقمي من مهمة Shuttle Radar Topography Mission (SRTM) التابعة لوكالة ناسا بدقة 30 مترًا: تحتوي مجموعة البيانات هذه على بيانات الارتفاع الرقمي من مهمة Shuttle Radar Topography Mission (SRTM). تم جمع البيانات بشكل أساسي في العام 2000، وهي متوفرة بدقة تبلغ 30 مترًا تقريبًا (ثانية قوسية واحدة). تحسب التعليمة البرمجية التالية طبقات الارتفاع والانحدار والاتجاه والظل التضاريسي من بيانات SRTM.
# NASA SRTM Digital Elevation 30m
terrain = ee.Algorithms.Terrain(ee.Image("USGS/SRTMGL1_003"))
تغيير الغطاء الحرجي العالمي (GFCC) للغطاء الشجري العالمي المتعدد السنوات بدقة 30 مترًا: تقدّر مجموعة بيانات "الحقول المستمرة للغطاء النباتي" (VCF) من Landsat نسبة الغطاء النباتي المسقط عموديًا عندما يكون ارتفاع الغطاء النباتي أكبر من 5 أمتار. تتوفّر مجموعة البيانات هذه لأربع فترات زمنية تتمحور حول الأعوام 2000 و2005 و2010 و2015، بدقة تبلغ 30 مترًا. في هذه الحالة، يتم استخدام قيم الوسيط من هذه الفترات الزمنية الأربع.
# Global Forest Cover Change (GFCC) Tree Cover Multi-Year Global 30m
tcc = ee.ImageCollection("NASA/MEASURES/GFCC/TC/v3")
median_tcc = (
tcc.filterDate("2000-01-01", "2015-12-31")
.select(["tree_canopy_cover"], ["TCC"])
.median()
)
يتم دمج bio (المتغيرات المناخية الحيوية) وterrain (التضاريس) وmedian_tcc (غطاء مظلة الأشجار) في صورة واحدة متعددة النطاقات. يتم اختيار النطاق elevation من terrain، ويتم إنشاء watermask للمواقع الجغرافية التي يكون فيها elevation أكبر من 0. يؤدي ذلك إلى إخفاء المناطق الواقعة تحت مستوى سطح البحر (مثل المحيط) وإعداد الباحث لتحليل العوامل البيئية المختلفة لمنطقة الاهتمام بشكل شامل.
# Combine bands into a multi-band image
predictors = bio.addBands(terrain).addBands(median_tcc)
# Create a water mask
watermask = terrain.select('elevation').gt(0)
# Mask out ocean pixels and clip to the area of interest
predictors = predictors.updateMask(watermask).clip(aoi)
عندما يتم تضمين متغيّرات تنبؤية مرتبطة بشكل كبير معًا في نموذج، يمكن أن تنشأ مشاكل التعدد الخطي. تعدّد الخطية هو ظاهرة تحدث عندما تكون هناك علاقات خطية قوية بين المتغيرات المستقلة في نموذج ما، ما يؤدي إلى عدم استقرار تقدير معاملات النموذج (الأوزان). ويمكن أن يؤدي عدم الاستقرار هذا إلى تقليل موثوقية النموذج وصعوبة تقديم توقّعات أو تفسيرات للبيانات الجديدة. لذلك، سنأخذ في الاعتبار مشكلة التعدد الخطي ونواصل عملية اختيار المتغيرات التنبؤية.
أولاً، سننشئ 5,000 نقطة عشوائية، ثم نستخرج قيم المتغيّر التوقعي لصورة النطاقات المتعددة الفردية عند هذه النقاط.
# Generate 5,000 random points
data_cor = predictors.sample(scale=grain_size, numPixels=5000, geometries=True)
# Extract predictor variable values
pvals = predictors.sampleRegions(collection=data_cor, scale=grain_size)
سنحوّل بعد ذلك قيم المتغير التوقعي المستخرَجة لكل نقطة إلى DataFrame، ثم نتحقّق من الصف الأول.
# Converting predictor values from Earth Engine to a DataFrame
pvals_df = geemap.ee_to_df(pvals)
pvals_df.head(1)
# Displaying the columns
columns = pvals_df.columns
print(columns)
حساب معاملات ارتباط "سبيرمان" بين المتغيّرات المتنبّئة المحدّدة وعرضها في خريطة تمثيل لوني
def plot_correlation_heatmap(dataframe, h_size=10, show_labels=False):
# Calculate Spearman correlation coefficients
correlation_matrix = dataframe.corr(method="spearman")
# Create a heatmap
plt.figure(figsize=(h_size, h_size-2))
plt.imshow(correlation_matrix, cmap='coolwarm', interpolation='nearest')
# Optionally display values on the heatmap
if show_labels:
for i in range(correlation_matrix.shape[0]):
for j in range(correlation_matrix.shape[1]):
plt.text(j, i, f"{correlation_matrix.iloc[i, j]:.2f}",
ha='center', va='center', color='white', fontsize=8)
columns = dataframe.columns.tolist()
plt.xticks(range(len(columns)), columns, rotation=90)
plt.yticks(range(len(columns)), columns)
plt.title("Variables Correlation Matrix")
plt.colorbar(label="Spearman Correlation")
plt.savefig('correlation_heatmap_plot.png')
plt.show()
# Plot the correlation heatmap of variables
plot_correlation_heatmap(pvals_df)
يُعدّ معامل ارتباط سبيرمان مفيدًا لفهم الارتباطات العامة بين المتغيرات المتنبئة، ولكنّه لا يقيّم بشكل مباشر كيفية تفاعل المتغيرات المتعددة، وتحديدًا رصد التعدد الخطي.
معامل تضخّم التباين (VIF أدناه) هو مقياس إحصائي يُستخدَم لتقييم التعدد الخطي وتوجيه عملية اختيار المتغيرات. يشير ذلك المصطلح إلى درجة العلاقة الخطيّة لكل متغيّر مستقل مع المتغيّرات المستقلة الأخرى، ويمكن أن تكون قيم VIF المرتفعة دليلاً على التعدد الخطي.
عادةً، عندما تتجاوز قيم VIF 5 أو 10، يشير ذلك إلى أنّ المتغيّر يرتبط ارتباطًا قويًا بمتغيّرات أخرى، ما قد يؤدي إلى التأثير في ثبات النموذج وقابليته للتفسير. في هذا البرنامج التعليمي، تم استخدام معيار لقيم VIF أقل من 10 لاختيار المتغيرات. تم اختيار المتغيّرات الستة التالية استنادًا إلى عامل تضخّم التباين.
# Filter variables based on Variance Inflation Factor (VIF)
def filter_variables_by_vif(dataframe, threshold=10):
original_columns = dataframe.columns.tolist()
remaining_columns = original_columns[:]
while True:
vif_data = dataframe[remaining_columns]
vif_values = [
variance_inflation_factor(vif_data.values, i)
for i in range(vif_data.shape[1])
]
max_vif_index = vif_values.index(max(vif_values))
max_vif = max(vif_values)
if max_vif < threshold:
break
print(f"Removing '{remaining_columns[max_vif_index]}' with VIF {max_vif:.2f}")
del remaining_columns[max_vif_index]
filtered_data = dataframe[remaining_columns]
bands = filtered_data.columns.tolist()
print("Bands:", bands)
return filtered_data, bands
filtered_pvals_df, bands = filter_variables_by_vif(pvals_df)
# Variable Selection Based on VIF
predictors = predictors.select(bands)
# Plot the correlation heatmap of variables
plot_correlation_heatmap(filtered_pvals_df, h_size=6, show_labels=True)
بعد ذلك، لنعرض على الخريطة المتغيّرات التنبؤية الستة المحدّدة.
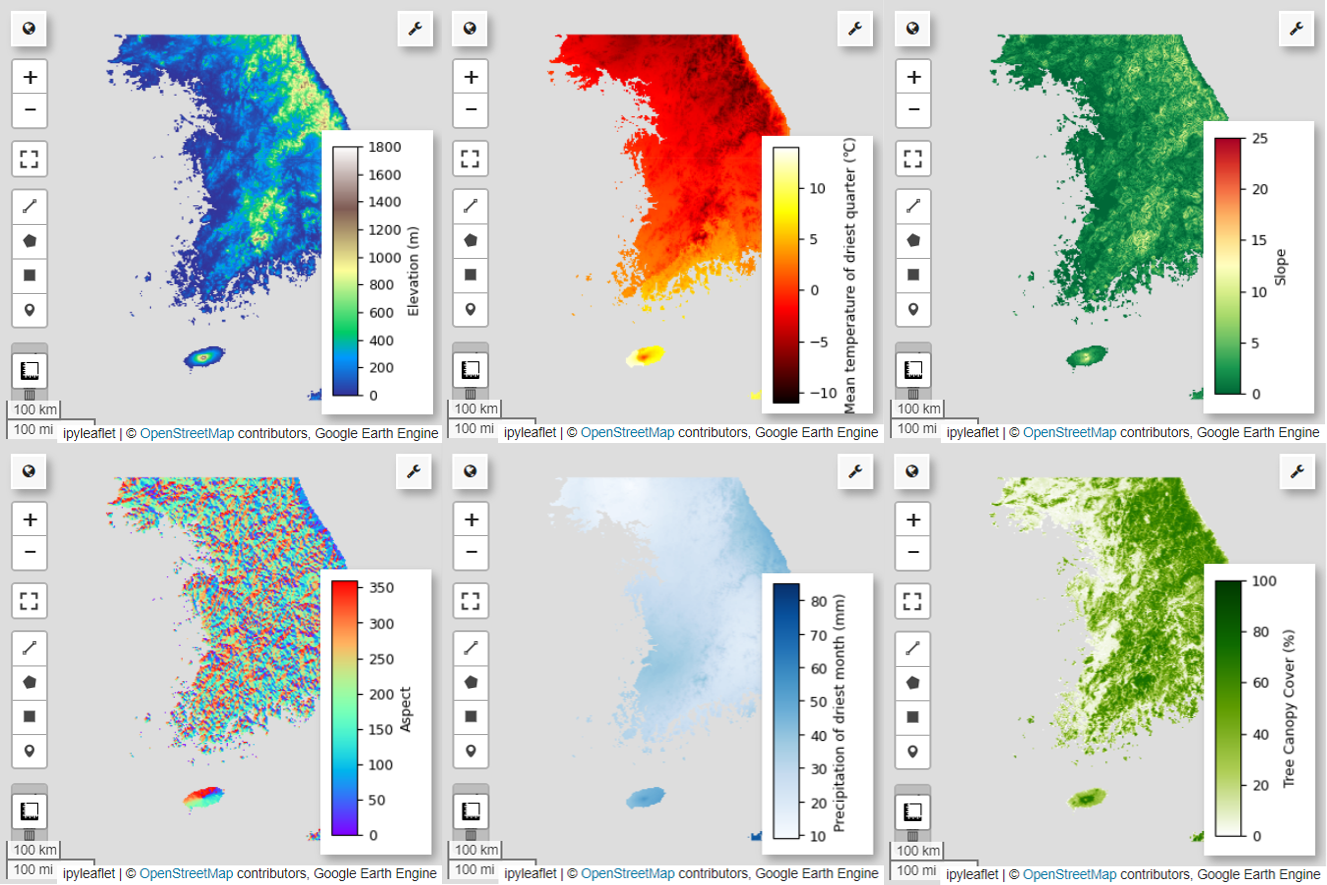
يمكنك استكشاف لوحات الألوان المتاحة لتمثيل الخريطة بصريًا باستخدام الرمز التالي. على سبيل المثال، تبدو لوحة terrain على النحو التالي.
cm.plot_colormaps(width=8.0, height=0.2)
cm.plot_colormap('terrain', width=8.0, height=0.2, orientation='horizontal')
# Elevation layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['elevation'], 'min': 0, 'max': 1800, 'palette': cm.palettes.terrain}
Map.addLayer(predictors, vis_params, 'elevation')
Map.add_colorbar(vis_params, label="Elevation (m)", orientation="vertical", layer_name="elevation")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio09
min_max_val = (
predictors.select("bio09")
.multiply(0.1)
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio09 (Mean temperature of driest quarter) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"min": math.floor(min_max_val["bio09_min"]),
"max": math.ceil(min_max_val["bio09_max"]),
"palette": cm.palettes.hot,
}
Map.addLayer(predictors.select("bio09").multiply(0.1), vis_params, "bio09")
Map.add_colorbar(
vis_params,
label="Mean temperature of driest quarter (℃)",
orientation="vertical",
layer_name="bio09",
)
Map.centerObject(aoi, 6)
Map
# Slope layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['slope'], 'min': 0, 'max': 25, 'palette': cm.palettes.RdYlGn_r}
Map.addLayer(predictors, vis_params, 'slope')
Map.add_colorbar(vis_params, label="Slope", orientation="vertical", layer_name="slope")
Map.centerObject(aoi, 6)
Map
# Aspect layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['aspect'], 'min': 0, 'max': 360, 'palette': cm.palettes.rainbow}
Map.addLayer(predictors, vis_params, 'aspect')
Map.add_colorbar(vis_params, label="Aspect", orientation="vertical", layer_name="aspect")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio14
min_max_val = (
predictors.select("bio14")
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio14 (Precipitation of driest month) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["bio14"],
"min": math.floor(min_max_val["bio14_min"]),
"max": math.ceil(min_max_val["bio14_max"]),
"palette": cm.palettes.Blues,
}
Map.addLayer(predictors, vis_params, "bio14")
Map.add_colorbar(
vis_params,
label="Precipitation of driest month (mm)",
orientation="vertical",
layer_name="bio14",
)
Map.centerObject(aoi, 6)
Map
# TCC layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["TCC"],
"min": 0,
"max": 100,
"palette": ["ffffff", "afce56", "5f9c00", "0e6a00", "003800"],
}
Map.addLayer(predictors, vis_params, "TCC")
Map.add_colorbar(
vis_params, label="Tree Canopy Cover (%)", orientation="vertical", layer_name="TCC"
)
Map.centerObject(aoi, 6)
Map
إنشاء بيانات الغياب الزائف
في عملية نمذجة التوزيع المكاني للأنواع (SDM)، يتم بشكل أساسي اختيار بيانات الإدخال الخاصة بنوع معيّن باستخدام طريقتَين:
طريقة "بيانات الحضور" في الخلفية: تقارن هذه الطريقة المواقع الجغرافية التي تم فيها رصد نوع معيّن من الكائنات الحية (بيانات الحضور) بالمواقع الجغرافية الأخرى التي لم يتم فيها رصد هذا النوع (بيانات الخلفية). في هذه الحالة، لا تشير بيانات الخلفية بالضرورة إلى المناطق التي لا تتواجد فيها الأنواع، بل يتم إعدادها لتعكس الظروف البيئية العامة لمنطقة الدراسة. ويُستخدَم للتمييز بين البيئات المناسبة التي يمكن أن توجد فيها الأنواع والبيئات الأقل ملاءمة.
طريقة التواجد أو عدم التواجد: تقارن هذه الطريقة بين المواقع الجغرافية التي تم فيها رصد الأنواع (التواجد) والمواقع الجغرافية التي لم يتم فيها رصدها بشكل قاطع (عدم التواجد). في هذه الحالة، تمثّل بيانات الغياب مواقع جغرافية محدّدة يُعرف أنّ هذا النوع من الكائنات الحية غير موجود فيها. ولا يشير إلى الظروف البيئية العامة لمنطقة الدراسة، بل إلى المواقع الجغرافية التي يُقدّر أنّ هذا النوع غير موجود فيها.
في الواقع، غالبًا ما يكون من الصعب جمع بيانات الغياب الحقيقي، لذلك يتم استخدام بيانات الغياب الزائف التي يتم إنشاؤها بشكل مصطنع بشكل متكرر. ومع ذلك، من المهم الإقرار بالقيود والأخطاء المحتملة لهذه الطريقة، لأنّ نقاط الغياب الزائف التي يتم إنشاؤها بشكل مصطنع قد لا تعكس بدقة مناطق الغياب الحقيقية.
يعتمد الاختيار بين هاتين الطريقتين على مدى توفّر البيانات وأهداف البحث ودقة النموذج وموثوقيته، بالإضافة إلى الوقت والموارد. سنستخدم هنا بيانات الوقوع التي تم جمعها من GBIF وبيانات الغياب الزائف التي تم إنشاؤها بشكل مصطنع لنموذج باستخدام طريقة "الوجود والغياب".
سيتم إنشاء بيانات الغياب الزائف من خلال "نهج تحديد السمات البيئية"، وفي ما يلي الخطوات المحددة:
التصنيف البيئي باستخدام التجميع العنقودي k-means: سيتم استخدام خوارزمية التجميع العنقودي k-means، استنادًا إلى مسافة إقليدية، لتقسيم وحدات البكسل داخل منطقة الدراسة إلى مجموعتين. ستُمثّل إحدى المجموعتين المناطق التي تتشابه في خصائصها البيئية مع 100 موقع جغرافي تم اختيارها عشوائيًا، بينما ستمثّل المجموعة الأخرى المناطق التي تختلف في خصائصها.
إنشاء بيانات الغياب الزائف ضمن المجموعات العنقودية غير المتشابهة: ضمن المجموعة العنقودية الثانية التي تم تحديدها في الخطوة الأولى (والتي تتضمّن خصائص بيئية مختلفة عن بيانات الحضور)، سيتم إنشاء نقاط غياب زائف تم إنشاؤها عشوائيًا. ستمثّل نقاط الغياب الزائف هذه المواقع الجغرافية التي لا يُتوقّع وجود الأنواع فيها.
# Randomly select 100 locations for occurrence
pvals = predictors.sampleRegions(
collection=data.randomColumn().sort('random').limit(100),
properties=[],
scale=grain_size
)
# Perform k-means clustering
clusterer = ee.Clusterer.wekaKMeans(
nClusters=2,
distanceFunction="Euclidean"
).train(pvals)
cl_result = predictors.cluster(clusterer)
# Get cluster ID for locations similar to occurrence
cl_id = cl_result.sampleRegions(
collection=data.randomColumn().sort('random').limit(200),
properties=[],
scale=grain_size
)
# Define non-occurrence areas in dissimilar clusters
cl_id = ee.FeatureCollection(cl_id).reduceColumns(ee.Reducer.mode(),['cluster'])
cl_id = ee.Number(cl_id.get('mode')).subtract(1).abs()
cl_mask = cl_result.select(['cluster']).eq(cl_id)
# Presence location mask
presence_mask = data.reduceToImage(properties=['random'],
reducer=ee.Reducer.first()
).reproject('EPSG:4326', None,
grain_size).mask().neq(1).selfMask()
# Masking presence locations in non-occurrence areas and clipping to AOI
area_for_pa = presence_mask.updateMask(cl_mask).clip(aoi)
# Area for Pseudo-absence
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(area_for_pa, {'palette': 'black'}, 'AreaForPA')
Map.centerObject(aoi, 6)
Map
ملاءمة النموذج والتوقّع
سنقسم البيانات الآن إلى بيانات تدريب وبيانات اختبار. سيتم استخدام بيانات التدريب للعثور على المَعلمات المثالية من خلال تدريب النموذج، بينما سيتم استخدام بيانات الاختبار لتقييم النموذج الذي تم تدريبه مسبقًا. من المفاهيم المهمة التي يجب مراعاتها في هذا السياق هو الارتباط الذاتي المكاني.
الارتباط الذاتي المكاني هو عنصر أساسي في نموذج SDM، وهو مرتبط بقانون توبلر. وهي تجسّد المفهوم القائل بأنّ "كل شيء مرتبط بكل شيء آخر، ولكن الأشياء القريبة تكون أكثر ارتباطًا من الأشياء البعيدة". يمثّل الارتباط الذاتي المكاني العلاقة المهمة بين موقع الأنواع والمتغيرات البيئية. ومع ذلك، إذا كان هناك ارتباط مكاني تلقائي بين بيانات التدريب وبيانات الاختبار، يمكن أن تتأثر الاستقلالية بين مجموعتَي البيانات. يؤثّر ذلك بشكل كبير في تقييم قدرة النموذج على التعميم.
إحدى طرق حلّ هذه المشكلة هي تقنية التحقّق المتبادل من صحة الحظر المكاني، والتي تتضمّن تقسيم البيانات إلى مجموعات بيانات للتدريب والاختبار. تتضمّن هذه التقنية تقسيم البيانات إلى عدة كتل، واستخدام كل كتلة بشكل مستقل كمجموعة بيانات تدريب واختبار لتقليل تأثير الارتباط الذاتي المكاني. ويعزّز ذلك الاستقلالية بين مجموعات البيانات، ما يسمح بتقييم أكثر دقة لقدرة النموذج على التعميم.
في ما يلي الإجراءات المحدّدة:
- إنشاء مربّعات مكانية: قسِّم مجموعة البيانات بأكملها إلى مربّعات مكانية متساوية الحجم (مثل 50x50 كم).
- تحديد مجموعات التدريب والاختبار: يتم تحديد كل وحدة مكانية عشوائيًا إما لمجموعة التدريب (70%) أو مجموعة الاختبار (30%). ويمنع ذلك النموذج من الإفراط في التكيّف مع البيانات من مناطق معيّنة ويهدف إلى تحقيق نتائج أكثر تعميمًا.
- التحقّق المتبادل التكراري: يتم تكرار العملية بأكملها n مرة (على سبيل المثال، 10 مرات). في كل تكرار، يتم تقسيم المربّعات عشوائيًا إلى مجموعات تدريب واختبار مرة أخرى، وذلك بهدف تحسين ثبات النموذج وموثوقيته.
- إنشاء بيانات الغياب الزائف: في كل تكرار، يتم إنشاء بيانات الغياب الزائف بشكل عشوائي لتقييم أداء النموذج.
Scale = 50000
grid = watermask.reduceRegions(
collection=aoi.coveringGrid(scale=Scale, proj='EPSG:4326'),
reducer=ee.Reducer.mean()).filter(ee.Filter.neq('mean', None))
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(grid, {}, "Grid for spatial block cross validation")
Map.addLayer(outline, {'palette': 'FF0000'}, "Study Area")
Map.centerObject(aoi, 6)
Map
يمكننا الآن ملاءمة النموذج. يتضمّن تركيب نموذج فهم الأنماط في البيانات وتعديل مَعلمات النموذج (الأوزان والانحيازات) وفقًا لذلك. تتيح هذه العملية للنموذج تقديم توقّعات أكثر دقة عند عرض بيانات جديدة عليه. لهذا الغرض، حدّدنا دالة باسم SDM() لتناسب النموذج.
سنستخدم خوارزمية الغابة العشوائية.
def sdm(x):
seed = ee.Number(x)
# Random block division for training and validation
rand_blk = ee.FeatureCollection(grid).randomColumn(seed=seed).sort("random")
training_grid = rand_blk.filter(ee.Filter.lt("random", split)) # Grid for training
testing_grid = rand_blk.filter(ee.Filter.gte("random", split)) # Grid for testing
# Presence points
presence_points = ee.FeatureCollection(data)
presence_points = presence_points.map(lambda feature: feature.set("PresAbs", 1))
tr_presence_points = presence_points.filter(
ee.Filter.bounds(training_grid)
) # Presence points for training
te_presence_points = presence_points.filter(
ee.Filter.bounds(testing_grid)
) # Presence points for testing
# Pseudo-absence points for training
tr_pseudo_abs_points = area_for_pa.sample(
region=training_grid,
scale=grain_size,
numPixels=tr_presence_points.size().add(300),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for training
tr_pseudo_abs_points = (
tr_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(tr_presence_points.size()))
)
tr_pseudo_abs_points = tr_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
te_pseudo_abs_points = area_for_pa.sample(
region=testing_grid,
scale=grain_size,
numPixels=te_presence_points.size().add(100),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for testing
te_pseudo_abs_points = (
te_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(te_presence_points.size()))
)
te_pseudo_abs_points = te_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
# Merge training and pseudo-absence points
training_partition = tr_presence_points.merge(tr_pseudo_abs_points)
testing_partition = te_presence_points.merge(te_pseudo_abs_points)
# Extract predictor variable values at training points
train_pvals = predictors.sampleRegions(
collection=training_partition,
properties=["PresAbs"],
scale=grain_size,
geometries=True,
)
# Random Forest classifier
classifier = ee.Classifier.smileRandomForest(
numberOfTrees=500,
variablesPerSplit=None,
minLeafPopulation=10,
bagFraction=0.5,
maxNodes=None,
seed=seed,
)
# Presence probability: Habitat suitability map
classifier_pr = classifier.setOutputMode("PROBABILITY").train(
train_pvals, "PresAbs", bands
)
classified_img_pr = predictors.select(bands).classify(classifier_pr)
# Binary presence/absence map: Potential distribution map
classifier_bin = classifier.setOutputMode("CLASSIFICATION").train(
train_pvals, "PresAbs", bands
)
classified_img_bin = predictors.select(bands).classify(classifier_bin)
return [
classified_img_pr,
classified_img_bin,
training_partition,
testing_partition,
], classifier_pr
يتم تقسيم المربّعات المكانية إلى% 70 لتدريب النموذج و% 30 لاختبار النموذج، على التوالي. يتم إنشاء بيانات الغياب الزائف بشكل عشوائي ضمن كل مجموعة تدريب واختبار في كل تكرار. نتيجةً لذلك، يؤدي كل تنفيذ إلى مجموعات مختلفة من بيانات الحضور والغياب الزائف لتدريب النموذج واختباره.
split = 0.7
numiter = 10
# Random Seed
runif = lambda length: [random.randint(1, 1000) for _ in range(length)]
items = runif(numiter)
# Fixed seed
# items = [287, 288, 553, 226, 151, 255, 902, 267, 419, 538]
results_list = [] # Initialize SDM results list
importances_list = [] # Initialize variable importance list
for item in items:
result, trained = sdm(item)
# Accumulate SDM results into the list
results_list.extend(result)
# Accumulate variable importance into the list
importance = ee.Dictionary(trained.explain()).get('importance')
importances_list.extend(importance.getInfo().items())
# Flatten the SDM results list
results = ee.List(results_list).flatten()
يمكننا الآن عرض خريطة مدى ملاءمة الموطن وخريطة التوزيع المحتمل لطائر البيتا الجنية. في هذه الحالة، يتم إنشاء خريطة ملاءمة الموائل باستخدام الدالة mean() لحساب المتوسط لكل موقع بكسل في جميع الصور، ويتم إنشاء خريطة التوزيع المحتمل باستخدام الدالة mode() لتحديد القيمة الأكثر تكرارًا في كل موقع بكسل في جميع الصور.
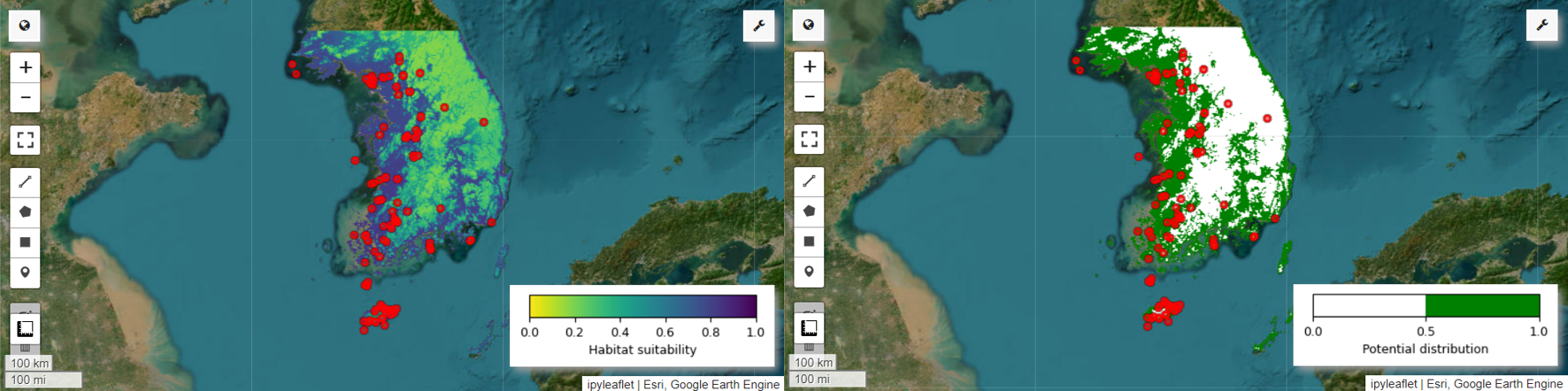
# Habitat suitability map
images = ee.List.sequence(
0, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x))
model_average = ee.ImageCollection.fromImages(images).mean()
Map = geemap.Map(layout={'height':'400px', 'width':'800px'}, basemap='Esri.WorldImagery')
vis_params = {
'min': 0,
'max': 1,
'palette': cm.palettes.viridis_r}
Map.addLayer(model_average, vis_params, 'Habitat suitability')
Map.add_colorbar(vis_params, label="Habitat suitability",
orientation="horizontal",
layer_name="Habitat suitability")
Map.addLayer(data, {'color':'red'}, 'Presence')
Map.centerObject(aoi, 6)
Map
# Potential distribution map
images2 = ee.List.sequence(1, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x)
)
distribution_map = ee.ImageCollection.fromImages(images2).mode()
Map = geemap.Map(
layout={"height": "400px", "width": "800px"}, basemap="Esri.WorldImagery"
)
vis_params = {"min": 0, "max": 1, "palette": ["white", "green"]}
Map.addLayer(distribution_map, vis_params, "Potential distribution")
Map.addLayer(data, {"color": "red"}, "Presence")
Map.add_colorbar(
vis_params,
label="Potential distribution",
discrete=True,
orientation="horizontal",
layer_name="Potential distribution",
)
Map.centerObject(data.geometry(), 6)
Map
أهمية المتغيرات وتقييم الدقة
"الغابة العشوائية" (ee.Classifier.smileRandomForest) هي إحدى طرق التعلّم المجمّع، وتعمل من خلال إنشاء عدة أشجار قرارات لإجراء توقّعات. تتعلّم كل شجرة قرار بشكل مستقل من مجموعات فرعية مختلفة من البيانات، ويتم تجميع نتائجها لتقديم توقّعات أكثر دقة وثباتًا.
أهمية المتغيّر هي مقياس يقيّم تأثير كل متغيّر في التوقّعات ضمن نموذج "الغابة العشوائية". سنستخدم importances_list المحدَّد سابقًا لحساب متوسط أهمية المتغير وطباعته.
def plot_variable_importance(importances_list):
# Extract each variable importance value into a list
variables = [item[0] for item in importances_list]
importances = [item[1] for item in importances_list]
# Calculate the average importance for each variable
average_importances = {}
for variable in set(variables):
indices = [i for i, var in enumerate(variables) if var == variable]
average_importance = np.mean([importances[i] for i in indices])
average_importances[variable] = average_importance
# Sort the importances in descending order of importance
sorted_importances = sorted(average_importances.items(),
key=lambda x: x[1], reverse=False)
variables = [item[0] for item in sorted_importances]
avg_importances = [item[1] for item in sorted_importances]
# Adjust the graph size
plt.figure(figsize=(8, 4))
# Plot the average importance as a horizontal bar chart
plt.barh(variables, avg_importances)
plt.xlabel('Importance')
plt.ylabel('Variables')
plt.title('Average Variable Importance')
# Display values above the bars
for i, v in enumerate(avg_importances):
plt.text(v + 0.02, i, f"{v:.2f}", va='center')
# Adjust the x-axis range
plt.xlim(0, max(avg_importances) + 5) # Adjust to the desired range
plt.tight_layout()
plt.savefig('variable_importance.png')
plt.show()
plot_variable_importance(importances_list)
باستخدام مجموعات بيانات الاختبار، نحسب المساحة تحت منحنى ROC والمساحة تحت منحنى PR لكل عملية تشغيل. بعد ذلك، نحسب متوسط AUC-ROC وAUC-PR على مدار n عملية تكرار.
يمثّل منطقة تحت منحنى ROC المساحة الواقعة تحت منحنى الرسم البياني "الحساسية (الاستدعاء) مقابل 1-النوعية"، ما يوضّح العلاقة بين الحساسية والنوعية عند تغيُّر الحدّ. تستند الدقة إلى جميع حالات عدم الظهور التي تم رصدها. لذلك، يشمل مقياس AUC-ROC جميع أرباع مصفوفة الالتباس.
يمثّل AUC-PR المساحة تحت منحنى الرسم البياني "الدقة مقابل الاكتمال (الحساسية)"، ما يوضّح العلاقة بين الدقة والاكتمال مع اختلاف الحدّ الأدنى. تستند الدقة إلى جميع مرات الظهور المتوقّعة. وبالتالي، لا يشمل مقياس AUC-PR القيم السالبة الصائبة (TN).
def print_pres_abs_sizes(TestingDatasets, numiter):
# Check and print the sizes of presence and pseudo-absence coordinates
def get_pres_abs_size(x):
fc = ee.FeatureCollection(TestingDatasets.get(x))
presence_size = fc.filter(ee.Filter.eq("PresAbs", 1)).size()
pseudo_absence_size = fc.filter(ee.Filter.eq("PresAbs", 0)).size()
return ee.List([presence_size, pseudo_absence_size])
sizes_info = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(get_pres_abs_size)
.getInfo()
)
for i, sizes in enumerate(sizes_info):
presence_size = sizes[0]
pseudo_absence_size = sizes[1]
print(
f"Iteration {i + 1}: Presence Size = {presence_size}, Pseudo-absence Size = {pseudo_absence_size}"
)
# Extracting the Testing Datasets
testing_datasets = ee.List.sequence(
3, ee.Number(numiter).multiply(4).subtract(1), 4
).map(lambda x: results.get(x))
print_pres_abs_sizes(testing_datasets, numiter)
def get_acc(hsm, t_data, grain_size):
pr_prob_vals = hsm.sampleRegions(
collection=t_data, properties=["PresAbs"], scale=grain_size
)
seq = ee.List.sequence(start=0, end=1, count=25) # Divide 0 to 1 into 25 intervals
def calculate_metrics(cutoff):
# Each element of the seq list is passed as cutoff(threshold value)
# Observed present = TP + FN
pres = pr_prob_vals.filterMetadata("PresAbs", "equals", 1)
# TP (True Positive)
tp = ee.Number(
pres.filterMetadata("classification", "greater_than", cutoff).size()
)
# TPR (True Positive Rate) = Recall = Sensitivity = TP / (TP + FN) = TP / Observed present
tpr = tp.divide(pres.size())
# Observed absent = FP + TN
abs = pr_prob_vals.filterMetadata("PresAbs", "equals", 0)
# FN (False Negative)
fn = ee.Number(
pres.filterMetadata("classification", "less_than", cutoff).size()
)
# TNR (True Negative Rate) = Specificity = TN / (FP + TN) = TN / Observed absent
tn = ee.Number(abs.filterMetadata("classification", "less_than", cutoff).size())
tnr = tn.divide(abs.size())
# FP (False Positive)
fp = ee.Number(
abs.filterMetadata("classification", "greater_than", cutoff).size()
)
# FPR (False Positive Rate) = FP / (FP + TN) = FP / Observed absent
fpr = fp.divide(abs.size())
# Precision = TP / (TP + FP) = TP / Predicted present
precision = tp.divide(tp.add(fp))
# SUMSS = SUM of Sensitivity and Specificity
sumss = tpr.add(tnr)
return ee.Feature(
None,
{
"cutoff": cutoff,
"TP": tp,
"TN": tn,
"FP": fp,
"FN": fn,
"TPR": tpr,
"TNR": tnr,
"FPR": fpr,
"Precision": precision,
"SUMSS": sumss,
},
)
return ee.FeatureCollection(seq.map(calculate_metrics))
def calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter):
# Calculate AUC-ROC and AUC-PR
def calculate_auc_metrics(x):
hsm = ee.Image(images.get(x))
t_data = ee.FeatureCollection(testing_datasets.get(x))
acc = get_acc(hsm, t_data, grain_size)
# Calculate AUC-ROC
x = ee.Array(acc.aggregate_array("FPR"))
y = ee.Array(acc.aggregate_array("TPR"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_roc = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
# Calculate AUC-PR
x = ee.Array(acc.aggregate_array("TPR"))
y = ee.Array(acc.aggregate_array("Precision"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_pr = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
return (auc_roc, auc_pr)
auc_metrics = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(calculate_auc_metrics)
.getInfo()
)
# Print AUC-ROC and AUC-PR for each iteration
df = pd.DataFrame(auc_metrics, columns=["AUC-ROC", "AUC-PR"])
df.index = [f"Iteration {i + 1}" for i in range(len(df))]
df.to_csv("auc_metrics.csv", index_label="Iteration")
print(df)
# Calculate mean and standard deviation of AUC-ROC and AUC-PR
mean_auc_roc, std_auc_roc = df["AUC-ROC"].mean(), df["AUC-ROC"].std()
mean_auc_pr, std_auc_pr = df["AUC-PR"].mean(), df["AUC-PR"].std()
print(f"Mean AUC-ROC = {mean_auc_roc:.4f} ± {std_auc_roc:.4f}")
print(f"Mean AUC-PR = {mean_auc_pr:.4f} ± {std_auc_pr:.4f}")
%%time
# Calculate AUC-ROC and AUC-PR
calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter)
قدّمت هذه الدورة التدريبية مثالاً عمليًا على استخدام Google Earth Engine (GEE) لنمذجة توزيع الأنواع (SDM). من أهم النقاط التي يجب تذكُّرها هي تنوّع استخدامات GEE ومرونتها في مجال إدارة البيانات المكانية. تتيح الاستفادة من إمكانات معالجة البيانات الجغرافية المكانية الفعّالة في Earth Engine للباحثين وخبراء الحفاظ على البيئة فرصًا لا حصر لها لفهم التنوّع البيولوجي والحفاظ عليه على كوكبنا. من خلال تطبيق المعرفة والمهارات المكتسبة من هذا البرنامج التعليمي، يمكن للأفراد استكشاف هذا المجال الرائع من الأبحاث البيئية والمساهمة فيه.
 التشغيل في Google Colab
التشغيل في Google Colab
 عرض المصدر على GitHub
عرض المصدر على GitHub