エンベディング フィールドは、分類に使用されるのと同じ方法で、回帰のフィーチャー入力/予測子として使用できます。
このチュートリアルでは、64D エンベディング フィールド レイヤを地上バイオマス(AGB)を予測する重回帰分析の入力として使用する方法について説明します。
NASA の Global Ecosystem Dynamics Investigation(GEDI)ミッションでは、地上トランセクトに沿って 30 m の空間分解能で 60 m 間隔で LIDAR 測定値を収集します。回帰モデルの予測変数として使用される地上バイオマス密度(AGBD)の点推定値を含む GEDI L4A Raster Aboveground Biomass Density データセットを使用します。
リージョンを選択
まず、関心領域を定義します。このチュートリアルでは、インドの西ガーツ山脈のリージョンを選択し、ポリゴンをジオメトリ変数として定義します。または、コードエディタの描画ツールを使用して、対象領域の周りにポリゴンを描画することもできます。このポリゴンは、インポートのジオメトリ変数として保存されます。また、衛星ベースマップを使用して、植生地域を簡単に特定できるようにしています。
var geometry = ee.Geometry.Polygon([[
[74.322, 14.981],
[74.322, 14.765],
[74.648, 14.765],
[74.648, 14.980]
]]);
// Use the satellite basemap
Map.setOptions('SATELLITE');

図: 地上バイオマス予測の対象地域を選択する
期間を選択する
回帰分析を実行する年を選択します。衛星エンベディングは 1 年単位で集計されるため、期間は 1 年全体に設定します。
var startDate = ee.Date.fromYMD(2022, 1, 1);
var endDate = startDate.advance(1, 'year');
衛星エンベディング データセットを準備する
64 バンドの衛星エンベディング画像が回帰の予測子として使用されます。Satellite Embedding データセットを読み込み、選択した年と地域の画像をフィルタします。
var embeddings = ee.ImageCollection('GOOGLE/SATELLITE_EMBEDDING/V1/ANNUAL');
var embeddingsFiltered = embeddings
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
衛星埋め込み画像はタイルに分割され、タイルの UTM ゾーンの投影で提供されます。その結果、対象地域をカバーする複数の衛星エンベディング タイルが取得されます。1 つの画像を取得するには、モザイク処理を行う必要があります。Earth Engine では、入力画像のモザイクにはデフォルトの投影(1 度のスケールの WGS84)が割り当てられます。このモザイクはチュートリアルの後半で集約して再投影するため、元の投影を保持しておくと便利です。setDefaultProjection() 関数を使用して、タイルの 1 つから投影情報を抽出し、モザイクに設定できます。
// Extract the projection of the first band of the first image
var embeddingsProjection = ee.Image(embeddingsFiltered.first()).select(0).projection();
// Set the projection of the mosaic to the extracted projection
var embeddingsImage = embeddingsFiltered.mosaic()
.setDefaultProjection(embeddingsProjection);
GEDI L4A モザイクを準備する
GEDI のバイオマス推定値は回帰モデルのトレーニングに使用されるため、使用する前に無効または信頼性の低い GEDI データをフィルタリングすることが重要です。潜在的に誤った測定値を削除するために、複数のマスクを適用します。
- 品質要件を満たしていないすべての測定結果を削除します(l4_quality_flag = 0 かつ degrade_flag > 0)。
- 相対誤差が大きい測定値(「agbd_se」/「agbd」> 50%)をすべて削除します
- Copernicus GLO-30 デジタル標高モデル(DEM)に基づいて、30% を超える斜面のすべての測定値を削除します。
最後に、対象期間と対象地域の残りのすべての測定値を選択して、モザイクを作成します。
var gedi = ee.ImageCollection('LARSE/GEDI/GEDI04_A_002_MONTHLY');
// Function to select the highest quality GEDI data
var qualityMask = function(image) {
return image.updateMask(image.select('l4_quality_flag').eq(1))
.updateMask(image.select('degrade_flag').eq(0));
};
// Function to mask unreliable GEDI measurements
// with a relative standard error > 50%
// agbd_se / agbd > 0.5
var errorMask = function(image) {
var relative_se = image.select('agbd_se')
.divide(image.select('agbd'));
return image.updateMask(relative_se.lte(0.5));
};
// Function to mask GEDI measurements on slopes > 30%
var slopeMask = function(image) {
// Use Copernicus GLO-30 DEM for calculating slope
var glo30 = ee.ImageCollection('COPERNICUS/DEM/GLO30');
var glo30Filtered = glo30
.filter(ee.Filter.bounds(geometry))
.select('DEM');
// Extract the projection
var demProj = glo30Filtered.first().select(0).projection();
// The dataset consists of individual images
// Create a mosaic and set the projection
var elevation = glo30Filtered.mosaic().rename('dem')
.setDefaultProjection(demProj);
// Compute the slope
var slope = ee.Terrain.slope(elevation);
return image.updateMask(slope.lt(30));
};
var gediFiltered = gedi
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
var gediProjection = ee.Image(gediFiltered.first())
.select('agbd').projection();
var gediProcessed = gediFiltered
.map(qualityMask)
.map(errorMask)
.map(slopeMask);
var gediMosaic = gediProcessed.mosaic()
.select('agbd').setDefaultProjection(gediProjection);
// Visualize the GEDI Mosaic
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(gediMosaic, gediVis, 'GEDI L4A (Filtered)', false);

図: 準備された GEDI バイオマス観測値
入力を再サンプリングして集計する
回帰モデルをトレーニングするためにピクセルをサンプリングする前に、入力を同じピクセル グリッドにリサンプリングして再投影します。GEDI の測定値の水平精度は +/- 9 m です。これは、GEDI AGB の値を Satellite Embedding ピクセルと照合する際に問題となります。この問題を解決するため、すべての入力画像を元のピクセルの平均値を持つ大きなピクセル グリッドに再サンプリングして集計します。これにより、データからノイズを除去し、より優れた機械学習モデルを構築できます。
// Choose the grid size and projection
var gridScale = 100;
var gridProjection = ee.Projection('EPSG:3857')
.atScale(gridScale);
// Create a stacked image with predictor and predicted variables
var stacked = embeddingsImage.addBands(gediMosaic);
// Set the resampling mode
var stacked = stacked.resample('bilinear');
// Aggregate pixels with 'mean' statistics
var stackedResampled = stacked
.reduceResolution({
reducer: ee.Reducer.mean(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// As larger GEDI pixels contain masked original
// pixels, it has a transparency mask.
// We update the mask to remove the transparency
var stackedResampled = stackedResampled
.updateMask(stackedResampled.mask().gt(0));
ピクセルの再投影と集計はコストのかかるオペレーションです。結果として得られたスタック画像をアセットとしてエクスポートし、後続のステップで事前計算された画像を使用することをおすすめします。これにより、大きなリージョンを操作する際に発生する computation timed out エラーや user memory exceeded エラーを回避できます。
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var mosaicExportImage = 'gedi_mosaic';
var mosaicExportImagePath = exportFolder + mosaicExportImage;
Export.image.toAsset({
image: stackedResampled.clip(geometry),
description: 'GEDI_Mosaic_Export',
assetId: mosaicExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
エクスポート タスクを開始し、完了するまで待ちます。完了したら、アセットをインポートしてモデルの構築を続行します。
// Use the exported asset
var stackedResampled = ee.Image(mosaicExportImagePath);
トレーニングの特徴量を抽出する
トレーニング特徴を抽出するための入力データが準備できました。回帰モデルでは、衛星エンベディング バンドを従属変数(予測子)として使用し、GEDI AGBD 値を独立変数(予測値)として使用します。各ピクセルの同時発生値を抽出し、トレーニング データセットを準備します。GEDI 画像はほとんどがマスクされており、値が含まれているのは一部のピクセルのみです。sample() を使用すると、ほとんど空の値が返されます。この問題を解決するために、GEDI マスクからクラスバンドを作成し、stratifiedSample() を使用してマスクされていないピクセルからサンプリングします。
var predictors = embeddingsImage.bandNames();
var predicted = gediMosaic.bandNames().get(0);
print('predictors', predictors);
print('predicted', predicted);
var predictorImage = stackedResampled.select(predictors);
var predictedImage = stackedResampled.select([predicted]);
var classMask = predictedImage.mask().toInt().rename('class');
var numSamples = 1000;
// We set classPoints to [0, numSamples]
// This will give us 0 points for class 0 (masked areas)
// and numSample points for class 1 (non-masked areas)
var training = stackedResampled.addBands(classMask)
.stratifiedSample({
numPoints: numSamples,
classBand: 'class',
region: geometry,
scale: gridScale,
classValues: [0, 1],
classPoints: [0, numSamples],
dropNulls: true,
tileScale: 16,
});
print('Number of Features Extracted', training.size());
print('Sample Training Feature', training.first());
回帰モデルをトレーニングする
これで、モデルをトレーニングする準備が整いました。Earth Engine の多くの分類子は、分類タスクと回帰タスクの両方に使用できます。(クラスではなく)数値を予測するため、分類子を REGRESSION モードで実行するように設定し、トレーニング データを使用してトレーニングできます。モデルをトレーニングしたら、モデルの予測を入力値と比較し、二乗平均平方根誤差(rmse)と相関係数 r^2 を計算して、モデルのパフォーマンスを確認できます。
// Use the RandomForest classifier and set the
// output mode to REGRESSION
var model = ee.Classifier.smileRandomForest(50)
.setOutputMode('REGRESSION')
.train({
features: training,
classProperty: predicted,
inputProperties: predictors
});
// Get model's predictions for training samples
var predicted = training.classify({
classifier: model,
outputName: 'agbd_predicted'
});
// Calculate RMSE
var calculateRmse = function(input) {
var observed = ee.Array(
input.aggregate_array('agbd'));
var predicted = ee.Array(
input.aggregate_array('agbd_predicted'));
var rmse = observed.subtract(predicted).pow(2)
.reduce('mean', [0]).sqrt().get([0]);
return rmse;
};
var rmse = calculateRmse(predicted);
print('RMSE', rmse);
// Create a plot of observed vs. predicted values
var chart = ui.Chart.feature.byFeature({
features: predicted.select(['agbd', 'agbd_predicted']),
xProperty: 'agbd',
yProperties: ['agbd_predicted'],
}).setChartType('ScatterChart')
.setOptions({
title: 'Aboveground Biomass Density (Mg/Ha)',
dataOpacity: 0.8,
hAxis: {'title': 'Observed'},
vAxis: {'title': 'Predicted'},
legend: {position: 'right'},
series: {
0: {
visibleInLegend: false,
color: '#525252',
pointSize: 3,
pointShape: 'triangle',
},
},
trendlines: {
0: {
type: 'linear',
color: 'black',
lineWidth: 1,
pointSize: 0,
labelInLegend: 'Linear Fit',
visibleInLegend: true,
showR2: true
}
},
chartArea: {left: 100, bottom: 100, width: '50%'},
});
print(chart);
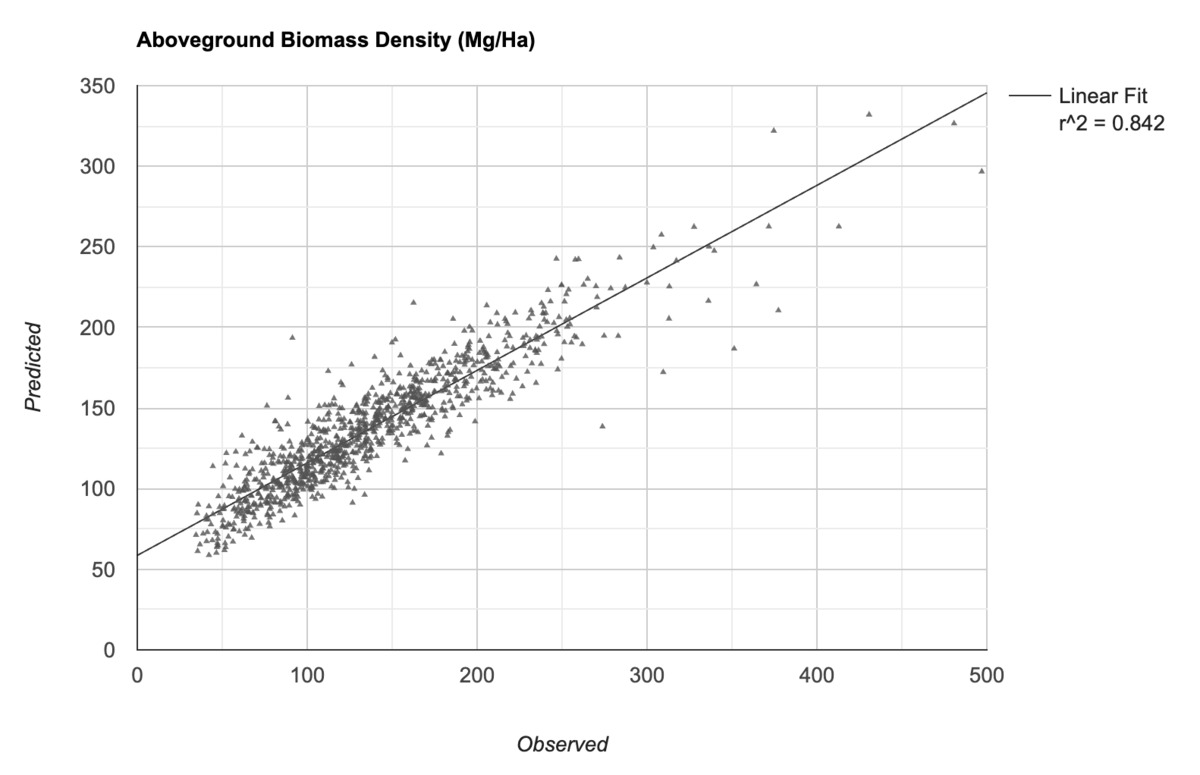
図: 実測値とモデルの予測値の AGBD
不明な値の予測を生成する
モデルに満足したら、トレーニング済みのモデルを使用して、予測子バンドを含む画像から未知の場所の予測を生成できます。
// We set the band name of the output image as 'agbd'
var predictedImage = stackedResampled.classify({
classifier: model,
outputName: 'agbd'
});
各ピクセルの予測 AGBD 値を含む画像がエクスポートの準備完了しました。これは、次のパートで結果を可視化するために使用します。
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var predictedExportImage = 'predicted_agbd';
var predictedExportImagePath = exportFolder + predictedExportImage;
Export.image.toAsset({
image: predictedImage.clip(geometry),
description: 'Predicted_Image_Export',
assetId: predictedExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
エクスポート タスクを開始し、完了するまで待ちます。完了したら、アセットをインポートして結果を可視化します。
var predictedImage = ee.Image(predictedExportImagePath);
// Visualize the image
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(predictedImage, gediVis, 'Predicted AGBD');

図: 予測された AGBD。緑色が濃いほど、予測されるバイオマス密度が高いことを示します。
バイオマスの合計を推定する
これで、画像の各ピクセルの AGBD の予測値が得られました。この値を使用して、地域の地上バイオマス(AGB)の総量を推定できます。ただし、まず植生のない領域に属するすべてのピクセルを削除する必要があります。ESA WorldCover の土地被覆データセットを使用して、植生のあるピクセルを選択できます。
// GEDI data is processed only for certain landcovers
// from Plant Functional Types (PFT) classification
// https://doi.org/10.1029/2022EA002516
// Here we use ESA WorldCover v200 product to
// select landcovers representing vegetated areas
var worldcover = ee.ImageCollection('ESA/WorldCover/v200').first();
// Aggregate pixels to the same grid as other dataset
// with 'mode' value.
// i.e. The landcover with highest occurrence within the grid
var worldcoverResampled = worldcover
.reduceResolution({
reducer: ee.Reducer.mode(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// Select grids for the following classes
// | Class Name | Value |
// | Forests | 10 |
// | Shrubland | 20 |
// | Grassland | 30 |
// | Cropland | 40 |
// | Mangroves | 95 |
var landCoverMask = worldcoverResampled.eq(10)
.or(worldcoverResampled.eq(20))
.or(worldcoverResampled.eq(30))
.or(worldcoverResampled.eq(40))
.or(worldcoverResampled.eq(95));
var predictedImageMasked = predictedImage
.updateMask(landCoverMask);
Map.addLayer(predictedImageMasked, gediVis, 'Predicted AGBD (Masked)');

図: 植生のない領域がマスクされた予測 AGBD
GEDI AGBD 値の単位は、メガグラム / ヘクタール(Mg/ha)です。AGB の合計を取得するには、各ピクセルにその面積(ヘクタール単位)を掛けて、それらの値を合計します。
var pixelAreaHa = ee.Image.pixelArea().divide(10000);
var predictedAgb = predictedImageMasked.multiply(pixelAreaHa);
var stats = predictedAgb.reduceRegion({
reducer: ee.Reducer.sum(),
geometry: geometry,
scale: gridScale,
maxPixels: 1e10,
tileScale: 16
});
// Result is a dictionary with key for each band
var totalAgb = stats.getNumber('agbd');
print('Total AGB (Mg)', totalAgb);